中国海洋湖沼学会主办。
文章信息
- 刘显通, 王宁宁, 吴瑞雪, 李超, 曹敏. 2024.
- LIU Xian-Tong, WANG Ning-Ning, WU Rui-Xue, LI Chao, CAO Min. 2024.
- 许氏平鲉(Sebastes schlegelii)TRAF基因的鉴定及其响应杀鱼爱德华氏菌侵染的表达模式研究
- IDENTIFICATION AND ANALYSIS OF TUMOR NECROSIS FACTOR RECEPTOR-ASSOCIATED FACTORS (TRAF) AND THEIR EXPRESSION PATTERN IN SEBASTES SCHLEGELII AFTER EDWARDSIELLA PISCICIDA INFECTION
- 海洋与湖沼, 55(1): 232-242
- Oceanologia et Limnologia Sinica, 55(1): 232-242.
- http://dx.doi.org/10.11693/hyhz20230700145
文章历史
-
收稿日期:2023-07-11
收修改稿日期:2023-10-13
肿瘤坏死因子超家族(tumor necrosis factor superfamily, TNFSF)和肿瘤坏死因子受体超家族(tumor necrosis factor receptor superfamily, TNFRSF)成员参与了多种先天性和适应性免疫过程, 对细胞增殖、发育、死亡、存活、免疫和各种疾病起到调节作用(Locksley et al, 2001; Collette et al, 2003)。通常, TNFSF配体通过结合TNFSF同源结构域(THD)和TNFRSF的富半胱氨酸结构域(CRDs)与TNFRSF通信, 从而介导免疫相关信号通路和其他发育过程(Bodmer et al, 2002)。活化的TNFRSF可以通过肿瘤坏死因子受体相关因子(tumor necrosis factor receptor associated factors, TRAF)在胞内进行信号传导(Ha et al, 2009)。TRAF是一种兼具重要性和特殊性的胞内转导通路分子, 可以通过与下游信号分子结合, 激活NF-κB信号通路, 介导免疫和炎症反应。TRAF作为一种有凝聚性的衔接蛋白, 其作用可分为两个方面: 一方面, 通过TRAF同源域与受体接受外界刺激; 另一方面, 通过N-端指环/锌指结构与其他蛋白质分子或DNA结合, 依靠这两种途径, 产生一个较为复杂的下端传递信号(李影等, 2015)。
大多数TRAFs成员都含有这样两种结构, N-末端指环状结构域和数目不等的锌指, C-末端TRAF结构域是由卷曲螺旋TRAF-N和TRAF-C结构域构成, 后者是保守的, 这两个结构域的主要功能是维持蛋白质的构象, 对维持其稳定性起到了重要作用。它们都具有特殊的蛋白质结构, 可以帮助识别和结合受体, 并参与细胞因子的分泌、信号转导、细胞增殖、凋亡等生理过程(Chen et al, 2013)。在哺乳动物中, TRAF分子的N端, 都含有1个指环结构域, 并且都含有5~7个锌指结构域(Häcker et al, 2011)。需要指出的是, 指环状结构可以介导DNA-蛋白质和蛋白质-蛋白质这两种形式的转换。研究表明, 除了具备介导功能外, 指环状结构还可以参与到TRAFs的蛋白酶体依赖的降解过程(Chapard et al, 2012)。其中, TRAF1结构是7种TRAFs家族中最为独特的, 它不仅没有上述提到过的TRAF结构N-末端的指环结构域, 也没有锌指结构(Zotti et al, 2012)。TRAF2的特点是其结构域中含有2个亚结构域, 分别是TRAF-N和TRAF-C (Song et al, 2011)。TRAF2作为一种多功能分子, 可以激活NF-κB诱导激酶(NF-κB inducing kinase, NIK)和c-Jun氨基端激酶(c-Jun N-terminal kinases, JNK)信号途径(Cabal-Hierro et al, 2014)。其中, NIK是一种可以通过磷酸化和去磷酸化激活NF-κB信号通路的蛋白质激酶; 而JNK则是一种可以通过磷酸化和去磷酸化激活c-Jun信号通路的蛋白质激酶。TRAF3的功能是TRAF家族中最丰富的。前期研究发现, TRAF3不但可以对NF-κB和MAPK这两种信号通路进行负性调控, 还可以对Ⅰ型干扰素进行正性调控, 从而发挥抗病毒的作用(Muro et al, 2014)。TRAF4是家族中唯一可以通过核定位的蛋白, 这是区别于其他TRAF家族成员的最大特征(Yoon et al, 2014)。TRAF5的特征是表达范围比较广, 在肺、脾、胸腺、肾等器官中表达。与其他成员不同的是, TRAF6在与受体结合时表现出特异性, 可以与白介素-1受体相关激酶、核因子κB受体激动剂、CD40这种信号分子直接结合(Liu et al, 2012)。TRAF7的形式比较特殊, 分为长型和短型。长型就是普通的TRAF7, 它可以编码的氨基酸蛋白数多达670个。短型则被定名为TRAFs (Fu et al, 2011)。因此, 对TRAF结构和功能的了解, 有助于明确TRAF与下游信号分子结合, 激活下游信号通路的分子机制。
在硬骨鱼中, 对于TRAF的报道较少, 仅在少数物种的先天免疫中发挥作用。前期研究证明TRAF2, TRAF3和TRAF6可能还参与了先天免疫系统的抗细胞凋亡途径(Manion, 2012)。例如, 在青鱼(Mylopharyngodon piceus)中, TRAF2可上调MAVS介导的EPC细胞中的IFN信号传导和抗病毒活性(Chen et al, 2017)。研究表明, TRAF3可以通过多种信号通路的介导, 参与多种抗病毒信号通路。在花鲈(Lateolabrax japonicas)中, TRAF3与抵抗RGNNV感染的先天免疫反应有关, 并可能通过RLR信号通路介导的IFN反应发挥其抗病毒活性(Zhang et al, 2018)。TRAF3还可以调节黄酸诱导基因Ⅰ (RIG-Ⅰ)和干扰素基因刺激因子(STING)基因的表达, 从而增强机体对病毒的防御能力(Feng et al, 2011; Wang et al, 2018)。凌露露等克隆获得了日本鳗鲡(Anguilla japonica) TRAF3, 发现poly(I: C)和迟缓爱德华氏菌刺激可显著增强AjTRAF3的表达水平且利用免疫荧光和免疫共沉淀等方法证实了AjTRAF3通过其MATH结构域结合位于线粒体上的MAVS, 从而调控由RLR介导的Ⅰ型IFN抗病毒免疫应答(凌露露等, 2023)。同样, TRAF6还在鱼类天然免疫应答中起着重要的调控作用(Phelan et al, 2005)。此外, 虹鳟(Oncorhynchus mykiss)中的TRAF6可以负调控LPS诱导的p38MAPK和JNK的磷酸化(Jang et al, 2019)。相比在哺乳动物中的研究, 目前对鱼类中TRAF的相关研究较少。因此解析鱼类中TRAF的类型、结构和响应病原菌刺激后的表达模式的研究, 有助于了解鱼类中TRAF基因与哺乳动物中的差异, 并且有助于后续鱼类中TRAF介导信号通路的研究, 对于进一步了解鱼类中TRAF的免疫应答机制具有重要意义。
许氏平鲉(Sebastes schlegelii)是我国主要的海水经济养殖鱼类之一, 具有养殖方便、适应水体环境能力强、生长至成体速度快、营养丰富等优点, 具有很高的经济价值(Kim et al, 2001)。因此, 解析许氏平鲉响应病原菌的免疫应答及调控机制将为认识其疾病的发生和预防提供理论依据。然而随着养殖规模的增大, 各种病原体引起的疾病病害日益严重。其中, 杀鱼爱德华氏菌(Edwardsiella piscicida)已被公认为全球淡水和海水养殖鱼类的主要病原体之一。感染杀鱼爱德华氏菌通常会导致一种称为爱德华氏菌病的系统性疾病的发展, 其特征是腹水、疝气、突眼、脑和内脏器官的严重损伤(Xu et al, 2022)。因此, 了解许氏平鲉免疫系统的运作方式具有非常重要的意义。许多研究者也发现了问题的关键所在, 开始聚焦于许氏平鲉的免疫相关基因和免疫应答机制的研究(Cao et al, 2021, 2022; 闫煦, 2022; Gao et al, 2023)。例如, 曹敏等人对许氏平鲉14个TNFSF和24个TNFRSF基因进行了系统鉴定, 并且检测了其在感染杀鱼爱德华氏菌后的表达模式, 证实了它们参与免疫应答反应, 并且通过蛋白质相互作用(protein-protein interaction, PPI)分析表明了TNFSF基因与免疫相关基因相互作用, 其中包括TRAF家族中的多个成员(Cao et al, 2022)。但是, 目前缺少许氏平鲉TRAF家族成员响应杀鱼爱德华氏菌的研究。因此, 本研究从分子水平上揭示了许氏平鲉TRAF家族基因的结构和功能, 揭示了TRAF可能依赖的免疫通路, 为进一步探究TRAF家族基因在许氏平鲉先天免疫过程中的作用奠定了基础。
1 材料与方法 1.1 实验用鱼及饲养条件本实验所用健康的许氏平鲉均购自山东威海裕源祥水产有限公司, 体长约为(12±2) cm。均饲养于长47 cm、宽32 cm、高约30 cm的海水循环系统中。海水的温度约为20 ℃。
1.2 许氏平鲉TRAF基因的全基因组鉴定本项目利用NCBI全基因组信息(物种包含人类、鼠和多种硬骨鱼), 在NCBI全基因组范围内搜索, 下载TRAF基因序列, 构建BLAST全序列比对数据库, 通过对许氏平鲉全基因组序列的分析, 将其与数据库中的数据进行比较, 最终确定了TRAF基因的候选基因。随后用hmmer搜索程序, 对许氏平鲉的全基因进行了初步筛选, 并对其中包含TRAF重复区域和TRAF功能域的基因进行了确定, 并将部分不包含该功能域的基因进行了初步筛选。在使用hmmer搜索程序后, 我们发现了一些具许氏平鲉的TRAF基因。通过对hmmer和blast的搜索结果的整合, 得到了关于许氏平鲉TRAF家族成员的基因信息。
1.3 许氏平鲉TRAF基因特征分析为了对许氏平鲉TRAF基因进行特性研究, 本项目首先确定每个TRAF基因的序列长度及氨基酸个数, 并在此基础上, 利用许氏平鲉的全基因组注释资料对其进行分类, 并抽取外显子和内含子的数目。利用TBtools将许氏平鲉TRAF基因的染色体定位可视化(Chen et al, 2020)。通过ProtParam在线基因分析工具和ProtComp 9.0在线亚细胞定位工具(http://www.softberry.com)对TRAF基因的分子量(MW)和等电点(pI)进行了计算并对这些TRAF基因的亚细胞定位信息作出了预测。与此同时, 利用SMART结构域分析数据库(http://smart.embl.de/)中获得已知结构的异常同源物和正常同源物, PFAM结构域, 信号肽以及部分内部重复序列等信息, 通过GSDS软件对候选TRAF基因进行功能验证, 构建候选TRAF基因的功能图谱, 并利用TMHMM对计算结果进行了进一步的验证。此外, 基于这些TRAF基因的氨基酸序列, 使用在线程序Protein Homology/Analogy Recognition Engine, V2.0生成了它们的蛋白质二维结构(3D视图)。
1.4 许氏平鲉TRAF基因共线性分析将许氏平鲉与斑马鱼(Danio rerio)、斑点叉尾
为了建立TRAF基因在不同种类间的亲缘关系。首先, 利用MUSCLE算法, 将获得的TRAF序列进行多个序列的比较, 进而构建出一个数据丰富的大样本(Edgar, 2004)。采用ProtTest (Abascal et al, 2005)方法, 通过对TRAF基因进行了进化分析, 筛选出最合适TRAF基因的模型。使用BEAST v.2.2软件重建了系统发育树, 选用G+I+T最优化模型, 不变位点比例选定为0.32, Gamma shape参数选定为0.56, 四个线程在同一时间内运行10 000 000代。最后选用进化树可视化软件iTOL (http://itol.embl.de/)对系统进化树进行调整。
1.6 TRAF家族基因的蛋白质相互作用网络预测及表达模式分析为探索许氏平鲉中TRAF基因的PPI网络, 参考STRING蛋白互作作用分析数据库11.0, 并将识别出的许氏平鲉TRAF家族基因的氨基酸序列引入到其中, 将最小的相互作用参数选定在0.40。
1.7 杀鱼爱德华氏菌攻毒及实时荧光定量实验将生长状况良好的许氏平鲉平均分成四份, 用杀鱼爱德华氏菌(终浓度为1×107 cfu/mL)浸泡感染两小时后, 将实验组和对照组鱼分别转移到正常海水中继续养殖观察。采集对照组和实验组感染后6 h、24 h和96 h许氏平鲉的肠道组织(每三个实验样本为一个生物学重复, 在每个时间点分别采集3个重复), 放入液氮中速冻, 用Trizol法提取RNA, 然后按照PrimeScriptTMRT试剂盒(TaKaRa)说明书合成cDNA进行qRT-PCR实验。
2 结果与讨论 2.1 许氏平鲉TRAF基因的鉴定与表征整合Pfam数据库中TRAF保守结构域和BLAST结果, 在许氏平鲉中共鉴定出9个TRAF基因, 分别命名为TRAF5、TRAF4.1、TRAF4.2、TRAF2.1、TRAF2.2、TRAF3.1、TRAF2.3、TRAF6、TRAF3.2。这些TRAF基因的mRNA长度范围为231 bp (TRAF4.1)到1 794 bp (TRAF2.1)不等。与此同时, TRAF蛋白的长度范围为76 bp (TRAF4.1)至597 bp (TRAF2.1)之间。其中, TRAF2.1的分子量最大(67.04 kDa), 其次为TRAF3.1 (66.93 kDa)和TRAF3.2 (66.23 kDa), 而TRAF4.1 (8.73 kDa)的相对分子质量最小。结果显示, 所有基因的pI值范围均在5.96~8.39之间。在亚细胞定位的统计分析中, 有7个基因定位于细胞核内。除此之外, 我们发现TRAF4.1定位于细胞质, TRAF3.2定位于线粒体(表 1)。
| 基因名字 | 基因长度/bp | 染色体位置 | 氨基酸长度/bp | 分子量/kDa | 等电点 | 亚细胞定位 |
| TRAF5 | 1 752 | 25 528 084~25 536 122 | 583 | 65.26 | 5.96 | Nuclear |
| TRAF4.1 | 231 | 27 326 510~27 328 357 | 76 | 8.73 | 6.18 | Cytoplasmic |
| TRAF4.2 | 1 644 | 27284 634~27 297 111 | 547 | 62.28 | 8.26 | Nuclear |
| TRAF2.1 | 1 794 | 26 089 584~26 107 487 | 597 | 67.04 | 8.39 | Nuclear |
| TRAF2.2 | 1 551 | 22 701 687~22 711 987 | 516 | 58.19 | 8.03 | Nuclear |
| TRAF3.1 | 1 776 | 10 957~21 465 | 591 | 66.93 | 7.02 | Nuclear |
| TRAF2.3 | 1 716 | 24 669 992~24 683 028 | 571 | 64.31 | 8.23 | Nuclear |
| TRAF6 | 1 728 | 36 347 852~36 353 070 | 575 | 64.61 | 6.01 | Nuclear |
| TRAF3.2 | 1 776 | 32 800 365~32 811 792 | 591 | 66.23 | 7.02 | Mitochondrial |
在此次预测结果中, 除了发现TRAF4.1和TRAF4.2外, 其他基因都含有RING结构域。同时, RING在不同基因中的相对位置也是有区别的。TRAF3.1、TRAF3.2、TRAF5、TRAF6、TRAF4.2、TRAF2.2、TRAF2.3、TRAF2.1各有一个MATH结构域。与典型的TRAF蛋白结构域相比, 在TRAF4.1中没有发现RING结构域和MATH结构域。而在TRAF4.2中没有发现RING结构域(图 1)。
 |
| 图 1 TRAF基因的蛋白质结构域 Fig. 1 Protein domain structure of the TRAF gene |
9个TRAF基因中, 有4个(TRAF2.1、TRAF3.1、TRAF3.2、TRAF2.3)含有5'UTR区域和3'UTR区域, 推测它们是全长基因(图 2)。在TRAF6、TRAF4.1、TRAF4.2中没有发现UTR区域, 而TRAF2.2和TRAF5只发现3′UTR区域, 未发现5′UTR区域。通过对其外显子和内含子进行了分析和比较, 进一步对这些TRAF基因的结构多样性进行了研究。研究结果表明, TRAF3.1和TRAF3.2的内含子/外显子数量最多, 分别有14个外显子和13个内含子(图 2, 其中黄色框表示CDs区域, 蓝色框表示UTR区域, 黑线表示内含子), 其余的TRAF基因都有2个以上外显子。同时, 基因功能结构域显示除TRAF4.1和TRAF4.2外, 其余基因大多均有Zf-TRAF superfamily和TRAF_BIRC3_ bd结构域(图 3)。
 |
| 图 2 许氏平鲉中TRAF基因的基因结构 Fig. 2 Gene structure of the TRAF gene in S. schlegelii 注: 黄色框表示CDs区域, 蓝色框表示UTR区域, 黑线表示内含子 |
 |
| 图 3 许氏平鲉中TRAF基因结构域 Fig. 3 TRAF gene domain in S. schlegelii |
为了更好地认识TRAF基因的特点, 我们采用共线性分析方法对许氏平鲉与其他硬骨鱼类的TRAF基因进行了标胶分析(图 4)。提取了来自许氏平鲉、斑马鱼、尼罗罗非鱼、斑点叉尾




 |
| 图 4 TRAF共线性分析 Fig. 4 Collinearity analysis of TRAF 注: a. TRAF2; b. TRAF3; c. TRAF4; d. TRAF5; e. TRAF6 |
结合其他硬骨鱼类的氨基酸序列, 对许氏平鲉的TRAF基因进行了系统演化研究。结果显示, 这些TRAF分为3大支(图 5), 包括TRAF2、TRAF5、TRAF6三大类群。在TRAF2分支中, TRAF2呈现多拷贝, 单独形成一个大的分支。从进化的角度来看, TRAF4亚家族和TRAF6家族成员关系较近。而TRAF3和TRAF5聚集在一起形成一个单独的分支。系统发育分析可以更好地解析TRAF家族基因的进化关系, 同时也证明了本研究对其鉴定及命名的准确性。
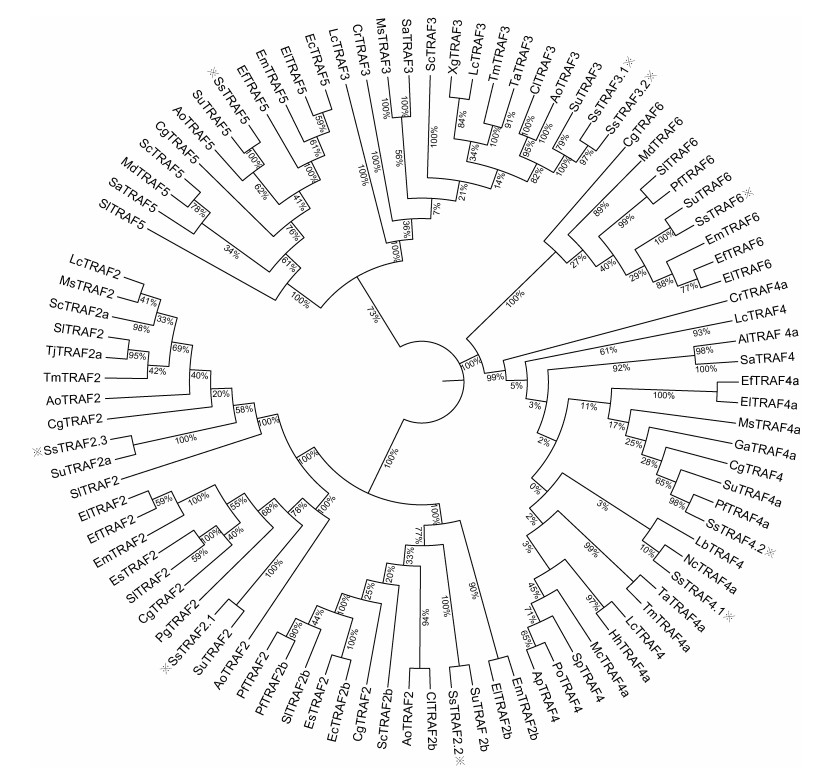 |
| 图 5 TRAF进化树 Fig. 5 The TRAF evolutionary tree |
通过PPI网络分析, 得到TRAF (TRAF2.2、TRAF2.1、TRAF6、TRAF5、TRAF3.1、TRAF3.2、TRAF4.1、TRAF2.3、TRAF4.2)与其互作蛋白的关联状况。如图 6, fadd、tradd、birc2均显示出与TRAF具有高度关联性, 并且在所有TRAF亚家族的整个PPI网络中都发现了map3k14中枢节点。此外, 预测结果也证明了TRAF与TNFRSF存在互作关系(图 6)。
 |
| 图 6 许氏平鲉中TRAF互作蛋白的预测分析 Fig. 6 Predictive analysis of interacting proteins of TRAF in S. schlegelii |
在杀鱼爱德华氏菌侵染许氏平鲉后的不同时间点, 分别取对照组和实验组的肠道组织, 探究TRAF基因在杀鱼爱德华氏菌刺激下的表达模式。结果显示除TRAF4.1外, 其他TRAF基因在感染后均存在不同程度的上调(图 7)。其中, TRAF2.1在感染后2 h上调倍数最大, 是对照组的2.08倍; TRAF2.2在感染后96 h上调倍数最大, 为3.11倍; TRAF2.3仅在感染后24 h时出现上调, 为1.62倍; TRAF3.1在感染后6 h、24 h和96 h分别上调3.17倍、1.32倍和2.39倍; TRAF3.2在感染后2 h上调倍数最高, 为对照组的2.47倍; TRAF4.2在感染后96 h出现峰值, 为对照组的14.24倍; TRAF5在感染后6 h、24 h和96 h分别上调2.66倍、4.57倍和1.96倍; TRAF6在感染后2 h上调倍数最大, 为对照组的2.27倍。
 |
| 图 7 杀鱼爱德华氏菌感染后不同时间点许氏平鲉肠道组织中TRAF基因表达变化 Fig. 7 Expression changes of TRAF in the intestinal tissues of S. schlegelii at different time points following infection by E. piscicida 注: **表示差异极显著(P < 0.01), *表示差异显著(P < 0.05) |
肿瘤坏死因子受体相关因子(TRAF)是TNF受体超家族和白细胞介素-1受体/Toll样受体超家族的主要信号转导子, 调节各种细胞活性和先天免疫反应(Wang et al, 2015)。在本研究中, 我们通过整合Pfam数据库中TRAF保守区域和BLAST结果, 在许氏平鲉中鉴定出9个TRAF基因, 发现部分TRAF家族成员存在多个拷贝, 然后获取了各家族基因的蛋白分子量、等电点以及在染色体上的位置等基本信息。并且通过系统的生物信息学分析了它们的基因结构、功能结构域、共线性排列, 蛋白互作网络以及系统进化关系, 发现它们在结构和功能上是比较保守的。在许氏平鲉中共鉴定到9个TRAF基因, 与黄颡鱼(Pelteobagrus fulvidraco) (8个) (Nie et al, 2022)、大菱鲆(8个) (Zhou et al, 2023)、半滑舌鳎(7个) (Li et al, 2020)等其他硬骨鱼中的数目类似。以前的研究证实大多数硬骨鱼缺乏TRAF1或TRAF5基因, 并且TRAF1基因的缺失比TRAF5基因的缺失更为普遍, 这表明TRAF5基因可能比TRAF1基因在进化上更为保守(Wajant et al, 1998)。在许氏平鲉中, 就是缺失了TRAF1基因, 保留了TRAF5基因, 与前期的研究结果一致。在鉴定到的9个TRAF基因中, 有5个(TRAF2.1、TRAF3.1、TRAF3.2、TRAF5、TRAF2.3)含有5′UTR区域和3′UTR区域, 依据这一点可以推测它们是全长基因。在TRAF6、TRAF4.1、TRAF4.2中没有发现UTR区域, 而TRAF2.2只发现3′UTR区域, 未发现5′UTR区域。其中TRAF3.1和TRAF3.2的外显子数量最多。蛋白质结构域分析结果显示, TRAF4.1既没有RING结构域, 也没有MATH结构域, 而TRAF4.2有MATH结构域却没有RING结构域, 其余的TRAF家族基因均含有MATH结构域和RING结构域。
TRAF2、TRAF3、TRAF4的多拷贝现象在硬骨鱼中较为常见(Li et al, 2020; Nie et al, 2022; Zhou et al, 2023)。另外, 我们发现TRAF5在许氏平鲉、大黄鱼(Larimichthys crocea)和斑点叉尾
已有研究表明鱼类中的TRAF基因也发挥重要的免疫防疫作用。在青鱼中, 经过病原刺激后, 鱼体内的TRAF基因表达水平均显著升高(Wang et al, 2018)。在东北七鳃鳗(Lethenteron morii)中, TRAF6在铜绿假单胞菌(Pseudomonas aeruginosa)感染后的成鱼的鳃、肠和肾组织中显著升高(丁少青等, 2019)。半滑舌鳎经哈维氏菌(Vibrio harveyi)刺激48 h后, 其TRAF基因在各组织中表达量显著升高(Li et al, 2020)。为了探究TRAF基因在许氏平鲉肠道中响应病原菌侵染后的表达模式, 我们构建了杀鱼爱德华氏菌感染的肠道模式, 并通过荧光定量检测了9个TRAF基因在肠道中的表达量。结果显示除TRAF4.1外, 其余TRAF基因均发生了显著上调表达。其中, TRAF4.2在感染后96 h后, 表达量增高了14.24倍。TRAF6在感染后2 h上调倍数最大, 为对照组的2.27倍。但是在嗜水气单胞菌感染的黄颡鱼的肝脏和脾脏中, TRAF4的基因是下调表达的, 而TRAF6也是呈现下调表达的, 与许氏平鲉中的表达模式相反, 但是TRAF2和TRAF3均被诱导表达(Nie et al, 2022)。研究表明TRAF2作为一种多功能分子, 可以激活NF-κB诱导激酶和c-Jun氨基端激酶信号途径(Cabal-Hierro et al, 2014)。因此, 我们推测TRAF2在许氏平鲉中也是通过这两种途径响应病原菌的侵染。但是前期研究发现, TRAF3可以对NF-κB信号通路进行负性调控(Muro et al, 2014)。但是在大菱鲆中的TRAF2的基因的表达是被抑制的(Zhou et al, 2023)。因此, 我们推测, TRAF3的上调表达可以抑制过渡的炎症反应, 对机体进行保护。TRAF5在感染后6 h、24 h和96 h分别上调2.66倍、4.57倍和1.96倍。研究证明尼罗罗非鱼中过表达的TRAF5激活NF-κB (Xia et al, 2019)。因此, 我们推测许氏平鲉中的TRAF5参与抗细菌免疫应答, 在信号转导中起着至关重要的作用。
4 结论以上研究表明许氏平鲉中的TRAF基因在维持免疫屏障的完整性和抗菌免疫应答等方面发挥着重要作用。但是我们对于许氏平鲉TRAF的研究还只是停留在基因层面, 还需拓展到蛋白层面进行进一步的研究。综上所述, 本研究从分子水平上揭示了许氏平鲉TRAF家族基因的结构、功能和系统进化关系, 揭示了TRAF可能依赖的免疫通路, 为进一步探究TRAF家族基因在许氏平鲉先天免疫过程中的作用奠定了基础。
丁少青, 周泽斌, 王雅倩, 等, 2019. 东北七鳃鳗TRAF6基因克隆、表达分析及亚细胞定位研究[J]. 水生生物学报, 43(1): 9-16. |
闫煦, 2022. 许氏平鲉模式识别受体TLR和NLR的鉴定及功能分析[D]. 青岛: 青岛科技大学: 12.
|
李影, 陈镜宇, 张玲玲, 等, 2015. 肿瘤坏死因子受体相关因子参与炎症免疫调节的研究进展[J]. 中国药理学通报, 31(9): 1206-1211. |
凌露露, 梁英, 黄文树, 等, 2023. 日本鳗鲡TRAF3基因的克隆及功能研究[J]. 水产学报, 47(8): 089414. |
ABASCAL F, ZARDOYA R, POSADA D, 2005. ProtTest: selection of best-fit models of protein evolution[J]. Bioinformatics, 21(9): 2104-2105. DOI:10.1093/bioinformatics/bti263 |
BODMER J L, SCHNEIDER P, TSCHOPP J, 2002. The molecular architecture of the TNF superfamily[J]. Trends in Biochemical Sciences, 27(1): 19-26. DOI:10.1016/S0968-0004(01)01995-8 |
CABAL-HIERRO L, RODRÍGUEZ M, ARTIME N, et al, 2014. TRAF-mediated modulation of NF-κB and JNK activation by TNFR2[J]. Cell Signal, 26(12): 2658-2666. DOI:10.1016/j.cellsig.2014.08.011 |
CAO M, WANG N N, YAN X, et al, 2022. Structures, evolutionary relationships and expression profiles of the tumour necrosis factor superfamily and their receptors in black rockfish (Sebastes schlegelii)[J]. Developmental & Comparative Immunology, 132: 104405. |
CAO M, YAN X, SU B F, et al, 2021. Integrated analysis of circRNA-miRNA-mRNA regulatory networks in the intestine of Sebastes schlegelii following Edwardsiella tarda challenge[J]. Frontiers in Immunology, 11: 618687. DOI:10.3389/fimmu.2020.618687 |
CHAPARD C, HOHL D, HUBER M, 2012. The role of the TRAF-interacting protein in proliferation and differentiation[J]. Experimental Dermatology, 21(5): 321-326. DOI:10.1111/j.1600-0625.2012.01477.x |
CHEN C J, CHEN H, ZHANG Y, et al, 2020. TBtools: an integrative toolkit developed for interactive analyses of big biological data[J]. Molecular Plant, 13(8): 1194-1202. DOI:10.1016/j.molp.2020.06.009 |
CHEN B B, COON T A, GLASSER J R, et al, 2013. A combinatorial F box protein directed pathway controls TRAF adaptor stability to regulate inflammation[J]. Nature Immunology, 14(5): 470-479. DOI:10.1038/ni.2565 |
CHEN H, XIAO J, LI J, et al, 2017. TRAF2 of black carp upregulates MAVS-mediated antiviral signaling during innate immune response[J]. Fish & Shellfish Immunology, 71: 1-9. |
COLLETTE Y, GILLES A, PONTAROTTI P, et al, 2003. A co-evolution perspective of the TNFSF and TNFRSF families in the immune system[J]. Trends in Immunology, 24(7): 387-394. DOI:10.1016/S1471-4906(03)00166-2 |
EDGAR R C, 2004. MUSCLE: a multiple sequence alignment method with reduced time and space complexity[J]. BMC Bioinformatics, 5(1): 113. DOI:10.1186/1471-2105-5-113 |
FENG H, LIU H, KONG R Q, et al, 2011. Expression profiles of carp IRF-3/-7 correlate with the up-regulation of RIG-Ⅰ/MAVS/TRAF3/TBK1, four pivotal molecules in RIG-Ⅰ signaling pathway[J]. Fish & Shellfish Immunology, 30(4/5): 1159-1169. |
FU D K, ZHANG Y, XIAO S, et al, 2011. The first homolog of a TRAF7 (TNF receptor-associated factor 7) gene in a mollusk, Crassostrea hongkongensis[J]. Fish & Shellfish Immunology, 31(6): 1208-1210. |
GAO Y L, HAN G S, QIANG L, et al, 2023. Hematological varieties, histological changes, and immune responses in the early stage of infection with Vibrio parahaemolyticus in black rockfish Sebastes schlegelii[J]. Aquaculture International, 31(1): 381-399. DOI:10.1007/s10499-022-00982-9 |
HA H, HAN D, CHOI Y, 2009. TRAF-mediated TNFR-family signaling[J]. Current Protocols in Immunology, 11: 11.9D.1-11.9D.19. |
HÄCKER H, TSENG P H, KARIN M, 2011. Expanding TRAF function: TRAF3 as a tri-faced immune regulator[J]. Nature Reviews Immunology, 11(7): 457-468. DOI:10.1038/nri2998 |
JANG J H, KIM H, CHO J H, 2019. Molecular cloning and functional characterization of TRAF6 and TAK1 in rainbow trout, Oncorhynchus mykiss[J]. Fish & Shellfish Immunology, 84: 927-936. |
KIM K H, HWANG Y J, KWON S R, 2001. Influence of daily water temperature changes on the chemiluminescent response and mortality of cultured rockfish (Sebastes schlegeli)[J]. Aquaculture, 192(2/3/4): 93-99. |
LI K M, LI M, WANG N, et al, 2020. Genome-wide identification, characterization, and expression analysis of the TRAF gene family in Chinese tongue sole (Cynoglossus semilaevis)[J]. Fish & Shellfish Immunology, 96: 13-25. |
LIU H, TAMASHIRO S, BARITAKI S, et al, 2012. TRAF6 activation in multiple myeloma: a potential therapeutic target[J]. Clinical Lymphoma, Myeloma & Leukemia, 12(3): 155-163. |
LOCKSLEY R M, KILLEEN N, LENARDO M J, 2001. The TNF and TNF receptor superfamilies: integrating mammalian biology[J]. Cell, 104(4): 487-501. DOI:10.1016/S0092-8674(01)00237-9 |
MANION B N, 2012. The role of Traf2 in zebrafish [D]. Orono: The University of Maine.
|
MURO I, FANG G, GARDELLA K, et al, 2014. The TRAF3 adaptor protein drives proliferation of anaplastic large cell lymphoma cells by regulating multiple signaling pathways[J]. Cell Cycle, 13(12): 1918-1927. DOI:10.4161/cc.28895 |
NIE Z W, WANG B, ZHANG Z X, et al, 2022. Genome-wide identification of the traf gene family in yellow catfish (Pelteobagrus fulvidraco) and analysis of their expression in response to bacterial challenge[J]. Journal of Fish Biology, 101(3): 573-583. DOI:10.1111/jfb.15126 |
PHELAN P E, MELLON M T, KIM C H, 2005. Functional characterization of full-length TLR3, IRAK-4, and TRAF6 in zebrafish (Danio rerio)[J]. Molecular Immunology, 42(9): 1057-1071. DOI:10.1016/j.molimm.2004.11.005 |
SONG Z F, JIN R, YU S Y, et al, 2011. CD40 is essential in the upregulation of TRAF proteins and NF-κB-dependent proinflammatory gene expression after arterial injury[J]. PLoS One, 6(8): e23239. DOI:10.1371/journal.pone.0023239 |
WAJANT H, MÜHLENBECK F, SCHEURICH P, 1998. Identification of a TRAF (TNF receptor-associated factor) gene in Caenorhabditis elegans[J]. Journal of Molecular Evolution, 47(6): 656-662. DOI:10.1007/PL00006423 |
WANG X, SONG X J, XIE X C, et al, 2018. TRAF3 enhances STING-mediated antiviral signaling during the innate immune activation of black carp[J]. Developmental & Comparative Immunology, 88: 83-93. |
WANG J, WANG R J, WANG S Y, et al, 2015. Genome-wide identification and characterization of TRAF genes in the Yesso scallop (Patinopecten yessoensis) and their distinct expression patterns in response to bacterial challenge[J]. Fish and Shellfish Immunology, 47(1): 545-555. DOI:10.1016/j.fsi.2015.09.050 |
XIA H L, LI Y, WANG Z W, et al, 2019. Expression and functional analysis of tumor necrosis factor receptor (TNFR)-associated factor 5 from Nile tilapia, Oreochromis niloticus[J]. Fish & Shellfish Immunology, 93: 781-788. |
XU Z H, SHAO S, DING Z H, et al, 2022. Therapeutic efficacies of two newly isolated Edwardsiella phages against Edwardsiella piscicida infection[J]. Microbiological Research, 263: 127043. DOI:10.1016/j.micres.2022.127043 |
YOON J H, CHO Y J, PARK H H, 2014. Structure of the TRAF4 TRAF domain with a coiled-coil domain and its implications for the TRAF4 signalling pathway[J]. Acta Crystallographica Section D Biological Crystallography, 70: 2-10. DOI:10.1107/S139900471302333X |
ZHANG W W, JIA P, LIU W, et al, 2018. Functional characterization of tumor necrosis factor receptor-associated factor 3 of sea perch (Lateolabrax japonicas) in innate immune[J]. Fish & Shellfish Immunology, 75: 1-7. |
ZHOU D Y, WANG X G, LI H S, et al, 2023. The TRAF gene family in turbot (Scophthalmus maximus): Identification, characterization, molecular evolution and expression patterns analysis[J]. Fish & Shellfish Immunology, 140: 108950. |
ZOTTI T, VITO P, STILO R, 2012. The seventh ring: exploring TRAF7 functions[J]. Journal of Cellular Physiology, 227(3): 1280-1284. DOI:10.1002/jcp.24011 |
 2024, Vol. 55
2024, Vol. 55


