中国海洋湖沼学会主办。
文章信息
- 王飞扬, 陈素阳, 解家松, 姜建湖, 郭建林, 张海琪. 2024.
- WANG Fei-Yang, CHEN Su-Yang, XIE Jia-Song, JIANG Jian-Hu, GUO Jian-Lin, ZHANG Hai-Qi. 2024.
- 美人鱼发光杆菌(Photobacterium damselae)感染对银鲳(Pampus argenteus)肠道微生物的影响
- EFFECTS OF PHOTOBACTERIUM DAMSELAE INFECTION ON GUT MICROBES IN SILVER POMFRET PAMPUS ARGENTEUS
- 海洋与湖沼, 55(1): 213-222
- Oceanologia et Limnologia Sinica, 55(1): 213-222.
- http://dx.doi.org/10.11693/hyhz20230700136
文章历史
-
收稿日期:2023-07-02
收修改稿日期:2023-10-29
2. 宁波大学海洋学院 浙江宁波 315211
2. School of Marine Sciences, Ningbo University, Ningbo 315211, China
银鲳(Pampus argenteus)属于鲈形目(Pereiformes), 鲳科(Stromateidae), 鲳属(Pampus), 主要分布于中国东海、南海和黄渤海等地区, 是我国沿海重要的经济鱼类之一(徐善良等, 2012)。银鲳肉质嫩滑、刺少骨软, 味道鲜美且具有很高的营养价值和经济价值(王佳等, 2021)。但由于过度捕捞, 其野生种群数量日渐减少(李建生等, 2014)。银鲳是我国水产养殖的重要品种, 目前国内宁波地区已经可进行银鲳的全人工养殖。然而, 在银鲳的规模化养殖中, 时常暴发由美人鱼发光杆菌引起的“出血病”(Tao et al, 2018)。美人鱼发光杆菌(Photobacterium damselae)曾被称为美人鱼弧菌, 为嗜盐的革兰氏阴性菌, 属于海水中常见的一类条件致病菌, 当外界环境恶化或鱼体免疫力下降, 可能导致鱼体感染弧菌(张飞等, 2012; 徐胜威等, 2020)。美人鱼发光杆菌分两个亚种: 美人鱼亚种(P. damselae subsp. damselae) (PDD)和杀鱼亚种(P. damselae subsp. piscicida), 美人鱼亚种广泛分布在水生环境中, 可感染包括爬行动物、鲸类、甲壳类、软体动物和鱼类在内的许多生物, 该亚种对人及其他哺乳动物也具有致病性, 其主要引起水生动物的出血性败血症, 发病较快, 死亡率较高(王洪彬等, 2018)。
微生物与生物之间联系非常密切, 大多数微生物生活在动物的消化道中(Bates et al, 2006)。这些微生物主要由细菌组成, 同时还包括古菌、病毒和真菌等(Sommer et al, 2013)。消化道是鱼类摄食和消化吸收的主要场所, 鱼类肠道微生物群落有助于消化, 并可以影响宿主鱼的营养、生长发育、繁殖和抗病能力(Mahdi et al, 2015)。鱼类肠道微生物群落作为鱼类特殊的功能器官, 主要取决于宿主、周围环境和饮食摄入之间的功能平衡(She et al, 2017)。环境因素, 如温度、盐度、pH等, 对鱼类肠道微生物的影响较为显著(Tarnecki et al, 2017)。此外, 遗传也有一定影响(Wu et al, 2013)。环境的改变或应激反应可能会削弱宿主的免疫功能, 进而改变肠道微生物群落结构, 容易导致外源性致病菌的入侵或内源性机会致病菌大量增殖(Li et al, 2016)。反之, 当疾病发生时, 病原体也会导致肠道微生物群的正常结构改变。
深入了解鱼类肠道微生物与其宿主之间的相互作用, 对于理解宿主的生命活动状态, 尤其是发生疾病的鱼类非常重要。研究病原微生物对宿主肠道微生物的种类和数目的直接或间接影响, 可帮助及时诊断和治疗疾病, 同时也有助于研究病原体的发展过程和机理。虽然已有研究表明消化道微生物变化与人类和动物的许多肠道相关疾病有关(Gao et al, 2015), 但关于PDD感染对银鲳肠道微生物影响的研究却未见报道。因此, 通过研究PDD感染后银鲳肠道微生物群落组成的变化, 不仅有利于研究PDD发病过程和机制, 而且有助于“出血病”的诊断和治疗。
采用扩增子测序方法来研究PDD感染对银鲳肠道微生物群落的动态变化是一种适宜的选择。16S rRNA位于原核细胞核糖体小亚基上, 含有10个保守区(Conserved Regions)和9个高变区(Hypervariable Regions), 其中保守区在不同种类细菌之间特异性较低, 而高变区则在属或种级别上有明显差异。因此, 16S rRNA序列被广泛用于鉴定和区分不同的细菌物种。16S rRNA扩增子测序技术通过选择一个或多个高变区的保守区域设计通用引物用于PCR扩增, 再对高变区域进行测序分析和菌种鉴定, 已成为研究环境样本中微生物群落组成结构的重要手段(Lozupone et al, 2005; Li et al, 2013; Lundberg et al, 2013; Bulgarelli et al, 2015)。
本研究通过分析银鲳对照组和PDD感染组在24 h和72 h所采集的中肠和后肠样本, 采用16S rRNA基因扩增子测序技术并结合Illumina Nova测序平台进行测序和数据分析, 研究PDD感染后不同时间银鲳体内微生物群落组成结构的动态变化。本研究结果有助于深入了解PDD感染对银鲳肠道微生物的影响, 并为银鲳养殖业的健康管理提供科学依据。同时, 本研究对于探究水产动物肠道微生物的多样性、功能和相互关系等问题也有着重要的意义。
1 材料与方法 1.1 银鲳的饲养与感染实验从水产养殖场(宁波象山)采集重量约为(16±4) g的银鲳, 在曝气循环海水中养殖一周。每天早晚各投喂一次颗粒饲料, 每两天换水一次。
在PDD感染试验中, 随机选取12只活力旺盛的银鲳, 其中一半作为对照组, 另一半作为试验组。参考本实验室前期已发表的人工感染方法(Nawaz et al, 2022), 用100 μL 2.6 × 108 CFU/mL PDD细菌悬浮液进行肌肉注射, 感染试验组的6只银鲳, 对照组的6只银鲳注射等体积的PBS。分别于24 h和72 h从这两个养殖池中采集3只感染的银鲳和3只对照组的银鲳。每只采集的银鲳都被保存在冰海水中进行低温麻醉, 然后采集中肠和后肠组织, 包括其内容物。每个肠道样本都用液氮快速冷冻, 并保存在–80 ℃下, 用于进一步分析。
1.2 样本处理采用Illumina MiSeq测序技术对采集到的肠道样本进行细菌群落组成分析。委托北京诺禾致源科技股份有限公司进行DNA的提取、PCR扩增、产物纯化以及16S rRNA基因扩增, 基于Illumina Nova测序平台测序, 构建PCR-free文库, 然后进行双末端(Paired-End)测序。具体操作如下:
用SDS方法提取样本的基因组DNA, 将生物细胞破碎后, 加入SDS使细胞膜裂解, 让蛋白质变性, 从而将蛋白质和多糖等杂质与DNA分开。之后利用琼脂糖凝胶电泳检测DNA的纯度和浓度, 取适量的样本DNA于离心管中, 使用无菌水稀释样本至1 ng/μL。以稀释后的基因组DNA为模板, 根据测序区域的选择, 使用带Barcode的特异引物和New England Biolabs公司的Phusion® High-Fidelity PCR Master Mix with GC Buffer, 结合高效高保真酶进行PCR, 确保扩增效率和准确性。PCR产物使用2%的琼脂糖凝胶进行电泳检测; 对检测合格的PCR产物进行磁珠纯化, 采用酶标定量, 根据PCR产物浓度进行等量混样, 充分混匀后使用2%的琼脂糖凝胶电泳检测PCR产物, 对目的条带使用Qiagen公司提供的胶回收试剂盒回收产物。最后使用TruSeq® DNA PCR-Free Sample Preparation Kit建库试剂盒进行文库构建, 构建好的文库经过Qubit和Q-PCR定量, 文库合格后, 使用NovaSeq6000进行上机测序。
1.3 数据分析根据Barcode序列和PCR扩增引物序列对样本数据进行拆分, 随后截去Barcode和引物序列, 对每个样本的Reads进行拼接, 从而得到原始Tags数据(Raw Tags), 由于存在一定比例的干扰数据(Dirty Data), 为了使信息分析的结果更加准确、可靠, 首先对原始数据进行严格的拼接、过滤, 从而得到高质量的Tags数据(Clean Data), 再经过进一步过滤及嵌合体序列的去除后, 最终获得有效数据(Effective Tags)。基于有效数据, 以97%的一致性(Identity)进行OTUs (Operational Taxonomic Units)聚类, 随后对OTUs的序列进行物种注释, 获得分类学信息并分别在各个分类水平: Kingdom(界)、Phylum(门)、Class(纲)、Order(目)、Family(科)、Genus(属)、Species(种)统计各样本的群落组成, 得到对应的各种物种信息和基于物种的丰度分布情况, 最后再对各样本的数据进行均一化处理, 以便进行后续的Alpha多样性分析和Beta多样性分析。
在Alpha多样性分析中, 使用Qiime软件(Version 1.9.1)计算Observed-otus和Chao1等指数, 使用R软件(Version 2.15.3)绘制稀释曲线, Rank abundance曲线, 物种累积曲线并进行Alpha多样性指数组间差异分析。在多样本比较分析(Beta Diversity)中, 使用R软件(Version 2.15.3)绘制PCA和NMDS图, 探究不同样本或组别间群落结构的差异。PCA分析使用R软件的ade4包和ggplot2软件包, NMDS分析使用R软件的vegan软件包。
2 结果 2.1 Illumina测序及银鲳肠道微生物复杂度分析通过测序, 总共获得了2 365 568条银鲳肠道微生物序列, 平均每样本测得98 565条tags, 经过质控处理得到1 950 750条银鲳肠道微生物序列, 平均每样本81 642条有效数据, 最终用于后续分析的质控有效数据量平均每样本为63 610条(51 649~69 729条质控数据), 质控有效率达65.00%, 平均读取长度为255 bp。碱基识别准确率在99%以上的碱基比例为99.22%~99.74%, 而碱基识别准确率在99.9%以上的碱基比例为97.70%~99.13%, 测序质量高, 适合用于后续的数据分析。以97%的一致性将序列聚类成为OTUs, 共得到4 496个OTUs (表 1)。
| 样本 | 原始PE | 原始Tags | 过滤Tags | 有效Tags | 最终碱基数目(nt) | 平均长度Len(nt) | Q20(%) | Q30(%) | GC(%) | 有效Tags(%) |
| D1.CM.1 | 100 649 | 86 857 | 86 482 | 69 011 | 17 458 728 | 253 | 99.56 | 98.7 | 54.99 | 68.57 |
| D1.CM.2 | 103 867 | 91 026 | 90 524 | 61 662 | 15 603 281 | 253 | 99.5 | 98.57 | 54.04 | 59.37 |
| D1.CM.3 | 111 940 | 84 086 | 83 720 | 69 508 | 17 628 431 | 254 | 99.49 | 98.53 | 54.23 | 62.09 |
| D1.PM.1 | 111 661 | 94 628 | 94 160 | 69 348 | 17 540 327 | 253 | 99.54 | 98.62 | 55.02 | 62.11 |
| D1.PM.2 | 105 039 | 100 764 | 100 318 | 63 151 | 15 982 023 | 253 | 99.45 | 98.47 | 53.36 | 60.12 |
| D1.PM.3 | 108 801 | 100 185 | 99 759 | 69 359 | 17 554 329 | 253 | 99.51 | 98.6 | 53.45 | 63.75 |
| D3.CM.1 | 98 236 | 72 152 | 71 916 | 60 266 | 15 262 292 | 253 | 99.59 | 98.79 | 53.98 | 61.35 |
| D3.CM.2 | 93 815 | 76 955 | 76 708 | 67 502 | 17 191 065 | 255 | 99.61 | 98.8 | 54.83 | 71.95 |
| D3.CM.3 | 86 586 | 52 791 | 52 646 | 51 649 | 13 054 010 | 253 | 99.57 | 98.68 | 55.21 | 59.65 |
| D3.PM.1 | 106 458 | 74 083 | 73 711 | 67 892 | 17 879 701 | 263 | 99.42 | 98.16 | 53.55 | 63.77 |
| D3.PM.2 | 91 018 | 59 477 | 59 301 | 57 090 | 14 481 235 | 254 | 99.54 | 98.58 | 54.18 | 62.72 |
| D3.PM.3 | 100 023 | 75 357 | 75 038 | 63 984 | 16 200 305 | 253 | 99.58 | 98.76 | 54.01 | 63.97 |
| D1.CH.1 | 103 280 | 73 695 | 73 319 | 68 679 | 17 386 748 | 253 | 99.5 | 98.59 | 55.03 | 66.5 |
| D1.CH.2 | 102 905 | 87 262 | 86 884 | 60 503 | 15 299 937 | 253 | 99.42 | 98.44 | 54.6 | 58.8 |
| D1.CH.3 | 107 060 | 95 653 | 95 219 | 68 641 | 17 342 441 | 253 | 99.44 | 98.5 | 52.08 | 64.11 |
| D1.PH.1 | 97 756 | 88 369 | 88 073 | 62 814 | 15 937 446 | 254 | 99.56 | 98.67 | 53.14 | 64.26 |
| D1.PH.2 | 64 836 | 60 291 | 60 072 | 56 758 | 14 389 612 | 254 | 99.59 | 98.73 | 53.61 | 87.54 |
| D1.PH.3 | 102 126 | 83 026 | 82 513 | 61 328 | 16 425 473 | 268 | 99.22 | 97.7 | 53 | 60.05 |
| D3.CH.1 | 103 716 | 93 264 | 92 810 | 63 534 | 16 283 065 | 256 | 99.49 | 98.5 | 51.58 | 61.26 |
| D3.CH.2 | 79 347 | 68 095 | 67 704 | 58 458 | 14 909 951 | 255 | 99.47 | 98.48 | 54.92 | 73.67 |
| D3.CH.3 | 86 688 | 59 919 | 59 709 | 55 447 | 14 058 275 | 254 | 99.59 | 98.75 | 56.04 | 63.96 |
| D3.PH.1 | 96 853 | 87 777 | 87 616 | 69 729 | 17 641 377 | 253 | 99.74 | 99.13 | 53.92 | 71.99 |
| D3.PH.2 | 107 107 | 100 173 | 99 537 | 68 502 | 17 493 826 | 255 | 99.43 | 98.37 | 52.04 | 63.96 |
| D3.PH.3 | 95 801 | 93 517 | 93 011 | 61 813 | 15 642 282 | 253 | 99.47 | 98.51 | 55.1 | 64.52 |
| 注: 原始PE: 原始下机的PE reads; Raw Tags: 拼接得到的Tags序列; Clean Tags: Raw Tags过滤低质量和短长度后的序列; Effective Tags: 最终用于后续分析的Tags序列; Base(nt): 最终Effective Data的碱基数目; AvgLen(nt): Effective Tags的平均长度; Q20(%)和Q30(%): Effective Tags中碱基质量值大于20 (测序错误率小于1%)和30 (测序错误率小于0.1%)的碱基所占的百分比; GC(%): Effective Tags中GC碱基的含量; Effective (%): Effective Tags的数目与Raw PE数目的百分比 | ||||||||||
以抽取的测序数据量与对应的物种数构建稀释曲线(Rarefaction Curve), 可看到曲线逐渐趋向平坦, 说明测序数据量合理, 每个样本都获得了足够的采样深度(图 1a)。构建Rank Abundance曲线, 在水平方向上, 曲线的跨度大, 垂直方向曲线较为平滑, 说明样本的丰富度较高且分布较均匀度(图 1b)。物种累积箱形图(species accumulation boxplot)是描述随着样本量的增加物种多样性增加的分析, 其结果可反映持续抽样下新OUT (新物种)出现的速率。随着样本量的加大, 箱形图位置逐渐平缓, 表明抽样充分, 适合进行数据分析(图 1c)。
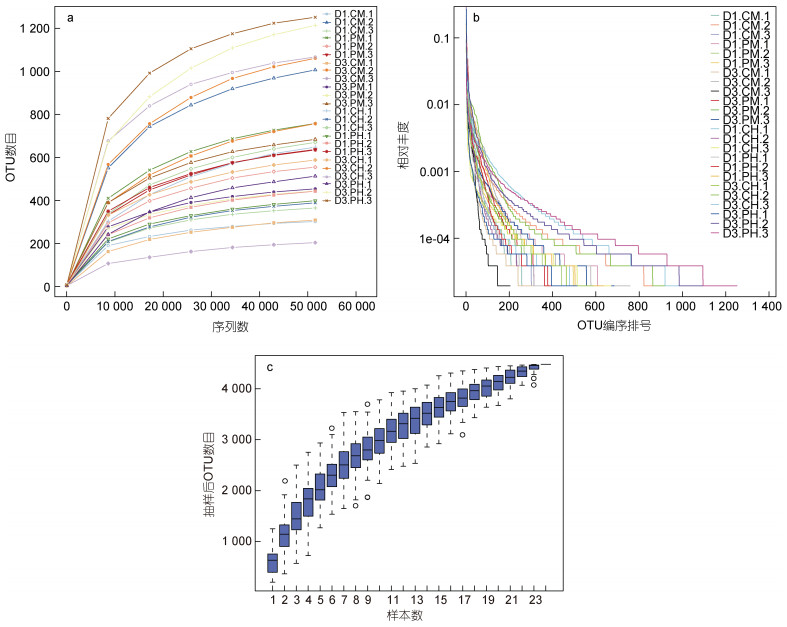 |
| 图 1 感染PDD的银鲳及对照组银鲳在24 h、72 h的中肠和后肠微生物群落的稀疏性分析(a)、Rank Abundance曲线(b)以及物种累积箱形图(c) Fig. 1 The rarefaction curve (a), the rank abundance curves (b) and the species accumulation boxplots (c) of PDD-infected silver pomfret and healthy silver pomfret at midgut and hindgut microbial communities at 24 h and 72 h 注: D1.CM.1~D1.CM.3: 第1d对照组三个中肠样本; D3.CM.1~D3.CM.3: 第3d对照组三个中肠样本; D1.CH.1~D1.CH.3: 第1d对照组三个后肠样本; D1.CH.1~D1.CH.3: 第3d对照组三个后肠样本; D1.PM.1~D1.PM.3: 第1d感染组三个中肠样本; D3.PM.1~D3.PM.3: 第3d感染组三个中肠样本; D1.PH.1~D1.PH.3: 第1d感染组三个后肠样本; D1.PH.1~D1.PH.3: 第3d感染组三个后肠样本 |
为了估计和比较各样本中的微生物多样性, 从OTUs中计算细菌丰富度和多样性指数, 对不同样本在97%一致性阈值下的Alpha Diversity分析指数(Chao1、observed_species)进行统计、制图(图 2)。可以看到感染组和对照组的中肠肠道微生物丰富度在72 h时都有所下降, 而两组的后肠肠道微生物丰富度在72 h时均大幅提高, 尤其是感染组, 提高较多; 对照组24 h及72 h的中肠样本的数据差异略大, 可能是由于银鲳的个体差异造成, 而感染组的差异较小, 可能是由于PDD感染引起肠道微生物菌群的变化导致; 对比G2和G4、G6和G8, 随着PDD感染时间的增加, 样本个别数据差异有所加大, 说明PDD感染可能会引起银鲳肠道微生物紊乱。
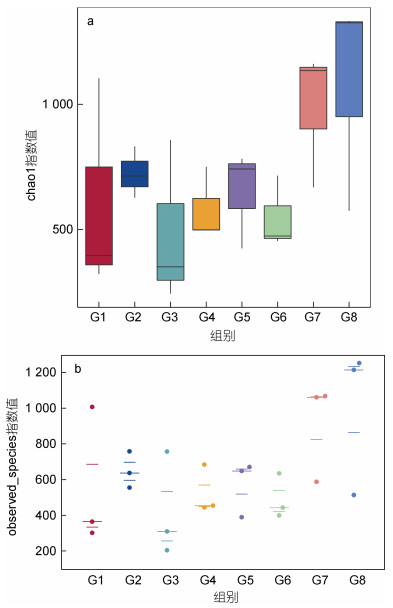 |
| 图 2 Chao1指数组间差异箱形图(a)和observed_species指数组间差异蜜蜂群图(b) Fig. 2 Chao1 Boxplot of differences between groups (a); observed_species index bee colonies of differences between groups (b) 注: G1: D1.CM.1~D1.CM.3; G2: D1.PM.1~D1.PM.3; G3: D3.CM.1~D3.CM.3; G4: D3.PM.1~D3.PM.3; G5: D1.CH.1~D1.CH.3; G6: D1.PH.1~D1.PH.3; G7: D3.CH.1~D3.CH.3; G8; D3.PH.1~D3.PH.3 |
为了探究PDD感染对银鲳肠道微生物结构的影响, 根据前两个主成分(PC1和PC2)构建了银鲳肠道样本微生物群落组成的PCA分析(图 3a)。从PCA图(图 3a)看出, G1和G3样本位置主要在右上方区域附近, G2和G4样本位置主要在左下方区域附近, 显示出了PDD感染后中肠与对照组中肠微生物结构的差异性; 而PDD感染组的后肠样本比对照组的表现出更多的离散性。为了克服线性模型(PCA)的缺点, 更好反映数据的非线性结构, 根据样本中的物种信息, 进行NMDS分析(图 3b)。NMDS图(图 3b)显示, G1和G3样本位置主要聚集在方框内, G2和G4样本在左上方靠近坐标轴的位置; G5和G7样本位置主要分布在横坐标轴上下, 而G6和G8样本位置较为离散, 和PCA分析结果相似。
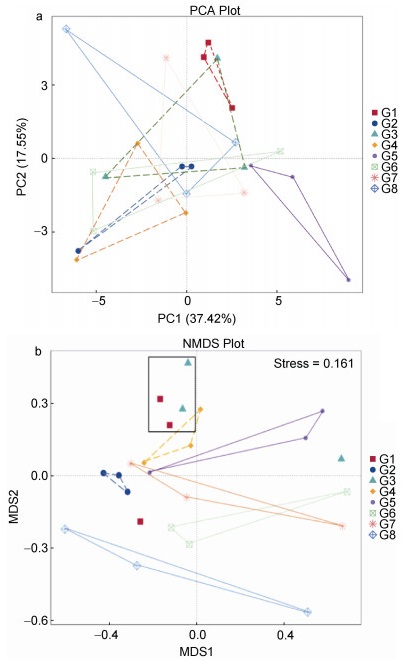 |
| 图 3 银鲳肠道样本微生物群落组成的PCA分析(a)和NMDS分析(b) Fig. 3 The PCA analysis (a) and The NMDS analysis (b) of microbial community composition of silver pomfret intestinal samples 注: a. 横坐标表示第一主成分, 百分比则表示第一主成分对样本差异的贡献值; 纵坐标表示第二主成分, 百分比表示第二主成分对样本差异的贡献值; 图中的每个点表示一个样本, 同一个组的样本使用同一种颜色表示。b. 图中的每个点表示一个样本, 同一个组的样本使用同一种颜色表示, 点与点之间的距离表示差异程度 |
为了进一步揭示PDD感染对银鲳肠道微生物群落组成的影响, 对银鲳肠道在属和门水平上对主要微生物的相对丰度进行了分析(图 4、图 5)。结果表明35.19%的有效序列被注释到属水平, 其中占据优势地位的主要有弧菌属(Vibrio)、发光杆菌属(Photobacterium)、未定义蓝藻门属(unidentified_ Chloroplast)和不动杆菌属(Acinetobacter) (图 4)。属水平微生物丰度图显示, 24 h时感染组的中肠和后肠样本中发光杆菌属比对照组的更少, 大多都是未定义蓝藻门属或弧菌属占主导地位, 而在72 h时发光杆菌属才逐渐增多, 且后肠中发光杆菌属增长得更快; 对照组的肠道内也有发光杆菌属的存在, 但一般不会随时间增多, 绝大部分都在72 h时出现减少(图 4)。而77.18%的有效序列被注释到门水平, 占据主导地位的主要包括变形菌门(Proteobacteria)、蓝藻门(Cyanobacteria)、梭杆菌门(Fusobacteria)、厚壁菌门(Firmicutes)和放线菌门(Actinobacteriota) (图 5)。通过门水平上微生物的平均相对丰度图(图 5b)可以看出, 感染组和对照组中肠样本中的变形菌门在72 h时对比24 h时都有所减少, 而蓝藻门均有所增加; 后肠样本中变形菌门在72 h时均有所增加, 而蓝藻门均有所减少, 结合属水平肠道微生物丰富度图(图 4), 已知发光杆菌属和弧菌属都是属于变形菌门, 于是可以得出以下结论, 对照组中肠中发光杆菌的减少以及感染组中肠中弧菌的减少可能导致了两组中肠样本中变形菌门的减少, 而后肠中感染组发光杆菌在72 h的大量增加及对照组72 h时未定义蓝藻门属的减少可能是后肠中变形菌门增多的原因, 这种变化可能是感染组肠道内PDD的大量增殖所导致; 不同于对照组72 h时中肠样本中厚壁菌门的减少, 感染组中肠中厚壁菌门在72 h增多了, 但感染组后肠样本中厚壁菌门在72 h则减少了, 这也可能是由于PDD感染引起; 再结合门水平上微生物的丰富度图(图 5a)进行分析, 当样本中的蓝藻门较多时, 变形菌门则相对较少, 说明蓝藻门与变形菌门之间可能存在竞争关系, 而PDD感染可能会影响这种竞争关系。
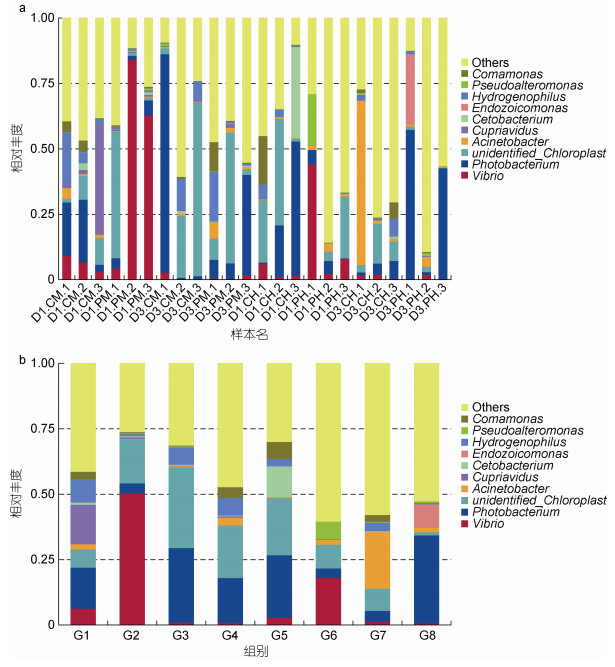 |
| 图 4 感染PDD的银鲳及对照组银鲳在24 h、72 h的中肠和后肠属水平微生物的相对丰度(a)以及平均相对丰度(b) Fig. 4 The relative abundance (a) and The mean relative abundance (b) of microorganisms at the midgut and hindgut of PDD-infected silver pomfret and control group silver pomfret at 24 h and 72 h at the genus level |
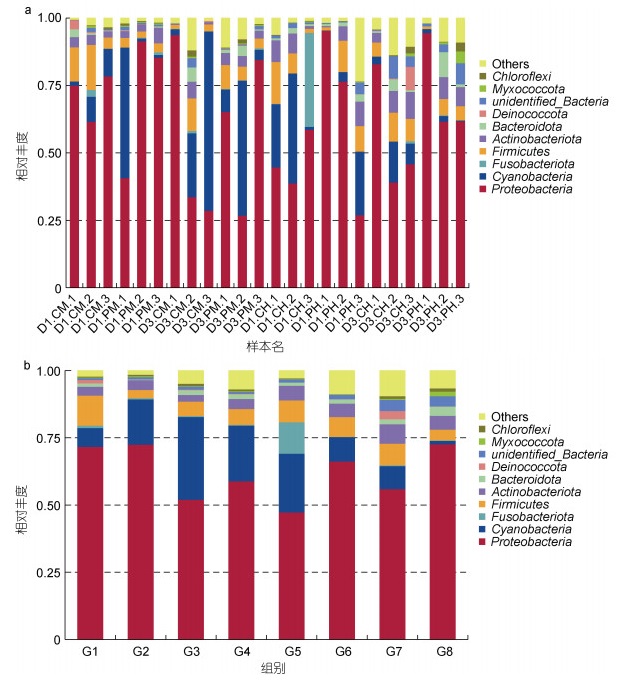 |
| 图 5 感染PDD的银鲳及对照组银鲳在24 h、72 h的中肠和后肠门水平微生物的相对丰度(a)以及平均相对丰度(b) Fig. 5 The relative abundance (a) and The mean relative abundance (b) of microorganisms at the midgut and hindgut of PDD-infected silver pomfret and control group silver pomfret at 24 h and 72 h at the phylum level |
肠道微生物群落在宿主的消化和免疫系统中发挥着重要作用(Ganguly et al, 2012)。在迄今为止正式描述的所有细菌门中, 变形菌门、梭杆菌门、厚壁菌门、拟杆菌门和放线菌门主要出现在鱼类肠道菌群中(Ghanbari et al, 2015), 这与我们的研究结果相符合。在鱼类肠道微生物中, 由于鱼类不同的食性, 因而存在满足不同营养物质消化吸收的标志性微生物, 蛋白酶和脂肪酶有助于肉食性鱼类的消化吸收, 所以在肉食性鱼类的肠道菌群中往往会存在单胞杆菌属、鲸杆菌属(Cetobacterium)等微生物(孟晓林等, 2019; 骆启豪等, 2021)。银鲳为广食性鱼类, 主要摄食桡足类、头足类、虾类、水母类、箭虫、幼鱼和浮游动物等(彭士明等, 2011)。本研究中, 单胞杆菌属、鲸杆菌属在银鲳的肠道内都有发现, 与前人研究结果一致。此外, 本研究还在银鲳肠道菌群中发现了较多的蓝藻门微生物, 其来源可能与银鲳摄食浮游动物有关, 而其较多的丰富度可能与养殖银鲳肠道菌群中产纤维素酶的微生物种类较少有关(王建建等, 2014)。
研究结果显示, 银鲳肠道微生物中的大多数都分类于变形菌门, 这与过去的发现相符(Wu et al, 2010; Roeselers et al, 2011)。变形菌门包含了弧菌科, 在银鲳肠道中, 弧菌科被确定为丰富度最大的科, 这一发现与海洋鱼类群落中肉食性鱼类肠道微生物群的其他研究一致(Sullam et al, 2012)。弧菌属被归类于弧菌科, 弧菌作为一种广泛分布于自然水域的条件致病菌, 易存在于杂食性海洋生物的体表和体内(陈梅等, 2002)。本研究结果显示银鲳的部分肠道样本中发现了弧菌, 但在养殖过程中并未发生疾病, 其可能作为一种正常菌群而存在于鱼类的肠道中(骆启豪等, 2021)。发光杆菌属细菌也属于弧菌科, 相关研究结果表明几种密切相关的发光杆菌菌株在大西洋鳕鱼的肠道中占绝大多数(Le Doujet et al, 2019)。而本研究结果发现健康银鲳肠道中也存在发光杆菌属细菌, 它们很可能也属于正常银鲳肠道微生物组成的一部分, 且通常不会导致银鲳发病。而在PDD感染后, 发光杆菌属在感染组银鲳肠道中的比例先小幅度减少, 随后呈现持续增加的趋势, 到感染后72 h时在鲳肠中肠和后肠中占据主要优势, 这很可能是由于PDD感染后, 随着其在银鲳体内增殖, 进入肠道, 影响了肠道中的其他发光杆菌属细菌的正常增殖, 最后PDD逐渐占据优势并在肠道内大量增殖。
上述研究结果表明, 银鲳感染PDD后, 病原体侵入肠道并可能与其他发光杆菌属产生了竞争。亲缘关系相近的细菌之间可能有着相似的生态位, 因此它们之间存在天然的竞争关系(查继伟, 2019)。Li等(2018)的发现也显示了生态位和适应性差异对病菌侵入的重要性。大肠杆菌和沙门氏菌同为肠杆菌科, Eberl等(2021)发现大肠杆菌可通过竞争限制性碳源来抑制鼠伤寒沙门氏菌的定植。银鲳肠道中PDD和其他发光杆菌属之间很有可能也存在这种竞争关系, 此外同为弧菌科的发光杆菌属和弧菌属之间可能也是如此, 24 h时银鲳中肠和后肠内弧菌属和发光杆菌属其中一属处于压倒性优势时, 另一属的丰富度则会更低; 同样地, PDD感染72 h后肠道内PDD的大量增殖很可能抑制了弧菌属, 导致其丰富度大幅下降。此外, 我们发现不同门的微生物之间也存在这种此消彼长的关系, 可观察到当肠道内蓝藻门较多时, 变形菌门就会较少; 变形菌门多, 则蓝藻门较少。王建建等(2014)曾在野生银鲳肠道检测出产消化酶的菌株, 主要以产纤维素酶和淀粉酶为主。因此推测此现象可能与变形菌门中含有产纤维素酶和淀粉酶等消化酶的菌株存在有关, 这些菌株较多时, 会减少蓝藻门的数量。
动物宿主肠道微生物的平衡对维持宿主健康及其抗病能力高低密切相关(De Schryver et al, 2014)。PDD感染后, 感染组银鲳中肠中厚壁菌门、拟杆菌门、蓝藻门的相对丰度增加, 变形菌门的相对丰度减少; 后肠中变形菌门、拟杆菌属、黏球菌门(Myxococcota)的相对丰度增加, 厚壁菌门、蓝藻门的相对丰度减少, 且感染组的中肠和后肠样本各时间点的细菌群落结构变化大多比对照组更为离散, 并随着时间的推移, 肠道细菌群落结构的离散性逐渐增大, 这可能与PDD的过度生长有关, 其改变了在患病银鲳肠道定植的其他微生物群的相对丰度, 破坏了肠道微生物的正常平衡。
一方面, 研究发生疾病时的肠道微生物构成与原本健康状态下肠道微生物群的差异或许可以为疾病的诊断提供新的视角, 然而, 目前PDD导致银鲳发病的机理, 特别是发病过程中如何引起肠道微生物结构改变的, 目前还尚不清楚。另一方面, 对于疾病的防治, 是否可以通过增强鱼类肠道微生物群落的稳定性, 从而预防病原体感染; 或是在发病过程中, 能否调节肠道菌群, 引入有益菌与病菌竞争, 进而抑制病原微生物的增殖, 使疾病向有利于宿主的方向转归。这些问题还需要更加深入地研究。
4 结论综上, 16S rRNA扩增子测序分析结果显示PDD感染可引起银鲳体内肠道微生物的动态变化。PDD的侵入及在银鲳肠道中的增殖打破了原本肠道微生物的竞争平衡。这些结果表明, 美人鱼发光杆菌病随着感染时间的延长而逐渐发展, PDD的过度生长与肠道微生物群落构成失调密切相关, 这显示了肠道微生物群落结构平衡的重要性。但是病原微生物与宿主及肠道微生物的关系, 以及它们之间如何相互影响, 有待进一步研究。
王佳, 徐善良, 2021. 浅谈银鲳鱼文化[J]. 中国水产, (10): 107-108. |
王建建, 高权新, 张晨捷, 等, 2014. 野生与养殖银鲳消化道菌群结构中产酶菌的对比分析[J]. 水产学报, 38(11): 1899-1909. |
王洪彬, 吴同垒, 张志强, 等, 2018. 美人鱼发光杆菌研究进展[J]. 黑龙江畜牧兽医, (9): 55-58. |
李建生, 胡芬, 严利平, 2014. 东海区银鲳资源合理利用的研究[J]. 自然资源学报, 29(8): 1420-1429. |
张飞, 苏永全, 王军, 等, 2012. 大黄鱼(Pseudosciaena crocea)源美人鱼发光杆菌(Pseudosciaenacrocea damselae)的分离鉴定及致病性研究[J]. 海洋与湖沼, 43(6): 1202-1208. |
陈梅, 李筠, 徐怀恕, 2002. 梭子蟹及牡蛎中致病性弧菌的研究[J]. 广西预防医学, 8(4): 193-196. |
孟晓林, 李文均, 聂国兴, 2019. 鱼类肠道菌群影响因子研究进展[J]. 水产学报, 43(1): 143-155. |
查继伟, 2019. 斑马鱼肠道菌群协同竞争关系对嗜水气单胞菌感染的影响[D]. 陕西杨凌: 西北农林科技大学.
|
骆启豪, 王卫军, 倪乐海, 等, 2021. 四种不同饵料对真蛸(Octopus vulgaris)生长与肠道微生物影响的研究[J]. 海洋与湖沼, 52(3): 729-737. |
徐胜威, 葛明峰, 许昊川, 等, 2021. 2020年宁波市主养海水鱼流行病学调查与药敏试验[J]. 宁波大学学报(理工版), 34(2): 109-114. |
徐善良, 王丹丽, 徐继林, 等, 2012. 东海银鲳(Pampus argenteus)、灰鲳(P. cinereus)和中国鲳(P. sinensis)肌肉主要营养成分分析与评价[J]. 海洋与湖沼, 43(4): 775-782. |
彭士明, 施兆鸿, 尹飞, 等, 2011. 利用碳氮稳定同位素技术分析东海银鲳食性[J]. 生态学杂志, 30(7): 1565-1569. |
BATES J M, MITTGE E, KUHLMAN J, et al, 2006. Distinct signals from the microbiota promote different aspects of zebrafish gut differentiation[J]. Developmental Biology, 297(2): 374-386. DOI:10.1016/j.ydbio.2006.05.006 |
BULGARELLI D, GARRIDO-OTER R, MÜNCH P C, et al, 2015. Structure and function of the bacterial root microbiota in wild and domesticated barley[J]. Cell Host & Microbe, 17(3): 392-403. |
DE SCHRYVER P, VADSTEIN O, 2014. Ecological theory as a foundation to control pathogenic invasion in aquaculture[J]. The ISME Journal, 8(12): 2360-2368. DOI:10.1038/ismej.2014.84 |
EBERL C, WEISS AS, JOCHUM LM, et al, 2021. E. coli enhance colonization resistance against Salmonella typhimurium by competing for galactitol, a context-dependent limiting carbon source[J]. Cell Host Microbe, 29(11): 1680-1692. DOI:10.1016/j.chom.2021.09.004 |
GANGULY S, PRASAD A, 2012. Microflora in fish digestive tract plays significant role in digestion and metabolism[J]. Reviews in Fish Biology and Fisheries, 22(1): 11-16. DOI:10.1007/s11160-011-9214-x |
GAO Z G, GUO B M, GAO R Y, et al, 2015. Microbiota disbiosis is associated with colorectal cancer[J]. Frontiers in Microbiology, 6: 20. |
GHANBARI M, KNEIFEL W, DOMIG K J, 2015. A new view of the fish gut microbiome: Advances from next-generation sequencing[J]. Aquaculture, 448: 464-475. DOI:10.1016/j.aquaculture.2015.06.033 |
LE DOUJET T, DE SANTI C, KLEMETSEN T, et al, 2019. Closely-related Photobacterium strains comprise the majority of bacteria in the gut of migrating Atlantic cod (Gadus morhua)[J]. Microbiome, 7(1): 64. DOI:10.1186/s40168-019-0681-y |
LI S P, TAN J Q, YANG X, et al, 2018. Niche and fitness differences determine invasion success and impact in laboratory bacterial communities[J]. The ISME Journal, 13(2): 402-412. |
LI T T, LONG M, JI C, et al, 2016. Alterations of the gut microbiome of largemouth bronze gudgeon (Coreius guichenoti) suffering from furunculosis[J]. Scientific Reports, 6(1): 30606. DOI:10.1038/srep30606 |
LI B, ZHANG X X, GUO F, et al, 2013. Characterization of tetracycline resistant bacterial community in saline activated sludge using batch stress incubation with high-throughput sequencing analysis[J]. Water Research, 47(13): 4207-4216. DOI:10.1016/j.watres.2013.04.021 |
LOZUPONE C, KNIGHT R, 2005. UniFrac: a new phylogenetic method for comparing microbial communities[J]. Applied and Environmental Microbiology, 71(12): 8228-8235. DOI:10.1128/AEM.71.12.8228-8235.2005 |
LUNDBERG D S, YOURSTONE S, MIECZKOWSKI P, et al, 2013. Practical innovations for high-throughput amplicon sequencing[J]. Nature Methods, 10(10): 999-1002. DOI:10.1038/nmeth.2634 |
MAHDI GHANBARI, WOLFGANG KNEIFEL, KONRAD J DOMIG, 2015. A new view of the fish gut microbiome: Advances from next-generation sequencing[J]. Aquaculture, 448: 464-475. DOI:10.1016/j.aquaculture.2015.06.033 |
NAWAZ M, LI X L, YUE X Y, et al, 2022. Transcriptome profiling and differential expression analysis of the immune-related genes during the acute phase of infection with Photobacterium damselae subsp. damselae in silver pomfret (Pampus argenteus)[J]. Fish & Shellfish Immunology, 131: 342-348. |
ROESELERS G, MITTGE E K, STEPHENS W Z, et al, 2011. Evidence for a core gut microbiota in the zebrafish[J]. The ISME Journal, 5(10): 1595-1608. DOI:10.1038/ismej.2011.38 |
SHE R, LI T T, LUO D, et al, 2017. Changes in the intestinal microbiota of gibel carp (Carassius gibelio) associated with Cyprinid herpesvirus 2 (CyHV-2) infection[J]. Current Microbiology, 74(10): 1130-1136. DOI:10.1007/s00284-017-1294-y |
SOMMER F, BÄCKHED F, 2013. The gut microbiota—masters of host development and physiology[J]. Nature Reviews Microbiology, 11(4): 227-238. DOI:10.1038/nrmicro2974 |
SULLAM K E, ESSINGER S D, LOZUPONE C A, et al, 2012. Environmental and ecological factors that shape the gut bacterial communities of fish: a meta-analysis[J]. Molecular Ecology, 21(13): 3363-3378. DOI:10.1111/j.1365-294X.2012.05552.x |
TAO Z, SHEN C, ZHOU S M, et al, 2018. An outbreak of Photobacterium damselae subsp. damselae infection in cultured silver pomfret Pampus argenteus in Eastern China[J]. Aquaculture, 492: 201-205. DOI:10.1016/j.aquaculture.2018.04.013 |
TARNECKI A M, BURGOS F A, RAY C L, et al, 2017. Fish intestinal microbiome: diversity and symbiosis unravelled by metagenomics[J]. Journal of Applied Microbiology, 123(1): 2-17. DOI:10.1111/jam.13415 |
WU S G, GAO T H, ZHENG Y Z, et al, 2010. Microbial diversity of intestinal contents and mucus in yellow catfish (Pelteobagrus fulvidraco)[J]. Aquaculture, 303(1/2/3/4): 1-7. |
WU S G, TIAN J Y, GATESOUPE F J, et al, 2013. Intestinal microbiota of gibel carp (Carassius auratus gibelio) and its origin as revealed by 454 pyrosequencing[J]. World Journal of Microbiology and Biotechnology, 29(9): 1585-1595. DOI:10.1007/s11274-013-1322-4 |
 2024, Vol. 55
2024, Vol. 55


