中国海洋湖沼学会主办。
文章信息
- 林筱钧, 许净, 郑江, 鄢庆枇, 高得骥, 范云庭, 黄将远. 2023.
- LIN Xiao-Jun, XU Jing, ZHENG Jiang, YAN Qing-Pi, GAO De-Ji, FAN Yun-Ting, HUANG Jiang-Yuan. 2023.
- 变形假单胞菌(Pseudomonas plecoglossicida)核酸适配体的SELEX筛选
- SELECTION OF APTAMERS OF PSEUDOMONAS PLECOGLOSSICIDA BY SELEX
- 海洋与湖沼, 54(2): 583-590
- Oceanologia et Limnologia Sinica, 54(2): 583-590.
- http://dx.doi.org/10.11693/hyhz20220700194
文章历史
-
收稿日期:2022-07-25
收修改稿日期:2022-09-21
2. 鳗鲡现代产业技术教育部工程研究中心 福建厦门 361021
2. Engineering Research Center of the Modern Technology for Eel Industry, Ministry of Education, Xiamen 361021, China
变形假单胞菌(Pseudomonas plecoglossicida)属于假单胞菌科, 假单胞菌属, 杀香鱼假单胞菌种, 是一种直或微弯、不呈螺旋状的革兰氏阴性杆菌(陈卓等, 2018)。该菌广泛存在于土壤环境和自然水体中, 能感染香鱼、大黄鱼、小黄鱼、虹鳟和黑鲷等多种鱼类, 是一种常见的条件致病菌(胡娇等, 2014)。对其进行快速的分析检测是其病害防控的前提和基础。目前对变形假单胞菌的检测主要是采用基于其特定基因的PCR检测法(Izumi et al, 2007; 袁雪梅等, 2015; 周涛等, 2015; 崔晓莹等, 2019), 该方法对仪器要求较高, 检测操作相对复杂, 难以实现现场的快速检测。
核酸适配体(Aptamer)是通过指数富集配体的系统进化技术(Systematic Evolution of Ligands by Exponential Enrichment), 即SELEX技术, 筛选出的对靶目标具有较高亲和特异性的寡核苷酸分子(李亚楠等, 2017)。核酸适配体具有茎、环、凸起、发夹、假结、三链体或四链体等复杂的三维结构, 使其可以很好地结合细菌、糖类、肽类、抗生素和蛋白质等多种靶目标(Reining et al, 2013; 杨歌等, 2020)。同时, 它还具有分子量小、结构简单, 易修饰、稳定性高、组织渗透率高等优点(Muhammad et al, 2021), 在微生物检测、生物传感、医学诊断等多个领域得到广泛应用(Crooks, 2014; Zheng et al, 2015b; Hu et al, 2016; Beloborodov et al, 2018; Cheung et al, 2018; Feng et al, 2018; Du et al, 2022; Pandit et al, 2022)。
本文以变形假单胞菌为靶目标, 通过SELEX筛选、高通量测序、亲和特异性的验证等过程获得能特异性结合变形假单胞菌的核酸适配体, 并对其亲和常数和最大亲和力进行了测定, 对其二级结构进行了模拟分析, 相关研究将为变形假单胞菌的检测及其病害防控奠定重要的基础。
1 材料与方法 1.1 材料 1.1.1 随机ssDNA文库和引物随机ssDNA文库序列为5′-TCAGTCGCTTCGCCGTCTCC TTC(N35) GCACAAGAGGGAGACCCCAGAGGG-3′, 中间的N35是35个随机碱基, 文库总长度为82 nt。不对称PCR的上游引物P1: 5′-TCAGTCGCTTCGCCGTCT CCTTC-3′; 下游引物P2: 5′-CCCTCTGGGGTCTCC CTCTTGTGC-3′。随机ssDNA文库和引物均由上海生工生物工程有限公司(简称上海生工)合成。
1.1.2 实验用菌和培养基实验中用到的变形假单胞菌(P. plecoglossicida)、铜绿假单胞菌(Pseudomonas aeruginosa)、大肠杆菌(Escherichia coli)、溶藻弧菌(Vibrio alginlyticus)、哈维氏弧菌(Vibrio harveyi)、迟钝爱德华氏菌(Edwardsiella tarda)、嗜水气单胞菌(Aeromonas hydrophila)均来自集美大学病原微生物实验室鉴定并提供。其中变性假单胞菌、铜绿假单胞菌和大肠杆菌用LB培养基培养, 其他菌则用TSB培养基培养。
1.1.3 缓冲液20×结合缓冲液(pH 7.4, 100 mL): 将NaCl 5.844 g、KCl 3.725 g、Tris-HCl 6.06 g、MgCl2·6H2O 2.033 g全部溶于超纯水中, 100 mL定容, 调节pH至7.4, 使用时用无菌超纯水稀释为2×和1×结合缓冲液; TE缓冲溶液(pH 8.0, 100 mL): 取1 mol/L Tris-HCl 1 mL、0.5 mol/L EDTA 0.2 mL混合均匀, 定容至100 mL, 调节pH至8.0。
1.2 SELEX筛选将100 μL、2 μmol/L的随机ssDNA文库, 95 ℃金属浴加热5 min, 4 ℃冰浴10 min后, 与1× 108 CFU/mL的变形假单胞菌100 μL混合, 在28 ℃下100 r/min摇床孵育结合2 h, 然后6 000 r/min离心5 min, 用200 μL的1×结合缓冲溶液洗涤1次, 洗去不与目标菌变形假单胞菌结合的ssDNA, 6 000 r/min离心5 min后弃上清, 用100 μL的1×结合缓冲溶液重悬菌沉淀, 此时得到的菌悬液就是含有结合ssDNA的变形假单胞菌。将上述菌悬液95 ℃加热5 min, 冷却至室温后12 000 r/min离心10 min, 取上清液, 此时上清液中就是筛选获得的能与变形假单胞菌结合的ssDNA。以筛选出的ssDNA为模板, 进行不对称PCR反应, 25 μL的反应体系如下: 2 μL的引物P1 (10 μmol/L), 2 μL的引物P2 (0.2 μmol/L), 4 μL的模板(ssDNA)、12.5 μL的2×Super Pfr Master Mix (PCR缓冲预混体系)、4.5 μL的ddH2O; 反应参数为: 98 ℃预变性3 min, 98 ℃变性10 s, 60 ℃退火30 s, 75 ℃延伸15 s, 共30个循环, 最后72 ℃延伸5 min, 4 ℃保温。此为第一轮筛选。得到PCR产物即为筛选产物, 也是下一轮筛选的初始文库。
之后按上述步骤再进行6轮筛选。其中在第6轮和第7轮依次加入100 μL、1×108 CFU/mL的铜绿假单胞菌、大肠杆菌、溶藻弧菌、哈维氏弧菌、迟钝爱德华氏菌和嗜水气单胞菌等非目标菌进行反筛, 第6轮非目标菌和目标菌都与筛选文库结合1 h, 第7轮时非目标菌与筛选文库要结合2 h, 而目标菌与筛选文库结合时间则缩短至30 min, 反筛过程中, 保留上清, 弃去含有非目标菌的菌沉淀。最后将总共7轮的PCR产物送上海生工公司进行高通量测序, 然后对测序序列进行统计分析, 重点选择测序结果中出现次数大于等于2次的高频序列进行分析研究。
1.3 核酸适配体亲和力的测定参考彭英林等(2020)方法进行测定, 具体如下: 将100 μL、2 μmol/L的核酸适配体95 ℃金属浴加热变性5 min, 再4 ℃冰浴10 min, 然后和100 μL、5×108 CFU/mL的变形假单胞菌混合, 作为实验组; 另外, 用100 μL的2×结合缓冲液代替核酸适配体, 和100 μL、5×108 CFU/mL的变形假单胞菌混合, 作为对照组。每组3个平行。实验组和对照组一同在28 ℃、100 r/min摇床孵育30 min。然后6 000 r/min离心5 min, 用1×结合缓冲溶液洗涤菌沉淀1次, 再用100 μL 1×结合缓冲溶液重悬菌沉淀。将上述实验组和对照组均放置于金属浴中, 95 ℃加热5 min, 恢复室温后15 000 r/min离心10 min, 取上清用超微量分光光度计测定实验组和对照组的ssDNA值, 该核酸适配体对变形假单胞菌的亲和力=实验组ssDNA值-对照组ssDNA值。
1.4 核酸适配体对变形假单胞菌的亲和特异性按1.3亲和力的测定方法, 分别测定不同核酸适配体对目标菌变形假单胞菌和非目标菌(铜绿假单胞菌、大肠杆菌、溶藻弧菌、哈维氏弧菌、迟钝爱德华氏菌和嗜水气单胞菌)的亲和力, 进而获得各核酸适配体对变形假单胞菌的亲和特异性。
1.5 核酸适配体亲和常数Kd和最大亲和力Am的测定先用2×结合缓冲溶液将10 μmol/L的核酸适配体分别稀释至10、20、40、60、80、100、120、140、200、250和300 nmol/L。各取100 μL各个浓度的核酸适配体于95 ℃金属浴加热变性5 min, 冰浴10 min后, 与100 μL、5×108 CFU/mL的变形假单胞菌混合, 作为实验组; 另将100 μL的2×结合缓冲液和100 μL、5×108 CFU/mL的变形假单胞菌混合, 作为对照组。每组3个平行。后续操作按照1.3的亲和力测定方法进行孵育结合、离心、洗涤、重悬、测定ssDNA值, 最后得到相应浓度的核酸适配体的亲和力=实验组ssDNA值-对照组ssDNA值。根据核酸适配体的分子量将亲和力的单位统一成nmol/L, 然后以核酸适配体的浓度为横坐标, 以核酸适配体的亲和力为纵坐标, 参考Zheng等(2015a)的方法, 采用Origin软件进行非线性拟合, 拟合函数为双曲线函数hyperbl, 曲线公式为
 (1)
(1)式中, Y表示亲和力, X表示核酸适配体的摩尔浓度, 根据拟合方程即可得到Kd值和Am值。
1.6 核酸适配体的二级结构模拟参考许净等(2021)的方法进行核酸适配体二级结构的模拟。通过RNAstructure网站(http://rna.urmc.rochester.edu/RNAstructureWeb/)进行在线预测, 选择核酸类型为DNA, 其他参数为默认值, 选择最大可能性的二级结构, 即MaxExpect二级结构作为各核酸适配体序列的二级结构。
1.7 统计分析采用t-检验函数进行组间的差异分析, 其中P < 0.05为显著差异, P < 0.01为极显著差异。
2 结果 2.1 核酸适配体候选序列的选择如表 1所示, 7轮筛选产物经高通量测序后, 对测序结果中出现2次以上的高频序列进行统计, 结果表明, 随着筛选进行, 高频序列的数量和占比都在逐步上升, 说明具有高亲和力、高竞争力的高频序列在逐步富集。7轮筛选共得到了707条高频序列。表 2列出了7轮筛选中出现轮数最多、总的出现次数最多的15个高频序列。由于高频序列众多, 无法全部验证, 因此, 研究中选择其中5个高频序列(#1、#2、#3、#4、#5)进行亲和特异性验证, 其中#1、#2是全部7轮筛选中都出现的高频序列, #4、#5则是出现了6轮而且是6轮中出现次数最多的两个序列, #3则是序列长度最短且是相同长度下出现次数最多的高频序列。
| 筛选轮数 | 总序列数/个 | 高频序列数/个 | 占比/% |
| 1 | 84 993 | 2 | 0.002 |
| 2 | 99 493 | 89 | 0.089 |
| 3 | 62 576 | 105 | 0.168 |
| 4 | 85 100 | 111 | 0.130 |
| 5 | 87 527 | 143 | 0.163 |
| 6 | 47 211 | 375 | 0.794 |
| 7 | 49 918 | 489 | 0.980 |
| 顺序 | 名称 | 中间序列(5′~3′) | 总出现次数 | 出现轮数 |
| 1 | #1 | AGCGGGATGAGGGAGTAGGAGGGCCACAGTGGACT | 85 763 | 7 |
| 2 | #2 | AGCGGGATGAGGGAGTAGGAGGGCCACAGTGTACT | 120 | 7 |
| 3 | #4 | AGCGGGATGAGGGAGTAGGAGGGCCACAGTAGACT | 549 | 6 |
| 4 | #5 | AGCGGGATGAGGGAGTAGGAGGGCCACAGTGAACT | 535 | 6 |
| 5 | #13 | AGCGGGATGAGGGAGTAGGAGGGCCACAATGGACT | 519 | 6 |
| 6 | #7 | AGCGGGGATGAGGGAGTAGGAGGGCCACAGTGGACT | 411 | 6 |
| 7 | #28 | AGCGGGATGAGGGAGTAGGAGGACCACAGTGGACT | 387 | 6 |
| 8 | #8 | AGCGGGATGAGGGAGTAGGAGAGCCACAGTGGACT | 370 | 6 |
| 9 | #16 | AGCGGGATGAGGGAGTAGGAAGGCCACAGTGGACT | 352 | 6 |
| 10 | #52 | AGCGGGATGATGGAGTAGGAGGGCCACAGTGGACT | 332 | 6 |
| 11 | #3 | AGCCGGGGTGGTCAGTAGGAGCA | 331 | 6 |
| 12 | #10 | AGCGGGATGAGGGAGTAGAAGGGCCACAGTGGACT | 273 | 6 |
| 13 | #6 | AGCGGGATGAGGGAGTAAGAGGGCCACAGTGGACT | 270 | 6 |
| 14 | #9 | AAGCGGGATGAGGGAGTAGGAGGGCCACAGTGGACT | 270 | 6 |
| 15 | #18 | AGCGGGATGAGGGGAGTAGGAGGGCCACAGTGGACT | 247 | 6 |
五个核酸适配体(#1、#2、#3、#4、#5)对目标菌变形假单胞菌(Pp), 以及非目标菌嗜水气单胞菌(Ah)、溶藻弧菌(Va)、哈维氏弧菌(Vh)、大肠杆菌(Ec)、迟钝爱德华氏菌(Et)、铜绿假单胞菌(Pa)的亲和力如图 1所示。由图 1可以看出, 这些适配体对变形假单胞菌的亲和力均极显著高于其他菌(P < 0.01), 对变形假单胞菌的平均亲和力都是其他菌的10倍, 表明这些适配体均对变形假单胞菌有较好的亲和特异性。比较而言, #3核酸适配体的亲和力更高, 特异性也相对更好些。
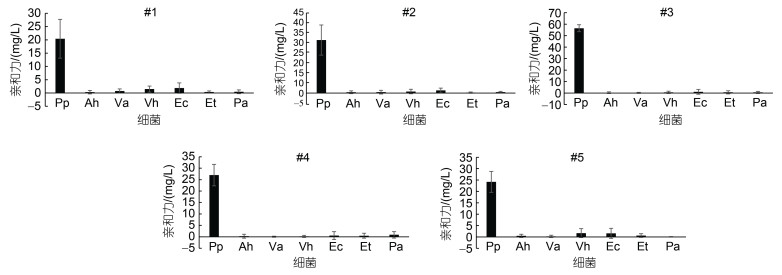 |
| 图 1 核酸适配体对变形假单胞菌的亲和特异性 Fig. 1 Affinity and specificity of aptamers against P. plecoglossicida 注: Pp: 变形假单胞菌; Ah: 嗜水气单胞菌; Va: 溶藻弧菌; Vh: 哈维氏弧菌; Ec: 大肠杆菌; Et: 迟钝爱德华氏菌; Pa: 铜绿假单胞菌; 图中**表示适配体对该菌的亲和力达到极显著水平(P < 0.01) |
五个核酸适配体的Kd和Am如表 3, 相应的饱和曲线如图 2。从中可以看出, 各适配体对变形假单胞菌均有较好的结合能力。其中, Kd最小, 结合能力最强的是#2核酸适配体, 但其Am只处于中间; Am最高的是#3核酸适配体, 而且其Kd排第二, 因此最终体现出的是#3核酸适配体的表观亲和力最高。
| 适配体 | 亲和常数Kd/(nmol/L) | 最大亲和力Am/(nmol/L) |
| #1 | 25.77±6.11 | 789.23±38.46 |
| #2 | 7.14±1.86 | 1 392.27±58.03 |
| #3 | 19.59±3.01 | 3 012.50±106.19 |
| #4 | 83.22±22.45 | 1 457.99±171.32 |
| #5 | 34.31±8.76 | 1 070.91±70.16 |
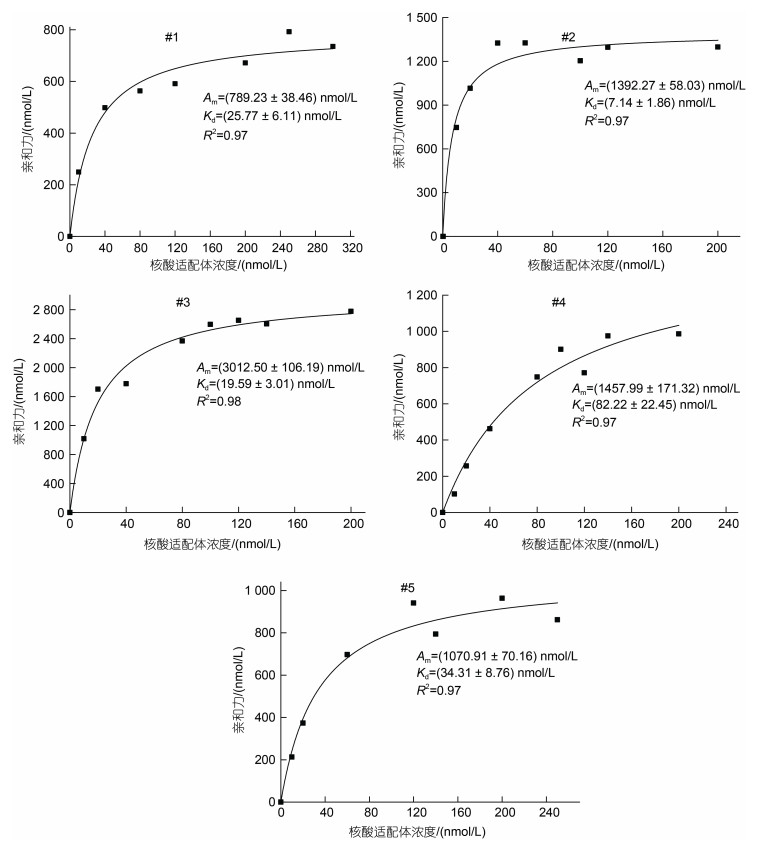 |
| 图 2 变形假单胞菌各个核酸适配体的亲和常数饱和曲线 Fig. 2 Saturation curve of the affinity constant of each aptamer of P. plecoglossicida |
5个核酸适配体的二级结构中均含有多个不同大小的闭合环状结构(如图 3)。表 4对这些闭合环进行了总结分析, 可以发现, 每个适配体都含有4~5个闭合环, 每个环由6~26核苷酸组成, 根据环中的核苷酸数量可以把这些环大致分为三种: 小型环(由8个以下核苷酸构成)、中型环(由9~21个核苷酸构成)和大型环(由22个以上的核苷酸构成)。5个适配体中, 每个适配体都有3个小型环, 其次是中型环, 只有#1、#2、#5这三个适配体有, 大型环则只有#3、#4适配体有。已知各适配体的亲和常数的大小依次为: #4 > #5、#1、#3 > #2, 表明各适配体对靶标的结合能力依次增强。由于这5个适配体都有3个小型环, 这些适配体对靶标结合能力的差异似乎与小型环关系不大, 而可能主要取决于适配体中的中大型环。当然, 这个结合能力还取决于与适配体相互作用的蛋白, 中大型环可能是适配体上主要参与互作的结构。
 |
| 图 3 核酸适配体二级结构模拟图 Fig. 3 Simulant secondary structures of the aptamers |
| 核酸适配体 | 环的数量 | 各个环的核苷酸数量 | 亲和常数 Kd/(nmol/L) |
| #1 | 5 | 6、8、8、9、10 | 25.77±6.11 |
| #2 | 4 | 6、7、8、12 | 7.14±1.86 |
| #3 | 4 | 6、7、7、22 | 19.59±3.01 |
| #4 | 4 | 6、6、8、26 | 83.22±22.45 |
| #5 | 4 | 6、7、8、12 | 34.31±8.76 |
| 注: 粗体数字表示中大型环 | |||
高通量测序得到的序列有成千上万条, 90%以上的序列都是出现1次的非高频序列, 从这些非高频序列中挑选亲和力高的候选适配体序列需要进行大量的验证实验, 不仅费时、费力, 验证成本也非常高。高频序列都是在筛选进化中具有竞争优势的序列, 不仅数量少, 只占总序列的10%不到, 而且大概率是对靶目标有较好亲和力的核酸适配体序列(郑江等, 2014, 2022), 因此从高频序列中挑选核酸适配体的候选序列进行验证研究, 将能大大节约时间和成本, 提高筛选的效率。
随着筛选进行, 每轮高频序列的数量和占比不断增加, 这很可能是因为在筛选竞争中, 在总序列中占比90%以上的、大量亲和力弱的非高频序列逐渐被淘汰, 而亲和力强的非高频序列则逐渐进化为高频序列, 从而导致含有大量核酸适配体的、有较高亲和力的高频序列在筛选竞争中逐渐占据优势。因此, 出现轮数较多、出现次数较多的序列则大概率也是竞争力较高、亲和力较高的序列, 从这些序列中挑选核酸适配体将有较高的成功率。亲和特异性的验证结果也表明, 出现轮数和次数较多的5个高频序列都是亲和特异性较好的核酸适配体。
亲和常数Kd和最大亲和力Am是表征核酸适配体亲和力的重要参数, 其他条件相同的前提下, Kd越低, Am越大, 相应的表观亲和力也越大。Kd反映的是与靶目标的结合能力, Am反映的是能与靶目标结合的最大数量。从目前已有的报道看, 病原菌核酸适配体的Kd大多介于10~300 nmol/L之间(Savory et al, 2014; Soundy et al, 2017; 郑江等, 2022)。其中, 同为假单胞菌属的铜绿假单胞菌, 其核酸适配体的Kd介于10~ 60 nmol/L, 最低为(12.5±2.4) nmol/L (Du et al, 2022)。本研究筛选到的变形假单胞菌的核酸适配体, 其Kd则介于5~110 nmol/L, 与铜绿假单胞菌的基本处于同一数量级, 但最低的#2核酸适配体, 其亲和常数Kd可达(7.14±1.86) nmol/L, 比铜绿假单胞菌的还低, 说明本研究筛选得到的核酸适配体, 其亲和常数还是较为理想的。目前有关最大亲和力Am的数据报道较少, 很可能是因为通常情况下同一靶目标的不同适配体的Am差异不是太大, 对表观亲和力的影响不如Kd大, 如本研究中Am的最大差异不到4倍, 而Kd的最大差异达到了11倍以上, 因此Kd的影响是决定性的。但有研究发现, 对于一些Kd差异不太大的核酸适配体, 其Am对表观亲和力的影响是不可忽略的(郑江等, 2022)。因此, 研究中同时考虑Kd和Am对表观亲和力的影响应该是比较稳妥和全面的。
研究中还发现, 长度最短、分子量最小的#3核酸适配体, 其Am是5个核酸适配体中最大的, 是其他核酸适配体Am的2~4倍, 而且其Kd也较低, 排在第二位, 因此最终#3核酸适配体的表观亲和力是最高的。有研究表明, 核酸适配体被截短后, 亲和力会明显提高(Zheng et al, 2015a), 在鳗弧菌核酸适配体的研究中也发现, 序列最短的H1适配体, 其亲和常数Kd值也是最低的(郑江等, 2022), 因此, 序列较短、分子量较小的核酸适配体, 可能在与靶目标的结合中有一定优势, 不过这一判断还需进一步研究确认。
核酸适配体的二级结构等空间结构是其与靶目标特异性结合的基础(汤学敏等, 2012), 因此研究核酸适配体的二级结构特点, 对于研究其与靶目标的特异性结合机制具有重要意义。有研究表明, 核酸适配体中环的大小是与其互作蛋白的空间结构域是相匹配的(郑江等, 2022)。有研究发现适配体二级结构中的不同环区可以联合参与靶标的识别与结合, 两个颈环的适配体要比一个颈环的适配体有更好的稳定性和亲和特异性(Bhardwaj et al, 2019)。本研究则表明, 核酸适配体二级结构中存在多个大小不同的环, 其中的中大型环可能与适配体的结合能力密切相关。
4 结论通过SELEX筛选获得了变形假单胞菌的5个核酸适配体(#1、#2、#3、#4、#5), 这5个核酸适配体对变形假单胞菌的亲和力显著高于嗜水气单胞菌、铜绿假单胞菌、溶藻弧菌、哈维氏弧菌、大肠杆菌、迟钝爱德华氏菌等非目标菌(P < 0.01), 是非目标菌的4~11倍。其中, #3核酸适配体的表观亲和力更高, 特异性也更好。测定了5个核酸适配体(#1、#2、#3、#4、#5)的亲和常数Kd和最大亲和力Am。并对其二级结构进行了分析研究, 发现二级结构含有多个大、中、小型的闭合环, 推测其中的大、中型环可能与适配体的结合能力密切相关。
汤学敏, 郑江, 郝聚敏, 等, 2012. 现代微生物识别技术在水产养殖环境研究中的应用. 微生物学通报, 39(6): 835-842 DOI:10.13344/j.microbiol.china.2012.06.013 |
许净, 刘慧敏, 林筱钧, 等, 2021. 鳗弧菌(Vibrio anguillarum)核酸适配体筛选中高频序列的进化多样性研究. 海洋与湖沼, 52(5): 1315-1322 |
李亚楠, 赵洁, 张傲哲, 等, 2017. 核酸适配体的体外筛选方法的最新研究进展. 生物技术通报, 33(4): 78-82 |
杨歌, 赵毅, 韩诗邈, 等, 2020. 毛细管电泳法筛选脱铁转铁蛋白的核酸适配体及筛选影响因素分析. 分析化学, 48(5): 632-641 DOI:10.19756/j.issn.0253-3820.191694 |
陈卓, 施慧, 谢建军, 等, 2018. 大黄鱼人工感染杀香鱼假单胞菌的病理形态学及病原分布的初步研究. 浙江海洋大学学报(自然科学版), 37(6): 483-488, 493 DOI:10.3969/j.issn.1008-830X.2018.06.002 |
周涛, 张继挺, 王国良, 2015. 香鱼假单胞菌SYBR Green Ⅰ荧光定量PCR检测方法的建立. 中国预防兽医学报, 37(1): 35-39 DOI:10.3969/j.issn.1008-0589.2015.01.09 |
郑江, 郝聚敏, 宋林生, 等, 2014. 哈维氏弧菌适配子的SELEX筛选及其亲和特异性研究. 生物化学与生物物理进展, 41(7): 704-711 |
郑江, 刘慧敏, 黄力行, 等, 2022. 鳗弧菌(Vibrio anguillarum)核酸适配体的筛选及其结合蛋白的分离鉴定. 生物化学与生物物理进展, 49(1): 250-261 |
胡娇, 张飞, 徐晓津, 等, 2014. 大黄鱼(Pseudosciaena crocea)内脏白点病病原分离鉴定及致病性研究. 海洋与湖沼, 45(2): 409-417 |
袁雪梅, 葛明峰, 安树伟, 等, 2015. 大黄鱼致病香鱼假单胞菌对环境因子的响应及其感染检测的分析. 应用海洋学学报, 34(4): 549-553 |
崔晓莹, 李泽宇, 李完波, 等, 2019. 一种定量检测大黄鱼感染变形假单胞菌方法的建立与应用. 集美大学学报(自然科学版), 24(5): 328-334 |
彭英林, 刘慧敏, 鄢庆枇, 等, 2020. 基于单链DNA浓度法的哈维氏弧菌核酸适配体亲和特异性研究. 中国水产科学, 27(5): 598-604 |
BELOBORODOV S S, BAO J Y, KRYLOVA S M, et al, 2018. Aptamer facilitated purification of functional proteins. Journal of Chromatography B, 1073: 201-206 DOI:10.1016/j.jchromb.2017.12.024 |
BHARDWAJ J, CHAUDHARY N, KIM H, et al, 2019. Subtyping of influenza A H1N1 virus using a label-free electrochemical biosensor based on the DNA aptamer targeting the stem region of HA protein. Analytica Chimica Acta, 1064: 94-103 |
CHEUNG Y W, DIRKZWAGER R M, WONG W C, et al, 2018. Aptamer-mediated Plasmodium-specific diagnosis of malaria. Biochimie, 145: 131-136 |
CROOKS R M, 2014. Detective work on drug dosage. Nature, 505(7482): 165-166 |
DU J, CHEN X, LIU K, et al, 2022. Dual recognition and highly sensitive detection of Listeria monocytogenes in food by fluorescence enhancement effect based on Fe3O4@ZIF-8-aptamer. Sensors and Actuators B: Chemical, 360: 131654 |
FENG J L, DAI Z Y, TIAN X L, et al, 2018. Detection of Listeria monocytogenes based on combined aptamers magnetic capture and loop-mediated isothermal amplification. Food Control, 85: 443-452 |
HU G Y, TAO F, WANG W, et al, 2016. Prognostic value of microRNA-21 in pancreatic ductal adenocarcinoma: a meta-analysis. World Journal of Surgical Oncology, 14(1): 82 |
IZUMI S, YAMAMOTO M, SUZUKI K, et al, 2007. Identification and detection of Pseudomonas plecoglossicida isolates with PCR primers targeting the gyrB region. Journal of Fish Diseases, 30(7): 391-397 |
MUHAMMAD M, HUANG Q, 2021. A review of aptamer-based SERS biosensors: design strategies and applications. Talanta, 227: 122188 |
PANDIT C, ALAJANGI H K, SINGH J, et al, 2022. Development of magnetic nanoparticle assisted aptamer-quantum dot based biosensor for the detection of Escherichia coli in water samples. Science of the Total Environment, 831: 154857 |
REINING A, NOZINOVIC S, SCHLEPCKOW K, et al, 2013. Three-state mechanism couples ligand and temperature sensing in riboswitches. Nature, 499(7458): 355-359 |
SAVORY N, NZAKIZWANAYO J, ABE K, et al, 2014. Selection of DNA aptamers against uropathogenic Escherichia coli NSM59 by quantitative PCR controlled Cell-SELEX. Journal of Microbiological Methods, 104: 94-100 |
SOUNDY J, DAY D, 2017. Selection of DNA aptamers specific for live Pseudomonas aeruginosa. PLoS One, 12(9): e0185385 |
ZHENG X, HU B, GAO S X, et al, 2015a. A saxitoxin-binding aptamer with higher affinity and inhibitory activity optimized by rational site-directed mutagenesis and truncation. Toxicon, 101: 41-47 |
ZHENG J, TANG X M, WU R X, et al, 2015b. Identification and characteristics of aptamers against inactivated Vibrio alginolyticus. LWT-Food Science and Technology, 64(2): 1138-1142 |
 2023, Vol. 54
2023, Vol. 54


