中国海洋湖沼学会主办。
文章信息
- 马晖, 王彩凤, 李桂玲, 李健, 刘静雯. 2023.
- MA Hui, WANG Cai-Feng, LI Gui-Ling, LI Jian, LIU Jing-Wen. 2023.
- 基于脂质组学的海洋颗石藻病毒甾醇去饱和酶及脂肪酸去饱和酶基因功能分析
- FUNCTIONAL ANALYSIS OF STEROL DESATURASE AND FATTY ACID DESATURASE GENES FROM COCCOLITHOPHORES EMILIANIA HUXLEYI VIRUS BASED ON LIPIDOMICS
- 海洋与湖沼, 54(1): 125-138
- Oceanologia et Limnologia Sinica, 54(1): 125-138.
- http://dx.doi.org/10.11693/hyhz20220400085
文章历史
-
收稿日期:2022-04-04
收修改稿日期:2022-04-28
颗石藻(Coccolithophores)是一种全球广泛分布且具有重要生态功能的真核微型浮游植物, 其中赫氏颗石藻Emiliania huxleyi (Eh)具有形成“球石粒”和高产二甲基硫化物(DMSP)的能力, 且几乎每年都在大洋(尤其是高纬度海域)中形成大面积赤潮(Daniels et al, 2018), 该藻赤潮迅速大规模消亡被证实是特异性病毒(E. huxleyi virus: EhVs)感染和裂解所致(Ruiz et al, 2017)。因此, EhV-Eh的互作过程是影响海洋碳、硫生物地化循环及气候变化的重要环节, 并在重塑海洋生物群落结构、地质演变中具有重要地位(Sheyn et al, 2018; Johns et al, 2019)。
在共进化过程中, EhV通过基因水平转移从宿主基因组中“劫获”了一组鞘脂从头生物合成途径中的关键酶基因, 如丝氨酸棕榈酰转移酶(SPT)、甾醇去饱和酶(SD)、脂肪酸去饱和酶(FAD)、脂类磷酸酶(LPP)、糖基转移酶(GT)以及二氢神经酰胺合成酶/长寿保障基因类蛋白(Cers/LAG1)等, 并在一定程度上掌控宿主鞘脂代谢, 为病毒谋福利(Wilson et al, 2005; Lindell et al, 2007; Rose et al, 2014; Nissimov et al, 2017)。鞘脂代谢相关酶基因广泛存在于高等动植物及酵母基因组中, 但在海洋病毒中尚属首次发现(Wilson et al, 2005)。作为一种大型双链DNA病毒, EhV感染颗石藻显著影响宿主脂质代谢, 特别是深度重塑了宿主鞘脂代谢过程以满足自身的代谢需求(Rosenwasser et al, 2014; Malitsky et al, 2016; Zeng et al, 2019)。鞘脂既是真核细胞膜的重要结构成分, 也是膜上“脂筏”的主要功能成分, 参与物质内吞、跨膜信号转导等多种细胞过程, 被认为是病原微生物进出细胞的一种门户(Rose et al, 2014)。以色列学者2016年首次详细报道了EhV编码的丝氨酸棕榈酰转移酶vSPT(鞘脂从头生物合成途径中的第一个关键限速酶)的功能, 在感染的藻宿主细胞中该酶催化合成病毒特有的新型鞘糖脂(vGSLs), 该物质是病毒组装的必要物质基础(Ziv et al, 2016)。另外, vGSLs还能作为信号分子诱发活性氧(reactive oxygen species, ROS) H2O2 (Sheyn et al, 2016)和一氧化氮(Vardi et al, 2009; 2012)的产生, 进而启动宿主依赖于凋亡特征蛋白酶metacaspase的程序性细胞死亡过程, 最终裂解藻细胞释放子代病毒粒子(Bidle et al, 2007; Liu et al, 2018; 苏金净等, 2019)。除此之外, vGSLs还可能参与调控颗石藻复杂的单倍体/二倍体生活史过程, 使某些宿主细胞停留于二倍体世代以利于病毒的感染和增殖(Hunter et al, 2015)。目前, 除了EhV-SPT基因外, EhV基因组中其他鞘脂代谢相关酶基因的功能尚不清楚。
在酵母细胞中, 甾醇去饱和酶主要包括C22-SD甾醇去饱和酶和C-5甾醇去饱和酶, C22-SD甾醇去饱和酶由ERG5基因编码, 是酵母细胞麦角甾醇生物合成的限速酶, 属于细胞色素P-450 (CYP450)的一种(Skaggs et al, 1996)。C22-SD能够在甾醇侧链中引入一个双键, 催化酵母麦角甾醇-5, 7, 24(28)-三烯醇转变为麦角甾醇-5, 7, 22, 24(28)-四烯醇, 然后ERG4p (C24=28亚甲基还原酶)将麦角甾醇-5, 7, 22, 24(28)-四烯醇去饱和形成麦角甾醇(林晓珊等, 2010)。在酿酒酵母(Saccharomyces cerevisiae)中过表达ERG5导致麦角甾醇生物合成能力降低(蔡鹏丽等, 2007)。C-5甾醇去饱和酶由ERG3基因编码, 该酶的作用是在表甾醇B环的C-5和C-6位置各引入一个双键(Nakamura et al, 2004)。另外, ERG3基因的表达同时也影响其他脂质的代谢, 如敲除ERG3基因的酵母大量合成甘油并分泌至胞外, 导致细胞耐盐性降低(王慧等, 2020)。
脂肪酸去饱和酶(FAD)是不饱和脂肪酸合成的关键酶, 决定脂肪酸的不饱和程度。FAD种类较多, 广泛存在于动植物、藻类及真菌中。FAD在酿酒酵母中主要催化合成单不饱和脂肪酸。Δ9FAD是不饱和脂肪酸合成过程中的第一个酶, 在酿酒酵母中该酶能将双键引入饱和脂肪酸C16:0/C18:0去饱和产生单不饱和脂肪酸C16:1/C18:1 (Stukey et al, 1990; Nagao et al, 2019)。由于酿酒酵母的基因组注释完整且遗传操作简单, 因此适合作为研究真核生物代谢遗传学及生化机制的模式微生物(杨志刚等, 2014)。有学者将螺旋蓝藻(Spirulina sp.) Δ6FAD基因在酿酒酵母中进行功能表达, 成功获得了亚麻酸(Kurdrid et al, 2005)。Ma等人(2011)将海洋微藻(Nannochloropsis oculata)中NANOC-D6D基因克隆入酿酒酵母成功表达, 该基因与酿酒酵母Δ6FAD基因具有相似的去饱和酶活性。最近, 在光滑球拟酵母(Candida glabrata) med15Δ菌株中过表达脂肪酸延伸酶基因elo1和Δ9FAD去饱和酶基因olb1能够通过提高不饱和脂肪酸的含量增强细胞对低pH胁迫的耐受能力(齐艳利等, 2021)。
本研究分别以SD (ERG5)和FAD (OLE1)基因敲除酿酒酵母菌株YMR015C和YGL055W作为实验菌株。PCR扩增EhV99B1基因组中的EhV-SD和EhV-FAD基因, 构建酿酒酵母重组表达载体pYES2/CT-SD和pYES2/CT-FAD, 分别转化相应基因缺陷型酵母细胞并诱导过表达, 然后采用UPLC-Q-Exactive-MS非靶向脂质组学技术比较分析重组酵母和缺陷型酵母细胞脂质组成和含量的变化, 初步确定病毒EhV99B1基因组编码的上述两个鞘脂代谢酶可能的功能。为深入了解脂代谢在颗石藻病毒与宿主互作过程中的调节作用提供新的理论认识, 也为进一步开发合成新型鞘脂的酵母细胞奠定基础。
1 材料与方法 1.1 实验材料颗石藻病毒EhV-99B1株系分离自挪威海域(Schroeder et al, 2002), 由挪威卑尔根大学Gunnar bratbak教授赠送。野生型酿酒酵母Saccharomyces cerevisiae BY4741及SD (ERG5)、FAD (OLE1)基因缺失型酵母菌株YMR015C和YGL055W均购自EUROSCARF菌种库(德国法兰克福大学微生物研究所); 大肠杆菌Top10购自泰京生物科技有限公司(中国, 厦门); 克隆载体pMD19-T购自TaKaRa公司(日本), 酿酒酵母表达载体pYES2/CT购自Invitrogen公司(美国)。
1.2 仪器与试剂四级杆轨道阱杂交傅里叶变换超高分辨质谱仪(美国Thermofisher, Q-Exactive), 超高液相色谱仪(美国Waters, ACQUITY UPLC), 高速冷冻离心机(美国BECKMAN COULTER, Avanti J-26S XP), 全自动样品快速研磨仪(上海净信, Tissuelyser-24)。戊二醛、甲醇、氯仿购自国药集团化学试剂有限公司, 均为色谱级纯。S.c.EasyCompTM酿酒酵母转化试剂盒购自Invitrogen公司(美国)。
1.3 酵母重组表达载体pYES2/CT-SD和pYES2/ CT-FAD的构建及转化以实验室保存的含病毒EhV-SD和EhV-FAD基因的重组质粒pMD19-T-SD和pMD19-T-FAD作为模板, PCR扩增目的基因, 扩增产物送至上海生工测序验证, EhV-SD和EhV-FAD基因GenBank登录号分别为CAZ369370和YP_293815。分别用限制性内切酶Xba I/Xho I和Xba I/Pst I对载体pMD19-T-SD、pMD19-T-FAD以及酿酒酵母表达载体pYES2/CT进行双酶切, 构建酵母重组表达载体pYES2/CT-SD和pYES2/CT-FAD, 分别转化大肠杆菌Top10感受态细胞, 筛选阳性克隆并进行质粒PCR和双酶切验证。采用酿酒酵母转化试剂盒(S.c.EasyCompTM)将重组表达载体pYES2/CT-SD和pYES2/CT-FAD分别转化酿酒酵母相应基因缺陷型菌株YMR015C和YGL055W感受态细胞, 空载pYES2/CT作为对照分别转化上述两株基因缺陷型菌株, 具体操作步骤参考试剂盒说明书。转化产物涂布于15 mL的SC选择培养基(含有50 μg/mL卡那抗生素、20 g/L葡萄糖)中, 30 ℃过夜培养, 挑取单菌落进行菌落PCR验证, 确定阳性子。
1.4 重组质粒在酿酒酵母细胞中的诱导表达将两株对照菌株及阳性转化菌株分别接种到含有2% D-半乳糖的SC-U培养基中, 30 ℃诱导培养24 h, 野生型酿酒酵母菌株BY4741在YDP培养基中培养24 h。分别离心收集20 mL细胞沉淀, 液氮速冻后保存于−80 ℃用于总脂和RNA的提取。每组样品设置4~5个生物学重复。
1.5 细胞总脂的提取向上述5组样品中分别加入500 μL甲醇溶液和8个小钢珠, 添加适量液氮, 在全自动样品快速研磨仪上快速研磨(65 Hz, 6次/3 min), 然后加入1 mL氯仿和417 μL Milli-Q水并充分混匀, 常温静止5 min待其分层, 取每管下层有机相250 μL作为粗脂样品。每管样品剩余的下相混合后取250 μL作为质控样本(quality control, QC), 将粗脂样品与QC同时离心浓缩, 置于−80 ℃保存备用。
1.6 UPLC-Q-Exactive-MS检测分析 1.6.1 液相条件本实验中所用的仪器为超高效液相色谱仪与四级杆和轨道阱杂交傅里叶变换超高分辨质谱仪串联系统(UPLC-Q-Exactive-MS)。色谱柱为AQUITY UPLC BEH C8 (2.5 mm×100 mm, 1.7 μm, 沃特世)。二元溶剂系统流动相A为己烷和异丙醇︰乙腈(IPA︰ACN)=90︰10 (含10 mmol/L的醋酸铵), 流动相B为CAN︰H2O=60︰40 (含10 mmol/L的醋酸铵), 洗脱梯度为: 初始比例为32%的流动相B持续1.5 min, 然后在14 min内线性增加到85%, 接着在0.1 min内从85%增加到97%, 并保持2.4 min, 最后在0.1 min内重新回到初始流动相比例并保持1.9 min, 流速为0.26 mL/min。
1.6.2 质谱条件电喷雾电离源(ESI), 正负离子检测, 鞘气流速(Sheath Gas Flow Rate)为55 arb, 辅助气流速(Aux Gas Flow Rate)为12, 喷雾电压(Spray Voltage)3 kV, 毛细管电压3.0 kV, 干燥气体(Desolvation Temperature)温度为265 ℃, 毛细管电压(Capillary Voltage)2.5 kV, 质量扫描范围为100~1 500质荷比, 采取针泵自动进样, 每次进样量为6 μL, 进样室温为10 ℃。
1.6.3 质谱检测数据分析采用面积归一化半定量分析方法, 根据待测组分峰面积与所有峰面积和的百分比对粗脂进行半定量分析。以准确的质荷比、串联质谱法碎裂模式和保留行为为基础, 基于LIPID MAPS数据库, 用LipidSearch和Xcalibur (Thermo, 美国)软件鉴定脂质种类。UPLC-Q-Exactive-MS获得的原始数据, 利用MS-DIAL v2.94进行数据预处理, 再利用每个样本的总离子流进行归一化处理。在统计分析之前, 将所有样品的峰面积标准化为细胞沉淀的干重, 然后每个脂类样品峰面积分别和总峰面积相比进行半定量分析。将标准化数据集导入SIMCA-P软件(瑞典Umetrics)进行单变量和多元变量统计分析, 用MeV构建热图以显现所有实验组中脂质和脂质类别的相对水平。采用SPSS 17.0进行独立样本t检验, 以变量在项目中的重要性(VIP值> 1), |FC| > 2以及t检验P < 0.001确定差异代谢物。
1.7 qRT-PCR检测EhV-SD和EhV-FAD基因的表达使用RNA提取试剂盒(RNA simple total RNA Kit, DP419, 天根)提取细胞总RNA。使用Hiscript逆转录试剂盒(Hiscript II Q RTSuperMix, 诺唯赞)将RNA反转录成cDNA, 反应程序为: 55 ℃, 15 min; 85 ℃, 5 s。利用Primer Premier 5.0设计内参基因(β-actin)和两种目的基因引物, 引物序列如表 1。qRT-PCR反应体系为10 μL, 其中包括1 μL cDNA, 5 μL 1×SYBR Green Premix预混液(Vazyme, 中国), 3.6 μL dd H2O和0.2 μL F/R引物, 每个样品三个生物学重复, qRT-PCR使用Applied Biosystems 7300 Real-Time PCR系统。qRT-PCR反应参数为: 第一阶段: 95 ℃ 30 s进行初始变性; 第二阶段: 95 ℃ 30 s, 60 ℃ 30 s, 50个循环; 第三阶段: 95 ℃ 15 s, 60 ℃ 60 s, 99 ℃ 15 s, 60 ℃ 15 s。
| 引物 | 引物序列(5′~3′) |
| Scβ-actin-F | ATGCAAAAGGAAATCACCGC |
| Scβ-actin -R | CAAGATAGAACCACCAATCCAGAC |
| EhV-SD-F | TCACCGAGCACTTCATCATCCTA |
| EhV-SD-R | GCATGAGCTGCGTATGGAAATAG |
| EhV-FAD-F | CGTCCCATCATCTTGAACTTACTC |
| EhV-FAD-R | AAACATTGGTCGGAGAAGATAGG |
采用SPSS 22.0进行统计学分析, 实验数据以平均值±标准差(mean ± SD)表示, 显著性差异采用t检验。*表示P < 0.05, 差异显著; **表示P < 0.01; ***表示P < 0.001, 差异极显著。利用Origin 2019b和Adobe illustrator软件对数据进行做图分析。
2 结果 2.1 酵母重组质粒的构建、转化及诱导表达重组表达载体pYES2/CT-SD和pYES2/CT-FAD的质粒PCR和双酶切结果见图 1, EhV-SD和EhV-FAD基因片段分别为987 bp (图 1a)和963 bp (图 1b), 符合预期大小, 重组表达载体构建成功。qRT-PCR检测结果显示, EhV-SD和EhV-FAD基因分别在相应的重组酵母细胞中发生了不同程度的表达, 含空载质粒的酵母细胞均未扩增出产物。
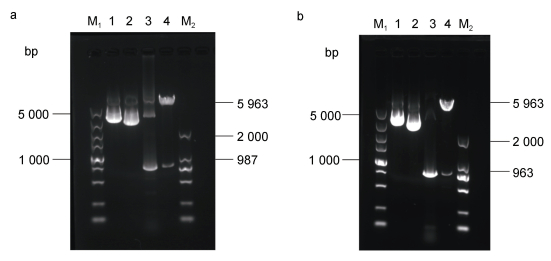 |
| 图 1 重组表达载体pYES2/CT-SD和pYES2/CT-FAD的质粒PCR及双酶切鉴定结果 Fig. 1 Agarose gel electrophoresis of plasmid PCR and double enzyme digestion of recombinant vector YES2/CT-SD and pYES2/CT-FAD 注: a. 重组表达载体pYES2/CT-SD的质粒PCR和双酶切电泳结果图; b. 重组表达载体pYES2/CT-FAD的质粒PCR和双酶切电泳结果图; M1: 5 000 bp DNA Ladder Marker; M2: 2 000 bp DNA Ladder Marker; 泳道1: 重组表达载体pYES2/CT-SD(a)和pYES2/CT-FAD(b); 泳道2: 表达载体pYES2/CT(a-b); 泳道3: 重组表达载体pYES2/CT-SD(a)和pYES2/CT-FAD(b)的质粒PCR结果; 泳道4: 重组表达载体pYES2/CT-SD(a)和pYES2/CT-FAD(b)的双酶切结果 |
在上述色谱与质谱条件下, 空载和重组酵母细胞的总脂提取物在反向色谱柱上得到了良好的分离, 选取正离子模式下四种酿酒酵母的总离子流(TIC)为代表展示不同酿酒酵母细胞中总脂的分离情况(图 2)。依据脂质精确质量数、保留时间、二级质谱信息及LIPID MAPS数据库等信息, 在正、负离子模式下, 空载和EhV-SD重组酵母细胞中共鉴定出27种脂质代谢物, 其中正离子模式下19种271个, 负离子模式下20种156个, 正负离子模式下同时鉴定出的为溶血磷脂酰胆碱(LPC)、溶血磷脂酰乙醇胺(LPE)、磷脂酰乙醇胺(PE)和磷脂酰胆碱(PC)等12种脂质。空载和EhV-FAD重组酵母细胞中共鉴定出29种脂质代谢物, 其中正离子模式下19种203个, 负离子模式下22种150个, 正负离子模式下同时鉴定出的为LPC、磷脂酰甘油(PG)、PE和PC等12种。
 |
| 图 2 正离子模式下酵母细胞总脂的总离子流图 Fig. 2 Total ion chromatogram of lipid in S. cerevisiae in ESI+ mode 注: a. 空载酵母YMR015C细胞总脂的离子流图; b. EhV-SD重组酵母YMR015C细胞总脂的离子流图; c. 空载酵母YGL055W细胞总脂的离子流图; d. EhV-FAD重组酵母YGL055W细胞总脂的离子流图 |
正负离子模式下五组酵母细胞脂质数据的主成分分析(PCA)得分图中, 各组样品在PCA得分图的第一、第二主成分上被清晰地区分开(图 3)。经建模分析, 该模型正离子模式下对X矩阵的解释率(R2X)为70.2% (图 3a), 负离模式下R2X为81.4% (图 3b), 表明脂质组学分析的稳定性和重复性较好。
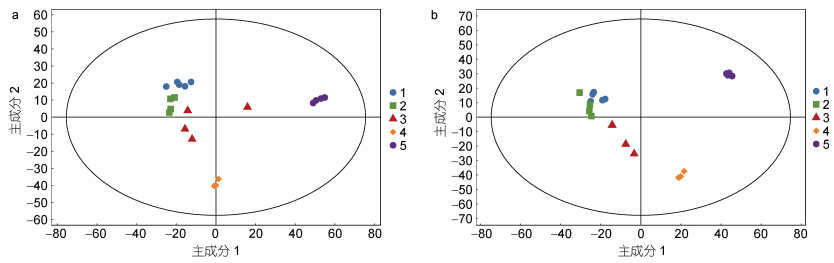 |
| 图 3 正负离子模式下五组酵母细胞总脂的主成分分析图 Fig. 3 Principal component analysis score scatter plot of total lipids in five S. cerevisiae cells in ESI+ and ESI− mode, respectively 注: a. 正离子模式下五组酵母细胞总脂的主成分分析图; b. 负离子模式下五组酵母细胞总脂的主成分分析图; 1. 蓝色圆圈代表空载酵母YMR015C; 2. 绿色方形代表EhV-SD重组酵母YMR015C; 3. 红色三角代表空载酵母YGL055W; 4. 橙色菱形代表野生型酵母BY4741; 5. 紫色圆圈代表EhV-FAD重组酵母YGL055W |
通过有监督的正交偏最小二乘判别分析OPLS-DA模型, 空载和EhV-SD重组酵母细胞脂质在图中能较好地分开(图 4)。正负离子模式下EhV-SD重组酵母细胞和空载酵母细胞样品之间, 该模型对Y矩阵的解释率(RY2)和模型的预测率(Q2)均高于60%, 模型对Y矩阵的预测率(QY2)值大于60%, 且RY2与QY2之间的差值小于0.3 (图 4a, 4c), 表明模型的预测能力良好。通过交叉排列验证对模型有效性进一步验证, 经过200次置换验证的结果显示(图 4b, 4d), R2与Q2接近, RY2的斜率大于0且QY2 < 0.05, 说明模型没有明显的过拟合现象, 能较好地反映空载和EhV-SD重组酵母样品脂类代谢物之间的差异。
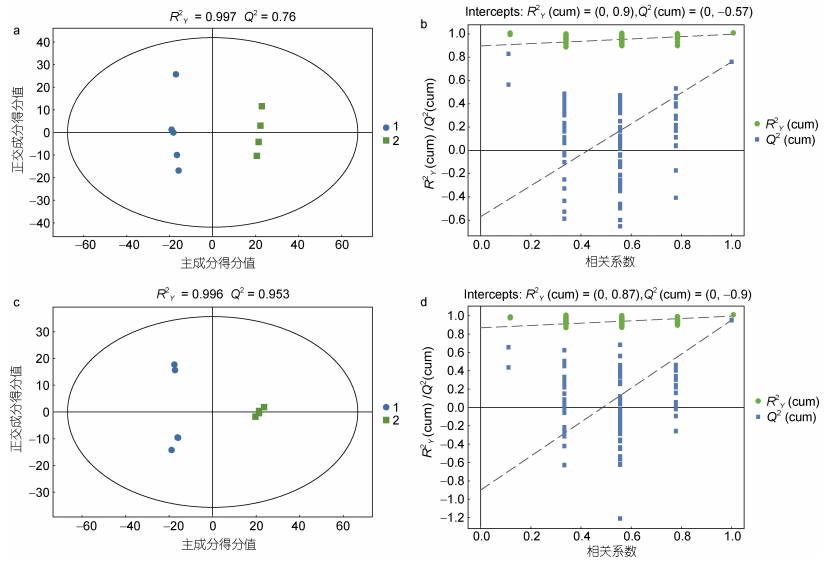 |
| 图 4 EhV-SD重组酵母细胞脂质OPLS-DA和交叉验证图 Fig. 4 OPLS-DA and cross-validation plots of lipid in EhV-SD recombinant S. cerevisiae 注: a. 正离子模式下OPLS-DA图; b. 正离子模式下交叉验证图; c. 负离子模式下OPLS-DA图; d. 负离子模式下交叉验证图; 1. 蓝色圆圈代表空载酵母YMR015C; 2. 绿色方形代表EhV-SD重组酵母YMR015C; OPSL-DA: 正交偏最小二乘判别分析; R2: 常规相关系数; Q2: 交叉验证系数; Correlation Coefficien: 样本得分值和变量间相关系数 |
EhV-FAD重组酿酒酵母细胞脂质OPLS-DA图和交叉验证图结果如图 5所示。在正、负离子模式下EhV-DAD重组酵母细胞和空载酵母样品RY2与QY2之间的差值小于0.3, 且QY2值大于60 %, 表明模型的预测能力良好(图 5a, 5c)。通过交叉排列验证对模型有效性进一步验证, 经过200次置换验证(图 5b, 5d), R2和Q2相交, RY2的斜率大于0且QY2 < 0.05, 说明所建立的模型没有过拟合现象。综上, 四组不同酿酒酵母在正、负离子检测模式下获得的UHPLC- (+)ESIMS数据矩阵所构建数据模型具有显著统计学意义。
 |
| 图 5 EhV-FAD重组酵母脂质OPLS-DA和交叉验证图 Fig. 5 OPLS-DA and cross-validation plots of lipid in EhV-FAD recombinant S. cerevisiae 注: a. 正离子模式下OPLS-DA图; b. 正离子模式下交叉验证图; c. 负离子模式下OPLS-DA图; d. 负离子模式下交叉验证图; 3. 红色三角代表空载酵母YGL055W; 5. 紫色圆圈代表EhV-FAD重组酵母YGL055W; OPSL-DA: 正交偏最小二乘判别分析; R2: 常规相关系数; Q2: 交叉验证系数; 相关系数: 样本得分值和变量间相关系数 |
利用火山图对不同酵母细胞中差异脂质代谢物的分布及统计学差异进行分析, 筛选条件为log2(FC) > 1、P < 0.05。进一步利用OPLS-DA模型中的变量权重值(variable importance for the projection, VIP)挖掘具有生物学意义的差异脂质分子, 根据VIP>1、|log2(FC)|>1和P<0.001的条件对脂质进行交叉筛选, 综合以上多变量与单变量分析, 在正、负离子模式下, 与空载酵母相比, EhV-SD重组酵母中共鉴定出20种差异脂质代谢物, 主要包括脂肪酸(FA)、神经酰胺(Cer)、甘油三酯(TAG)、1, 2二棕榈酰甘油三甲基高丝氨酸(DGTS)、甘油二酯(DAG)及磷脂类(PE、磷脂酰丝氨酸PS、LPE)等, 其中正离子模式下10种34个, 负离子模式下10种30个。与空载酵母相比, EhV-FAD重组酵母中共鉴定出22种差异脂质代谢物, 主要是甘油酯(TAG、DAG)、磷脂(PC、PG、PE)、脂肪酸(FA、羟基脂肪酸支链脂肪酸(FAHFA)和神经酰胺类(Cer), 其中正离子模式下15种64个, 负离子模式下17种114个。
将总峰面积法矫正后获得的各种脂类的含量进行具体分析, 运用MeV4.9.0软件对鉴定出的差异脂质代谢物绘制热图(图 6)。与空载酵母相比, EhV-SD重组酵母细胞中的差异脂质代谢物根据Pearson相关系数被聚类为3种明显的变化趋势(图 6a), 丰度相对较低的脂类主要集中在Ⅰ组, 包括神经酰胺类的二羟鞘氨醇(Cer-NDS)和无羟基神经酰胺(Cer-NS), 磷脂PC、PG、PS及LPE, 脂肪酸FAHFA和FA及葡萄糖醛酸糖脂(Glc-ADG)。α-羟基鞘氨醇神经酰胺(Cer-AP), 磷脂LPE、LPG和溶血磷脂酰肌醇(LPI), 甘油酯DAG、DGTS和TAG的丰度则相对较高, 主要集中在Ⅱ、Ⅲ组。与空载酵母相比, EhV-FAD重组酵母中差异脂质代谢物丰度变化趋势也聚为3类(图 6b), 其中磷脂PE、PI、LPC、PC及LPA, 单半乳糖二酰甘油(MGDG)、TAG及DAG和神经酰胺Cer-NS、Cer-AP、二羟鞘氨醇神经酰胺(Cer-ADS)及鞘氨醇神经酰胺(Cer-AS的丰度显著下降, 主要集中在第Ⅰ组; 而磷脂PG、PS和SM, 甘油酯Glc-ADG、DGTS、硫代异鼠李糖甘油二酯(SQDG)和溶血酰基甘油三甲基高丝氨酸(LDGTS)及脂肪酸FA和FAHFA的丰度显著增加, 主要集中在第II组。
 |
| 图 6 酵母细胞中显著差异脂质代谢物聚类热图 Fig. 6 The heatmap of significant differential lipids in S. cerevisiae 注: a. EhV-SD重组酵母细胞(YMR015C-SD)与空载酿酒酵母细胞(YMR015C)显著差异脂质丰度热图; b. EhV-FAD重组酵母细胞(YGL055W-FAD)与空载酿酒酵母细胞(YGL055W)显著差异脂质丰度热图; Cer-NDS: 二氢鞘氨醇; FAHFA: 羟基脂肪酸支链脂肪酸酯; PC: 磷脂酰胆碱; Cer-NS: 无羟基神经酰胺; P: G磷脂酰甘油; PS: 磷脂酰丝氨酸; PE: 磷脂酰乙醇胺; PI: 磷脂酰肌醇; FA: 脂肪酸; LPE: 溶血磷脂酰乙醇胺; LPC: 溶血磷脂酰胆碱; LPA: 溶血磷脂酸; GlcADG: 葡萄糖醛酸糖脂; Cer-AS: 鞘氨醇神经酰胺; Acar: 酰基肉碱; Cer-ADS: 二羟鞘氨醇神经酰胺; Cer-AP: α-羟基鞘氨醇神经酰胺; DAG: 甘油二酯; LPG: 溶血磷脂酰甘油; LPI: 溶血磷脂酰肌醇; DGTS1, 2: 二棕榈酰甘油三甲基高丝氨酸; MGD: G单半乳糖二酰甘油; SQDG: 硫代异鼠李糖甘油二酯; TAG: 甘油三酯; SM: 神经鞘磷脂; LDGTS: 溶血酰基甘油三甲基高丝氨酸 |
从空载和重组酵母细胞中选取具有代表性差异脂质代谢物进行统计学分析, 结果如图 7所示。与空载酵母相比, EhV-SD重组酵母细胞中含多不饱和酰基链的甘油三酯(TAG, 12︰3)和磷脂酰胆碱(PC, 20︰4/20︰5/20︰6)种类的含量显著升高(图 7a, 7b), 而含多不饱和酰基链的磷脂酰甘油(PG, 16︰2/18︰4)和部分多不饱和脂肪酸(FA, 16︰2/16︰4)的丰度则显著降低(图 7c, 7d)。EhV-FAD重组酵母细胞中, 不饱和脂肪酸特别是单不饱和脂肪酸, 如棕榈油酸(16︰1)和油酸(18︰1)的合成显著积累, 而饱和脂肪酸的含量则显著降低(图 7e~7g), 同时含不饱和酰基链的鞘磷脂(SM, 14︰1/14︰3/16︰3)种类也显著增加(图 7h)。
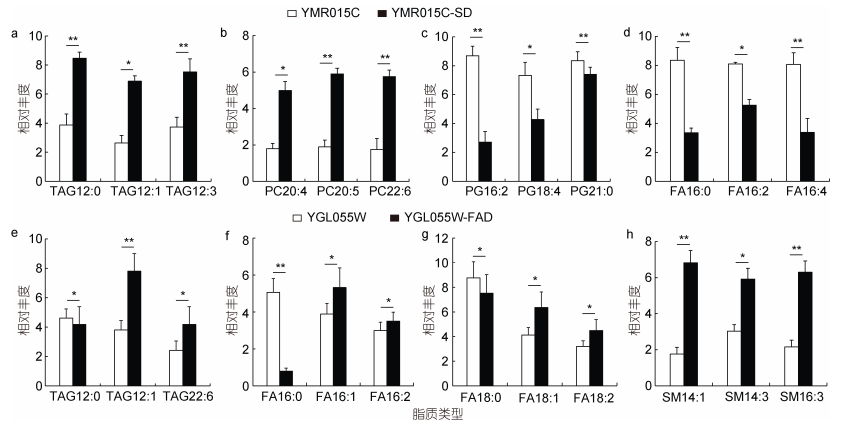 |
| 图 7 EhV-SD和EhV-FAD基因过表达对酵母细胞中几种典型脂质丰度的影响 Fig. 7 Effect of genes EhV-SD and EhV-FAD overexpression on several typical lipids of S. cerevisiae 注: 相对丰度指与野生型酵母相比, 空载酵母和重组酵母中脂质的相对丰度; *: P < 0.05; **: P < 0.01 |
在正、负离子模式下, 从EhV-SD重组酵母细胞中筛选出20种差异脂质代谢物, 从EhV-FAD重组酵母细胞中筛选出22种差异脂质代谢物, 基于KEGG数据库构建相关代谢通路图(图 8)。与酵母细胞C22-SD类似, EhV-SD调控酵母细胞麦角甾醇生物合成的最后一步反应, 可能是由于本研究采用的是非靶向脂质组学技术, 限于总脂提取方法及检测方法的原因, 本实验条件下没有检测到该酶直接催化形成的甾醇类化合物。但我们发现EhV-SD除了调控甾醇化合物的合成外, 还可能通过其它中间代谢过程改变酵母细胞中多种脂质的水平, 如鞘脂和甘油酯含量显著增加, 而脂肪酸和部分磷酯则显著降低(图 8a)。EhV-FAD基因的过表达直接导致了单不饱和脂肪酸, 如油酸(18︰1)和棕榈油酸(16︰1)的积累。另外, 该基因的过表达还可能通过其他中间代谢过程在不同程度上间接抑制部分脂肪酸、甘油酯、鞘脂及磷脂等的代谢过程(图 8b)。
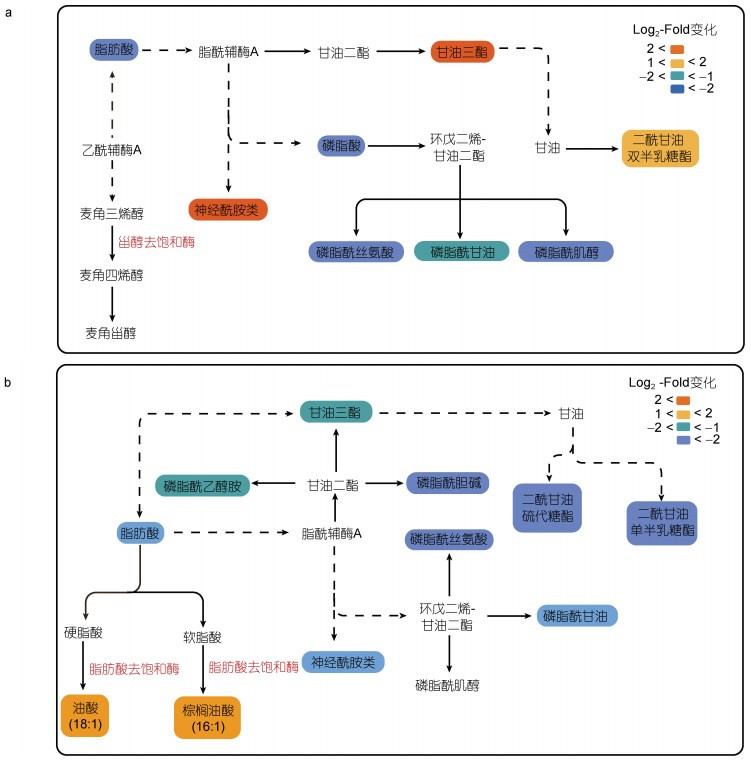 |
| 图 8 EhV-SD和EhV-FDA介导的酿酒酵母细胞脂质代谢通路图 Fig. 8 EhV-SD and EhV-FAD mediated lipid metabolic pathways in S. cerevisiae 注: a. EhV-SD重组酿酒酵母细胞脂质代谢通路图; b. EhV-FAD重组酿酒酵母细胞脂质代谢通路图; Log2-Fold change代表脂质含量的变化倍数: 蓝色系列代表脂质含量减少; 红色系列表示脂质含量增加 |
本研究通过在SD和FAD基因缺陷型酿酒酵母中过表达颗石藻病毒EhV99B1甾醇去饱和酶基因EhV-SD和脂肪酸去饱和酶基因EhV-FAD, 初步明确了病毒EhV-SD和EhV-FAD基因的基本功能。酵母中甾醇的生物合成是一个多酶催化的复杂过程, 且其生物合成的调控是多层次的。在酿酒酵母中, ERG5基因编码的C-22去饱酶催化酵母麦角甾醇生物合成途径中的第20步反应, 在甾醇侧链的C-22位引入反式双键, 从而形成四烯甾醇。ERG3编码的酵母C-5去饱和酶催化表甾醇B环的C-5和C-6位双键的引入(Taton et al, 2000)。如ERG3基因缺失导致酵母细胞不能合成麦角固醇, 而合成了5, 6-二氢麦角固醇(王慧等, 2020)。甾醇C-24还原酶基因(ERG4)的过表达导致酵母细胞中麦角甾醇含量提高, 而C-22去饱和酶基因(ERG5)的过表达则导致酵母细胞中麦角甾醇含量降低(蔡鹏丽等, 2007)。可见, 就酵母本身而言, 其SD基因的功能十分复杂, 这些酶在麦角甾醇生物合成中的调控作用还不十分清楚。本研究将EhV-SD基因克隆入ERG5基因缺陷型酵母菌株, EhV-SD基因成功表达并改变了酵母多种脂质的含量, 但在EhV-SD重组酵母及野生型酵母细胞中均未检测到甾醇类化合物, 这可能由于本实验采用的细胞总脂提取及非靶向脂质检测条件等原因所致。不过, 本研究结果发现EhV-SD基因的过表达可能通过其他中间代谢过程改变酵母细胞中多种脂质的水平, 如TAG和Cer水平显著升高, 特别是TAG中含单不饱和及多不饱和酰基链的种类均显著增加(图 7a, 7b), 表明EhV-SD具有明显的去饱和酶作用。另外, 在酿酒酵母细胞中过表达ERG5基因导致酵母细胞内极性脂含量降低(蔡鹏丽等, 2007), 而对酵母ERG5基因实施突变后, 不仅改变甾醇的含量还间接影响鞘脂的代谢(Guan et al, 2009)。可见, 在酵母细胞中, 甾醇C-22去饱和酶酶促反应下游除了已有的合成代谢途径外, 可能还存在其他的分支代谢途径, 通过这些途径间接调控细胞中其它脂质的代谢。除酵母外, 高等植物中也存在类似现象, 如在番茄中表达食用金针菇的C-5-SD基因能够促进番茄中多不饱和脂肪酸的合成(Kamthan et al, 2012)。最新一篇对拟南芥的研究发现, 甾醇能够通过影响脂滴表面的物理特性干扰TAG合成酶的作用, 从而影响拟南芥种子中TAG的积累(Yu et al, 2021)。以上结果表明, SD酶的功能复杂多样, 且不同物种SD功能之间的差异也较大, 这可能是重组EhV-SD酵母细胞中脂质发生多样性变化的一个原因。在EhV感染的颗石藻细胞中, 包括甾醇在内的几种萜类化合物含量均显著降低, 甾醇作为病毒包膜的组成部分, 宿主感染下调甾醇等脂类含量可能是宿主的一种抗病毒防御策略(Rosenwasser et al, 2014)。因此我们推测, EhV-SD也可能在一定程度上能够抑制酵母细胞甾醇类化合物的合成。此外, EhV感染能够诱导宿主藻细胞中性脂的积累并形成大量脂滴以利于病毒的复制和组装(Malitsky et al, 2016; Zeng et al, 2019)。以上结果提示, 在EhV感染的藻细胞中, EhV-SD基因的表达可能改变了宿主甾醇的生物合成, 而甾醇作为信号分子调节脂类相关基因的转录从而促进TAG的合成和积累, 为病毒的复制和组装提供条件。
脂肪酸去饱和酶(FAD)属于酰基-CoA去饱和酶, FAD能在饱和脂肪酸链中引入双键生成不饱和脂肪酸, 降低细胞膜中饱和脂肪酸含量, 增加不饱和脂肪酸含量(舒海燕等, 2021)。Δ9FAD基因编码的FAD是酵母不饱和脂肪酸生物合成过程中关键的限速酶, 可以将第一个双键分别引入软脂酸(16︰0)和硬脂酸(18︰0), 生成棕榈油酸(16︰1Δ9)和油酸(18︰1Δ9) (Jeffcoat et al, 1977)。EhV-FAD的过表达显著增加了重组酵母中不饱和脂肪酸的生物合成, 特别是促进棕榈油酸(16︰1)和油酸(18︰1)的积累, 而饱和脂肪酸含量则显著降低(图 7f~7g), 表明EhV-FAD基因与酿酒酵母细胞Δ9FAD基因具有类似的生物学功能。EhV 86感染颗石藻E. huxleyi CCMP1516诱导脂肪酸由多不饱和向单不饱和漂移, 特别是显著促进C18︰1(n-9)(Evans et al, 2009; Zeng et al, 2019)和C8-C16不饱和脂肪酸的显著积累(Rosenwasser et al, 2014)。可见, 病毒介导的颗石藻脂肪酸代谢可能是EhV- FAD基因和宿主相关同源基因共同作用的结果。另外, 在哺乳动物细胞中发现, D5D和D6D脂肪酸去饱和酶催化产生的不饱和脂肪酸可作为转录因子(如胆固醇调控原件结合蛋白1和过氧化物酶体增殖激活物受体)的配体, 参与调控与脂肪合成和脂肪酸氧化相关酶基因的表达, 从而影响甘油酯和脂肪酸的组成(Warensjö et al, 2009)。类似地, 本研究结果显示, EhV-FAD的过表达不仅显著影响酵母脂肪酸代谢, 同时也影响其他脂质的特性, 如抑制甘油三酯、磷脂及鞘脂类的丰度, 而显著增加了它们的不饱和程度(图 7a~7g)。在SPT基因缺陷型酿酒酵母中表达EhV-SPT, 发现病毒SPT利用鞘脂的从头合成途径并偏好以C15-CoA(肉豆蔻酰辅酶A)而非常规的C16-CoA (棕榈酰辅酶A)为底物, 合成基于奇数C17-LCB长链的病毒性鞘脂(Han et al, 2006)。同时, 在EhV感染宿主过程中, 病毒通过启动EhV-SPT基因的表达, 抑制宿主鞘脂的合成, 转而大量合成病毒特有的奇数碳链、富含羟基和不和键的新型鞘糖脂(Ziv et al, 2016)。基于以上结果, 我们推测在病毒感染重构宿主鞘脂代谢过程中, EhV-SPT通过其底物偏好性启动病毒性鞘脂的合成, 而EhV-FAD则催化病毒性鞘糖脂结构中不饱和脂肪酸链的形成。另外, 与EhV-SD重组酵母相反, EhV-FAD重组酵母中Cer含量显著减少(图 8b), 而其降解产物鞘氨醇(SM)含量显著升高(图 7h)。众所周知, Cer作为一种细胞信号分子, 参与调控高等动植物众多细胞代谢, 并作为典型的信号分子诱导细胞凋亡和程序性死亡。在病毒感染的颗石藻细胞中, EhV-FAD的表达间接促进毒性Cer向SM的转变, 这可能也是病毒抑制宿主过早诱导细胞死亡的一种策略。综上, 本研究结果提示, 在病毒感染的颗石藻细胞中, EhV-SD和EhV-FAD的协同作用调控宿主细胞中鞘脂类物质的水平, 从而决定细胞命运以调控颗石藻赤潮消亡过程。
4 结论本研究结合酵母突变体功能互补和非靶向脂质组学方法, 初步确定了赫氏颗石藻病毒基因组中甾醇去饱和酶基因(EhV-SD)和脂肪酸去饱和酶基因(EhV-FAD)的功能。EhV-SD主要功能是催化甾醇脂类的生物合成, 产物甾醇可能作为信号分子间接调控了酵母细胞中磷脂类、鞘脂类及甘油酯类和脂肪酸的含量, 从而影响酵母细胞酯类代谢; EhV-FAD则主要催化酵母细胞中脂肪酸代谢, 特别是促进单不饱和脂肪酸油酸和棕榈油酸的积累, 同时也影响酵母细胞中鞘脂类、磷脂类及甘油酯类的代谢。EhV-SD和EhV-FAD作为颗石藻病毒基因组编码的关键辅助代谢基因, 在感染过程中与其他相关酶基因协同作用, 驱动宿主鞘脂代谢流向病毒的转移以合成病毒性鞘脂, 并最终诱导宿主细胞凋亡。可见, 自然海域中颗石藻病毒-宿主在鞘脂合成代谢中存在激烈的博弈过程。然而, 包括颗石藻在内的光合作用单细胞微生物的基因组中缺乏编码如Bcl-2、p53和caspases等哺乳动物典型的细胞凋亡因子, 但却能以细胞凋亡方式应答多种生物和非生物胁迫, 其确切调控机制尚不十分清楚。针对颗石藻病毒宿主这一模式体系, 开展鞘脂代谢相关辅助基因功能以及鞘脂介导的细胞死亡分子机制的研究, 将有助于深入认识单细胞生物程序性细胞死亡途径及其生态学意义。
王慧, 陈雄, 林卫潮, 等, 2020. C-5固醇去饱和酶ERG3对酿酒酵母耐盐性的影响. 食品科学, 41(8): 83-90 |
齐艳利, 刘晖, 周配, 等, 2021. 过表达基因elo1和ole1增强光滑球拟酵母Δmed15B菌株的低pH耐受能力. 微生物学报, 61(5): 1359-1369 |
苏金净, 蔡伟聪, 李桂玲, 等, 2019. 浮游植物metacaspase的分布、结构及其功能特性. 海洋与湖沼, 50(5): 1059-1069 |
杨志刚, 郭子好, 姚琴琴, 等, 2014. 脂肪酸去饱和酶基因的研究进展. 生物技术通报, (12): 21-26 |
林晓珊, 江宏文, 张毅, 2010. 酵母麦角固醇生物合成及其基因调控的研究. 生物学杂志, 27(6): 83-86 DOI:10.3969/j.issn.1008-9632.2010.06.083 |
舒海燕, 冼淑云, 欧艳妃, 等, 2021. 通过转化脂肪酸去饱和酶基因AcoFAD2提高菠萝植株的抗寒能力. 分子植物育种, 19(21): 7132-7137 |
蔡鹏丽, 何秀萍, 刘楠, 等, 2007. 甾醇C-22去饱和酶高表达对酵母细胞麦角甾醇合成的影响. 微生物学报, 47(2): 274-279 |
BIDLE K D, HARAMATY L, RAMOS J B E, et al, 2007. Viral activation and recruitment of metacaspases in the unicellular coccolithophore, Emiliania huxleyi. Proceedings of the National Academy of Sciences of the United States of America, 104(14): 6049-6054 DOI:10.1073/pnas.0701240104 |
DANIELS C J, POULTON A J, BALCH W M, et al, 2018. A global compilation of coccolithophore calcification rates. Earth System Science Data Discussions, 10(4): 1-29 |
EVANS C, POND D W, WILSON W H, 2009. Changes in Emiliania huxleyi fatty acid profiles during infection with E. huxleyi virus 86: physiological and ecological implications. Aquatic Microbial Ecology, 55(3): 219-228 DOI:10.3354/ame01295 |
GUAN X L, SOUZA C M, PICHLER H, et al, 2009. Functional interactions between sphingolipids and sterols in biological membranes regulating cell physiology. Molecular Biology of the Cell, 20(7): 2083-2095 DOI:10.1091/mbc.e08-11-1126 |
HAN G S, GABLE K, YAN L Y, et al, 2006. Expression of a novel marine viral single-chain serine palmitoyltransferase and construction of yeast and mammalian single-chain chimera. Journal of Biological Chemistry, 281(52): 39935-39942 |
HUNTER J E, FRADA M J, FREDRICKS H F, et al, 2015. Targeted and untargeted lipidomics of Emiliania huxleyi viral infection and life cycle phases highlights molecular biomarkers of infection, susceptibility, and ploidy. Frontiers in Marine Science, 2: 81 |
JEFFCOAT R, BRAWN P R, SAFFORD R, et al, 1977. Properties of rat liver microsomal stearoyl-coenzyme a desaturase. Biochemical Journal, 161(2): 431-437 |
JOHNS C T, GRUBB A R, NISSIMOV J I, et al, 2019. The mutual interplay between calcification and coccolithovirus infection. Environmental Microbiology, 21(6): 1896-1915 |
KAMTHAN A, KAMTHAN M, AZAM M, et al, 2012. Expression of a fungal sterol desaturase improves tomato drought tolerance, pathogen resistance and nutritional quality. Scientific Reports, 2: 951 |
KURDRID P, SUBUDHI S, HONGSTHONG A, et al, 2005. Functional expression of Spirulina-Δ6 desaturase gene in yeast, Saccharomyces Cerevisiae. Molecular Biology Reports, 32(4): 215-226 |
LINDELL D, JAFFE J D, COLEMAN M L, et al, 2007. Genome- wide expression dynamics of a marine virus and host reveal features of co-evolution. Nature, 449(7158): 83-86 |
LIU J W, CAI W C, FANG X, et al, 2018. Virus-induced apoptosis and phosphorylation form of metacaspase in the marine coccolithophorid Emiliania huxleyi. Archives of Microbiology, 200(3): 413-422 |
MA X L, YU J Z, ZHU B H, et al, 2011. Cloning and characterization of a delta-6 desaturase encoding gene from Nannochloropsis oculate. Chinese Journal of Oceanology and Limnology, 29(2): 290-296 |
MALITSKY S, ZIV C, ROSENWASSER S, et al, 2016. Viral infection of the marine alga Emiliania huxleyi triggers lipidome remodeling and induces the production of highly saturated triacylglycerol. New Phytologist, 210(1): 88-96 |
NAGAO K, MURAKAMI A, UMEDA M, 2019. Structure and function of Δ9-fatty acid desaturase. Chemical and Pharmaceutical Bulletin, 67(4): 327-332 |
NAKAMURA M T, NARA T Y, 2004. Structure, function, and dietary regulation of Δ6, Δ5, and Δ9 desaturases. Annual Review of Nutrition, 24: 345-376 |
NISSIMOV J I, PAGARETE A, MA F R, et al, 2017. Coccolithoviruses: a review of cross-kingdom genomic thievery and metabolic thuggery. Viruses, 9(3): 52 |
ROSE S L, FULTON J M, BROWN C M, et al, 2014. Isolation and characterization of lipid rafts in Emiliania huxleyi: a role for membrane microdomains in host-virus interactions. Environmental Microbiology, 16(4): 1150-1166 |
ROSENWASSER S, MAUSZ M A, SCHATZ D, et al, 2014. Rewiring host lipid metabolism by large viruses determines the fate of Emiliania Huxleyi, a bloom-forming alga in the ocean. The Plant Cell, 26(6): 2689-2707 |
RUIZ E, OOSTERHOF M, SANDAA R A, et al, 2017. Emerging interaction patterns in the Emiliania huxleyi-EhV system. Viruses, 9(3): 61 |
SCHROEDER D C, OKE J, MALIN G, et al, 2002. Coccolithovirus (Phycodnaviridae): characterization of a new large dsDNA algal virus that infects Emiliana huxleyi. Archives of Virology, 147(9): 1685-1698 |
SHEYN U, ROSENWASSER S, BEN-DOR S, et al, 2016. Modulation of host ROS metabolism is essential for viral infection of a bloom-forming coccolithophore in the ocean. The ISME Journal, 10(7): 1742-1754 |
SHEYN U, ROSENWASSER S, LEHAHN Y, et al, 2018. Expression profiling of host and virus during a coccolithophore bloom provides insights into the role of viral infection in promoting carbon export. The ISME Journal, 12(3): 704-713 |
SKAGGS B A, ALEXANDER J F, PIERSON C A, et al, 1996. Cloning and characterization of the Saccharomyces cerevisiae C-22 sterol desaturase gene, encoding a second cytochrome P-450 involved in ergosterol biosynthesis. Gene, 169(1): 105-109 |
STUKEY J E, MCDONOUGH V M, MARTIN C E, 1990. The OLE1 gene of Saccharomyces cerevisiae encodes the delta 9 fatty acid desaturase and can be functionally replaced by the rat stearoyl-CoA desaturase gene. Journal of Biological Chemistry, 265(33): 20144-20149 |
TATON M, HUSSELSTEIN T, BENVENISTE P, et al, 2000. Role of highly conserved residues in the reaction catalyzed by recombinant Δ7-sterol-C5(6)-desaturase studied by site-directed mutagenesis. Biochemistry, 39(4): 701-711 |
VARDI A, HARAMATY L, VAN MOOY B A S, et al, 2012. Host-virus dynamics and subcellular controls of cell fate in a natural coccolithophore population. Proceedings of the National Academy of Sciences of the United States of America, 109(47): 19327-19332 |
VARDI A, VAN MOOY B A S, FREDRICKS H F, et al, 2009. Viral glycosphingolipids induce lytic infection and cell death in marine phytoplankton. Science, 326(5954): 861-865 |
WARENSJÖ E, ROSELL M, HELLENIUS M L, et al, 2009. Associations between estimated fatty acid desaturase activities in serum lipids and adipose tissue in humans: links to obesity and insulin resistance. Lipids in Health and Disease, 8: 37 |
WILSON W H, SCHROEDER D C, ALLEN M J, et al, 2005. Complete genome sequence and lytic phase transcription profile of a Coccolithovirus. Science, 309(5737): 1090-1092 |
YU L H, FAN J L, ZHOU C, et al, 2021. Sterols are required for the coordinated assembly of lipid droplets in developing seeds. Nature Communications, 12(1): 5598 |
ZENG J, LIU S S Y, CAI W C, et al, 2019. Emerging lipidome patterns associated with marine Emiliania huxleyi-virus model system. Science of the Total Environment, 688: 521-528 |
ZIV C, MALITSKY S, OTHMAN A, et al, 2016. Viral serine palmitoyltransferase induces metabolic switch in sphingolipid biosynthesis and is required for infection of a marine alga. Proceedings of the National Academy of Sciences of United States America, 113(13): E1907-E1916 |
 2023, Vol. 54
2023, Vol. 54


