中国海洋湖沼学会主办。
文章信息
- 李雯, 张明清, 赖星星, 张翠萍, 覃伟健, 杨金霖, 朱雅婷, 舒琥. 2022.
- LI Wen, ZHANG Ming-Qing, LAI Xing-Xing, ZHANG Cui-Ping, QIN Wei-Jian, YANG Jin-Lin, ZHU Ya-Ting, SHU Hu. 2022.
- 两种混养方式对异枝江蓠(Gracilaria bailinae)生长性能、表面附生细菌群落及抗生素抗性基因的影响
- EFFECTS OF TWO TYPES OF POLYCULTURE MODE ON GROWTH PERFORMANCE, SURFACE EPIPHYTIC BACTERIAL COMMUNITY AND ANTIBIOTIC RESISTANCE GENES OF GRACILARIA BAILINAE
- 海洋与湖沼, 53(1): 235-246
- Oceanologia et Limnologia Sinica, 53(1): 235-246.
- http://dx.doi.org/10.11693/hyhz20210800182
文章历史
-
收稿日期:2021-08-18
收修改稿日期:2021-09-16
水产养殖被认为是向人类提供可食用蛋白质的主要来源之一。随着经济的发展, 人们生活水平日益提高, 对优质蛋白的需求越来越大, 水产品养殖产量也在不断增长(阮思甜, 2021)。目前, 中国已经成为世界上水产品养殖规模最大、水产品消费市场最大的国家(李永涛, 2021)。为了进一步提高养殖产量, 满足市场不断增长的需要, 高密度、集约化养殖成为水产养殖业的发展方向, 这也造成了养殖水体富营养化、养殖环境日益恶化、病害频发等问题(陈聪聪等, 2021)。为减少养殖损失及降低养殖成本, 抗生素被大量投入使用(甘华田, 2019)。但水产养殖中抗生素类药物的使用会带来很多不利影响, 如抗生素抗性基因(antibiotic resistance genes, ARGs)与耐药菌株的产生(金明兰等, 2019)、水产品中抗生素药物残留、破坏微生态平衡以及污染水源等(肖倩, 2020; 包樱钰等, 2021)。其中, 养殖环境中ARGs增多引起的抗生素耐药性是当前面临的一个重大挑战。ARGs使细菌能够耐受更高浓度的抗生素, 可在环境中传播(朱光平等, 2020)且较难清除。研究表明, 水产养殖中抗生素的残留与ARGs的产生和传播息息相关(余军楠, 2020)。ARGs还可以利用整合子、转座子和质粒等可移动遗传元件(mobile genetic elements, MGEs), 通过水平基因转移(horizontal gene transfer, HGT)等方式在人、动物和环境中循环传播, 最终危害人类健康(迟小惠等, 2019; 刘艳英, 2020)。
水产健康养殖是一个复杂的多尺度问题, 涉及养殖活动及其与环境直接和间接的相互作用(Edwards, 2015)。目前, 世界范围内关于有利于水产养殖可持续发展的生产设施和管理方法备受关注(Buck et al, 2018)。大型海藻能够通过光合作用等途径改善和净化养殖水质, 且操作便利, 是一种具有潜力的生物修复技术(Kang et al, 2008; 王春华, 2020)。研究表明, 曲褶刚毛藻(Cladophora flexuosa)可显著降低对虾养殖尾水中的氨态氮和硝酸态氮的含量(廖秀睿等, 2021), 类似的结果在关于芋根江蓠和龙须菜的研究中也有报道(秦文娟等, 2017; 吴翔宇等, 2021)。龙须菜还被证实具有极强的重金属富集能力, 从而对水体重金属污染具有一定的生物修复作用(罗洪添, 2019)。此外, 海藻可应用于食品、水产饲料、化工原料、能源等领域, 具有较高的经济价值(王向辉等, 2021; 周伟等, 2021)。目前, 关于水产养殖对大型海藻表面细菌群落及ARGs的影响尚未见报道。
为了阐明养殖方式对大型海藻生长性能、表面附生细菌群落及ARGs的影响, 本研究构建了两种大型海藻养殖方式。由于异枝江蓠(Gracilaria bailinae)在中国华南沿海分布广泛, 适应性强(Yang et al, 2015), 因此选择其作为代表物种。利用16S rDNA高通量测序技术和Real-time qPCR技术, 研究异枝江蓠表面附生细菌群落及ARGs对养殖方式的响应, 包括: (1) 比较不同养殖方式对异枝江蓠生长性能的影响; (2) 揭示不同养殖方式中异枝江蓠表面附生细菌群落和ARGs的组成与差异; (3) 阐明异枝江蓠表面附生细菌群落与生长性能、ARGs之间的相互关系。本研究将丰富海水养殖微生物生态学知识, 有助于海水养殖环境的优化, 为大型海藻在海水养殖业中的应用与推广提供相关依据。
1 材料与方法 1.1 实验设计本研究于2019年7~9月在广东省阳江梁氏水产养殖有限公司鱼苗繁育基地, 利用南美白对虾(Litopenaeus vannamei)、异枝江蓠(Gracilaria bailinae)构建了两种虎龙杂交石斑鱼(Epinephelus fuscoguttatus ♀ × E. lanceolatus ♂)生态育苗池, 即: “鱼-藻”混养(FA; 虎龙杂交斑100尾/池, 异枝江蓠1 500 g/池)和“鱼-虾-藻”混养(FSA; 虎龙杂交斑100尾/池, 南美白对虾1 000尾/池, 异枝江蓠1 500 g/池)。每个育苗池体积为19.2 m3 (4 m×4 m×1.2 m), 配备独立的进出水系统。养殖品种的养殖密度与其池塘养殖的密度相同。虎龙杂交斑每天投喂两次(8:00和19:00), 日投喂量为虎龙杂交斑体质量的3%。
1.2 海藻生长性能测定于养殖30 d和60 d时, 称量每个育苗池中异枝江蓠的重量。生长性能包括平均增重(average weight gain, AWG)、增重率(percentage of weight gain, PW)、特定生长率(specific growth rate, SGR), 按以下公式计算:
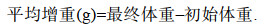 (1)
(1) (2)
(2) (3)
(3)分别于养殖30 d和60 d, 采用“五点取样法”从每个育苗池中采集适量异枝江蓠样品, 装入无菌采样袋中, 液氮速冻并储存于–80 ℃冰箱中, 直到进一步分析。
1.4 DNA提取与高通量测序使用植物DNA小量提取试剂盒(迈宝, 中国)从异枝江蓠样品中提取细菌总基因组DNA。使用超微量微孔板分光光度计(BioTek, 美国)对提取的DNA进行浓度和纯度测定。使用BioRad S1000 (Bio-Rad Laboratory, 美国)扩增16S rDNA高变结构域V4和V5, 扩增引物带有barcodes, 用以区分来自单个样品的PCR产物。PCR扩增结束后, 用1.5%琼脂糖凝胶电泳检测16S rDNA扩增产物。使用Illumina MiSeq高通量测序平台(美格, 中国)对每个样品16S rDNA扩增产物进行测序。
1.5 ARGs和MGEs检测使用LightCycler® 480 Instrument II (Roche, 瑞士)对20个ARGs和4个MGEs进行Real-time qPCR检测, 即: 氨基糖苷类[aac(6')-Ib-01, aac(6')-Ib-02, aac(6')-Ib-03, strB, aac(6')-II, aadA-01, aadA-02, aadA2-01]、β-内酰胺酶类(blaOXA10-01, blaOXA10-02, cphA-01)、MLSB类(mphA-01, mphA-02)、多重耐药类(floR, mexF)、磺胺类(sul2)、四环素类(tetG-02, tetG-01, tetA-02和tetR-02)、转座酶类(tnpA-02, tnpA-04)、整合子类(cIntI-1, intI-1)。用于ARGs/MGEs检测的qPCR引物见表 1。
| 基因名称 | 正向引物 | 反向引物 |
| 16S rRNA | GGGTTGCGCTCGTTGC | ATGGYTGTCGTCAGCTCGTG |
| aac(6')-Ib-01 | GTTTGAGAGGCAAGGTACCGTAA | GAATGCCTGGCGTGTTTGA |
| aac(6')-Ib-02 | CGTCGCCGAGCAACTTG | CGGTACCTTGCCTCTCAAACC |
| aac(6')-Ib-03 | AGAAGCACGCCCGACACTT | GCTCTCCATTCAGCATTGCA |
| aac(6')-II | CGACCCGACTCCGAACAA | GCACGAATCCTGCCTTCTCA |
| aadA-01 | GTTGTGCACGACGACATCATT | GGCTCGAAGATACCTGCAAGAA |
| aadA-02 | CGAGATTCTCCGCGCTGTA | GCTGCCATTCTCCAAATTGC |
| aadA2-01 | ACGGCTCCGCAGTGGAT | GGCCACAGTAACCAACAAATCA |
| blaOXA10-01 | CGCAATTATCGGCCTAGAAACT | TTGGCTTTCCGTCCCATTT |
| blaOXA10-02 | CGCAATTATCGGCCTAGAAACT | TTGGCTTTCCGTCCCATTT |
| cIntI-1 | GGCATCCAAGCAGCAAG | AAGCAGACTTGACCTGA |
| cphA-01 | GCGAGCTGCACAAGCTGAT | CGGCCCAGTCGCTCTTC |
| floR | ATTGTCTTCACGGTGTCCGTTA | CCGCGATGTCGTCGAACT |
| intI-1 | CGAACGAGTGGCGGAGGGTG | TACCCGAGAGCTTGGCACCCA |
| mexF | CCGCGAGAAGGCCAAGA | TTGAGTTCGGCGGTGATGA |
| mphA-01 | CTGACGCGCTCCGTGTT | GGTGGTGCATGGCGATCT |
| mphA-02 | TGATGACCCTGCCATCGA | TTCGCGAGCCCCTCTTC |
| strB | GCTCGGTCGTGAGAACAATCT | CAATTTCGGTCGCCTGGTAGT |
| sul2 | TCATCTGCCAAACTCGTCGTTA | GTCAAAGAACGCCGCAATGT |
| tetA-02 | CTCACCAGCCTGACCTCGAT | CACGTTGTTATAGAAGCCGCATAG |
| tetG-01 | TCAACCATTGCCGATTCGA | TGGCCCGGCAATCATG |
| tetG-02 | CATCAGCGCCGGTCTTATG | CCCCATGTAGCCGAACCA |
| tetR-02 | CGCGATAGACGCCTTCGA | TCCTGACAACGAGCCTCCTT |
| tnpA-02 | GGGCGGGTCGATTGAAA | GTGGGCGGGATCTGCTT |
| tnpA-04 | CCGATCACGGAAAGCTCAAG | GGCTCGCATGACTTCGAATC |
Real-time qPCR反应条件(Suzuki et al, 2019; Wu et al, 2019)。在384孔板上制备12.5 μL qPCR反应体系, 包含6.25 μL SYBR qPCR Master Mix (诺唯赞, 中国)、4.25 μL ddH2O、0.5 μL正、反引物和1 μL模板DNA。qPCR反应条件为: 95 ℃ 10 min, 95 ℃ 15 s, 60 ℃ 60 s, 40个循环。ARGs/MGEs拷贝数计算公式为10×[(31–Ct)/(10/3)], 其中31为检测限。为了减少提取效率的差异, 将ARGs或MGEs拷贝数归一化为16S rRNA基因拷贝(ARGs或MGEs拷贝/16S rRNA基因拷贝), 定义为相对丰度(Han et al, 2018)。
1.6 数据分析使用MOTHUR 1.37.6对测序产生的原始序列进行处理, 并根据97%的相似性阈值聚类可操作分类单元(OTUs)。随后, 根据SILVA数据库, 使用MOTHUR软件以80%置信阈值对代表OTU进行物种注释。采用SPSS 20.0进行数据统计分析, 使用Origin 9.0绘制图表。Canoco 4.5软件进行冗余分析。
2 实验结果 2.1 生长性能由表 2可以看出, 在两次采样中, FSA组的异枝江蓠末均重(final weight, FW)、平均增重(average weight gain, AWG)、增重率(percentage of weight gain, PW)均显著高于FA组(P < 0.05)。在30 d时, FSA组的异枝江蓠特定生长率显著高于FS组(P < 0.05), 而到60 d时则无显著差异(P > 0.05)。
| 组别 | FW/g | AWG/g | PW/% | SGR/% |
| FA-1 | 347.66±12.45a | 47.66±12.45a | 15.89±4.15a | 0.48±0.12a |
| FSA-1 | 434.84±27.60bc | 134.84±27.60bc | 44.95±9.20bc | 1.20±0.21b |
| FA-2 | 386.96±17.49ab | 86.96±17.49ab | 28.99±5.83ab | 0.41±0.07a |
| FSA-2 | 511.31±49.81c | 211.31±49.81c | 70.44±16.60c | 0.84±0.17ab |
| 注: FW: 末均重; AWG: 平均增重; PW: 增重率; SGR: 特定生长率; FA: “鱼-藻”混养; FSA: “鱼-虾-藻”混养; 1: 养殖30 d; 2: 养殖60 d。下同 | ||||
如图 1a所示, 变形菌门(Proteobacteria, 42.81%~ 58.65%)、蓝藻门(Cyanobacteria, 9.32%~28.10%)、浮霉菌门(Planctomycetes, 6.65%~17.46%)和拟杆菌门(Bacteroidetes, 6.86%~11.29%)是最主要的门。
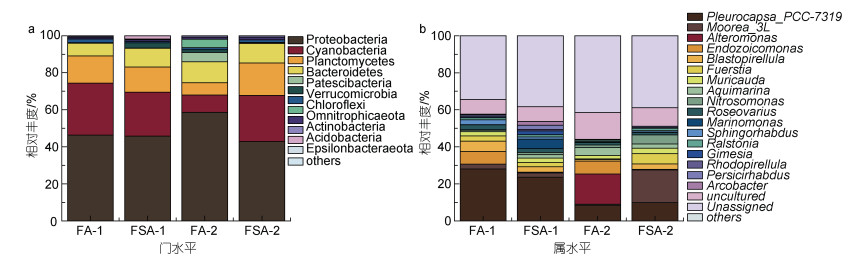 |
| 图 1 异枝江蓠门(a)和属(b)水平(前0.5%)细菌群落的相对丰度 Fig. 1 Relative abundance of bacterial communities at phylum (a) or genus (b) level (first 0.5%) of G. bailinae |
图 1b进一步揭示了属水平(前0.5%)细菌群落的组成特征。各养殖方式中优势菌属组成不同, 且随养殖时间的增加而动态变化。Pleurocapsa_PCC-7319在整个采样期间均占优势, 相对丰度分别为28.07%、23.50%、8.48%、9.90%。此外, FA-1的优势属(相对丰度 > 2%, 下同)有7个, 分别是Moorea_3L (2.60%)、Endozoicomonas (6.90%)、Blastopirellula (5.55%)、Fuerstia (2.77%)、Muricauda (2.30%)、Roseovarius (2.66%)和Sphingorhabdus (2.63%); FSA-1的优势属有9个, 分别是Moorea_3L (2.42%)、Blastopirellula (2.91%)、Fuerstia (2.26%)、Muricauda (2.45%)、Roseovarius (2.07%)、Marinomonas (5.09%)、Gimesia (2.08%)、Persicirhabdus (2.29%)和Arcobacter (2.02%); FA-2的优势属有3个, 分别是Alteromonas (16.32%)、Endozoicomonas (7.02%)和Aquimarina (4.21%); FSA-2的优势属有6个, 分别是Moorea_3L (17.59%)、Blastopirellula (2.99%)、Fuerstia (5.47%)、Muricauda (2.95%)、Aquimarina (2.34%)和Nitrosomonas (4.90%)。
两种养殖方式中异枝江蓠表面附生细菌群落的优势菌属有部分重合, 但FA的优势菌属比FSA的优势菌属少(FA-1比FSA-1少2个优势属, FA-2比FSA-2少3个优势属)。而比较不同的采样时间, 30 d时所采集的海藻中优势菌属比60 d时多(FA-1比FA-2多4个优势属, FSA-1比FSA-2多3个优势属)。可见, 养殖方式和养殖时长对属水平上异枝江蓠表面附生细菌群落组成有显著影响, FSA组异枝江蓠表面附生细菌群落多样性大于FA组。
2.3 ARGs和MGEs的组成特征Pruden等(2006)首次将ARGs作为一种新型的环境污染物提出, 指出ARGs对人类健康及生态环境存在潜在风险。通过检测异枝江蓠表面ARGs的丰度, 可以反映异枝江蓠表面附生细菌群落潜在的耐药性。
本研究两次采样中FSA组(FSA-1: 9.23×10–2; FSA-2: 1.68×10–1)异枝江蓠总的ARGs相对丰度均高于FA组(FA-1: 2.68×10–2; FA-2: 4.90×10–2), 且随着养殖时间的增长, 两种养殖方式中总的ARGs相对丰度均有所上升。
在养殖30 d时, FA-1检测的所有ARGs相对丰度均比FSA-1低(图 2, 图 3, 图 4, 图 5), 其中有13个基因[包括氨基糖苷类4个基因: aac(6')-Ib-01, aac(6')-Ib-03, aac(6')-II和strB; β-内酰胺酶类3个基因: blaOXA10-01, blaOXA10-02和cphA-01; MLSB类2个基因: mphA-01和mphA-02; 多药类1个基因: floR; 磺胺类1个基因: sul2; 四环素类2个基因: tetG-01和tetA-02]显著低于FSA-1 (P < 0.05)。在养殖60 d时, FA-2的β-内酰胺酶类、MLSB类、多药类和磺胺类ARGs相对丰度均低于FSA-2 (图 3, 图 4, 图 5)。在氨基糖苷类和四环素类检测的12个基因中, 有6个基因[氨基糖苷类4个: aac(6')-II, aadA-01, aadA2-01, aadA-02; 四环素类2个: tetA-02, tetR-02]的相对丰度在FA-2中高于FSA-2, 其中aac(6')-II、aadA-01、aadA2-01的相对丰度显著高于FSA-2 (P < 0.05)。
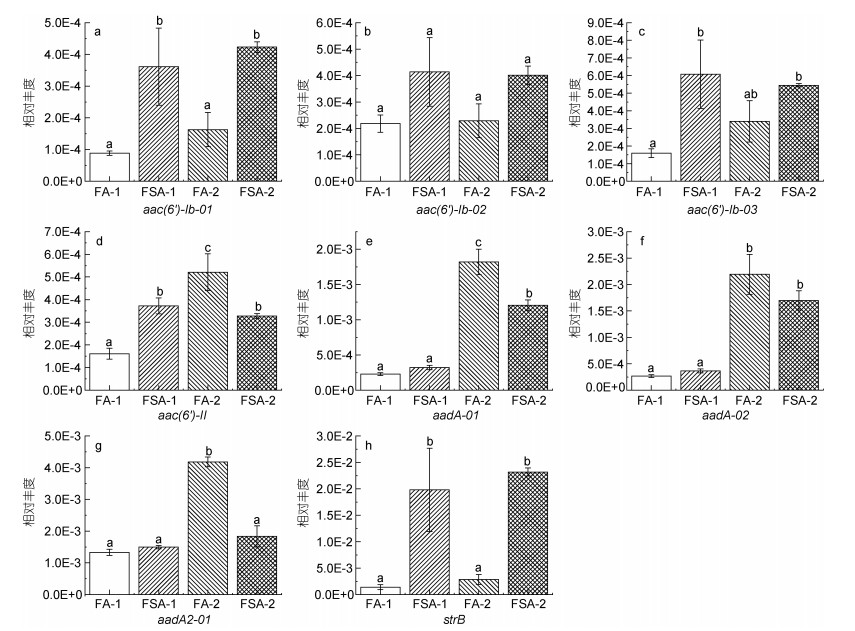 |
| 图 2 氨基糖苷类抗生素抗性基因的相对丰度 Fig. 2 Relative abundance of aminoglycoside antibiotic resistance genes 注: a. aac(6')-Ib-01; b. aac(6')-Ib-02; c. aac(6')-Ib-03; d. aac(6')-II; e. aadA-01; f. aadA-02; g. aadA2-01; h. strB |
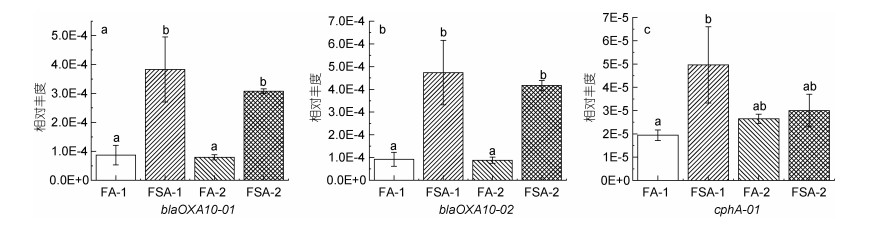 |
| 图 3 β-内酰胺酶类抗生素抗性基因的相对丰度 Fig. 3 Relative abundance of β-lactamase antibiotic resistance genes 注: a. blaOXA10-01; b. blaOXA10-02; c. cphA-01 |
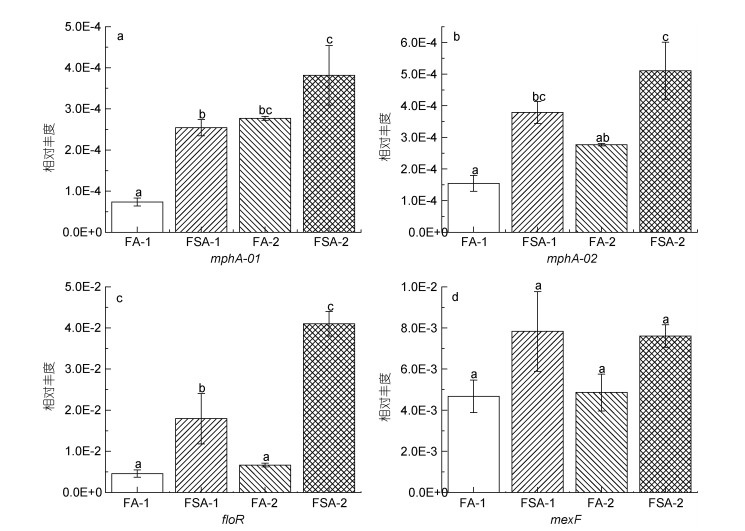 |
| 图 4 MLSB (a, b)和多药(c, d)类抗生素抗性基因的相对丰度 Fig. 4 Relative abundance of MLSB (a, b) and multidrug (c, d) antibiotic resistance genes 注: a. mphA-01; b. mphA-02; c. floR; d. mexF |
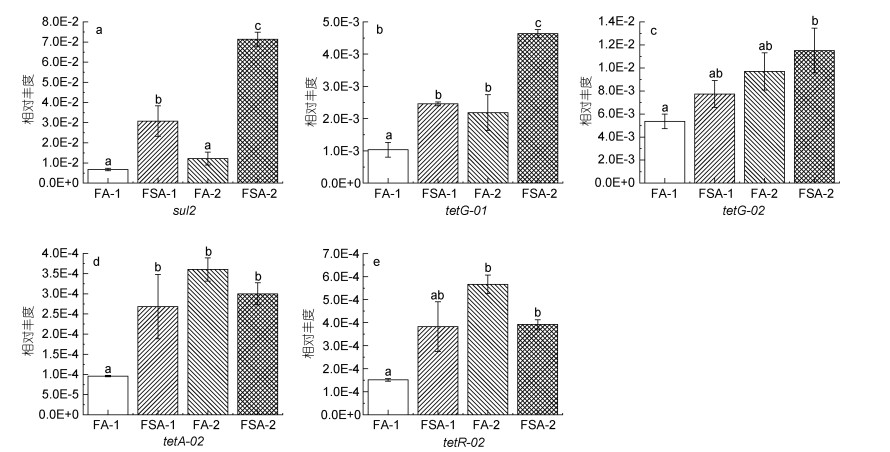 |
| 图 5 磺胺(a)和四环素(b~e)类抗生素抗性基因的相对丰度 Fig. 5 Relative abundance of sulfonamide (a) and tetracycline (b~e) antibiotic resistance genes 注: a. sul2; b. tetG-01; c. tetG-02; d. tetA-02; e. tetR-02 |
ARGs常伴随着MGEs的出现。如图 6所示, 两次采样中FSA组(FSA-1: 1.12×10–2; FSA-2: 1.30×10–2)异枝江蓠总的MGEs相对丰度均高于FA组(FA-1: 7.79×10–3; FA-2: 1.24×10–2)。在两种养殖方式下, FSA组转座酶类基因(tnpA-02和tnpA-04)相对丰度均高于FA组, 其中FSA-1中tnpA-04相对丰度显著高于FA-1组(P < 0.05)。整合子类基因中, cIntI-1在FSA-1中的相对丰度高于FA-1 (P > 0.05), 而在FSA-2中则显著低于FA-2 (P < 0.05); intI-1在FSA中的相对丰度高于FA, 但均无显著差异(P > 0.05)。
 |
| 图 6 可移动遗传元件[转座酶(a, b)和整合子(c, d)]的相对丰度 Fig. 6 Relative abundance of mobile genetic elements [transposase (a, b) and integron (c, d)] 注: a. tnpA-02; b. tnpA-04; c. cIntI-1; d. intI-1 |
细菌群落与异枝江蓠生长性能、ARGs/MGEs之间关系的RDA分析结果见图 7。结果表明特定生长率、增重率与Ralstonia、Muricauda、Fuerstia、Nitrosomonas、Moorea_3L、Aquimarina呈显著正相关, 而与Blastopirellula、Sphingorhabdus、Endozoicomonas、Alteromonas呈显著负相关。Moorea_3L、Nitrosomonas和Fuerstia与tetG-01、mphA-01和tnpA-04呈显著正相关, Alteromonas与aac(6')-Ib-01、aac(6')-Ib-03、blaOXA10-01和blaOXA10-02呈显著正相关, Pleurocapsa_PCC-7319、Gimesia和Endozoicomonas等与tetG-01、mphA-01、tnpA-04、aac(6')-Ib-01、aac(6')-Ib-03、blaOXA10-01和blaOXA10-02呈显著负相关。
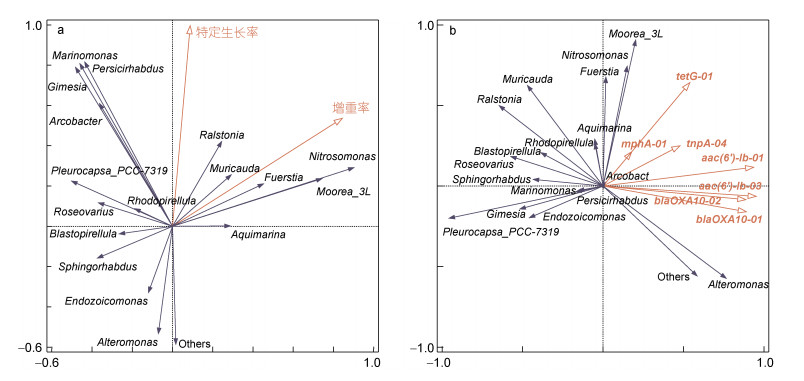 |
| 图 7 细菌群落(属水平, 相对丰度 > 0.5%)与生长性能(a)、ARGs/MGEs (b)之间的相关性 Fig. 7 Correlation between bacterial community (genus level, relative abundance > 0.5%) with growth performance (a) and ARGs/MGEs (b) |
已有多项研究表明, 在水产养殖中不同的养殖方式所产生的经济效益不同(李杰等, 2012; 郭文学等, 2014), 人们也一直在探索最优的养殖方式。Kang等(2021)研究表明, 刺松藻(Codium fragile)、孔石莼(Ulva pertusa)等能有效去除鱼类养殖废水中的氮、磷, 适合于综合养殖和生物修复。Nardelli等(2019)研究表明, 用含鱼类和贝类排泄物的废水培养石莼(Ulva lactuca L.), 可以促进其生长。本研究以虎龙杂交斑、南美白对虾和大型海藻异枝江蓠为养殖对象, 构建了两种混养方式。FSA组异枝江蓠的末均重、平均增重、增重率均显著高于FA组, 这可能与南美白对虾的生物扰动有关。研究表明, 生物扰动可以促进沉积物中营养物质的释放, 增加水体中无机营养盐的含量(Nicholaus et al, 2019), 这可能会促进异枝江蓠的生长。
微生物和藻类之间的互相作用会影响藻类的生长(Goecke et al, 2010)。在本研究中, 异枝江蓠表面附生细菌群落主要为变形菌门、蓝藻门、浮霉菌门和拟杆菌门, 与Huo等(2012)的研究结果相似。FSA组的异枝江蓠表面附生细菌群落多样性较高, 细菌群落之间相互作用可能会促进藻类的生长(Mouget et al, 1995; Marshall et al, 2006)。RDA分析表明, 异枝江蓠生长性能与Ralstonia、Muricauda、Fuerstia等呈显著正相关, 说明这些菌属可能对异枝江蓠的生长有益。Rawat等(2021)的研究发现, 索氏小球藻(Chlorella sorokiniana)和罗尔斯顿菌(Ralstonia pickettii)的共生关系提高了藻类的生长速率, 这与本研究结果类似。
3.2 两种不同混养方式中ARGs/MGEs丰度、类型及其与细菌群落的相关性分析抗生素因其治疗传染病的能力而被广泛用于维持人类和动物的健康(Yan et al, 2020)。然而, 随着抗生素类药物在水产养殖中的持续使用, ARGs和耐药菌株的产生所带来的问题也日益突出(Zhang et al, 2009)。Manage(2018)指出水生环境中细菌的抗性基因可以通过水平基因转移传播到人类病原体上, 最终危害人类健康。而MGEs被认为是促进ARGs水平基因转移的关键因素(Nikolakopoulou et al, 2008)。此外, 多项研究表明, 某些海藻提取物具有一定的抑菌效果, 可能诱导其表面附生菌群产生ARGs (Turcotte et al, 2016; Ge et al, 2019; Xiao et al, 2019)。因此, 通过检测ARGs和MGEs的丰度和多样性可以反映其潜在的生态风险(Nnadozie et al, 2019)。本研究中, 所检测20种ARGs和4种MGEs在所有样品中均有出现。值得注意的是, 两次采样中FSA组异枝江蓠ARGs和MGEs的相对丰度大多高于FA组。
ARGs/MGEs的丰度和种类与细菌群落密切相关(芦科堃等, 2019; Zheng et al, 2021)。Zhang等(2020)的研究表明变形菌门(Proteobacteria)是ARGs的潜在宿主之一, 这与本研究的结果一致。在本研究中, 属于变形菌门的Nitrosomonas与多种ARGs显著正相关, 可能是其潜在宿主。此外, Nitrosomonas在FSA组的相对丰度较高, 可能是导致FSA组ARGs的相对丰度高于FA组的原因之一。蓝藻门、浮霉菌门等被认为是ARGs的重要储存库, 且具有含ARGs的MGEs, 会促进ARGs的增殖与传播(Cayrou et al, 2010; Dias et al, 2019; Wang et al, 2020), 这与本研究的结果一致。本研究中, 转座酶基因tnpA-04与Moorea_3L (属于蓝藻门)、Fuerstia (属于浮霉菌门)等密切正相关, 而这些菌属在FSA组中丰度较高, 这进一步促进了FSA组ARGs的增多。Wang等(2019)研究表明, 虾池水中ARGs的总丰度高于水源地和农场污水。Fang等(2019)研究发现, ARGs的相对丰度与MGEs的水平呈显著正相关。因此, 混养南美白对虾可能会进一步增加FSA组ARGs和MGEs相对丰度。
混养物种和细菌群落共同影响了异枝江蓠的生长性能和ARGs/MGEs的组成与分布。但是, 本研究养殖时间相对较短, 需要在后续的试验中进一步深入探究不同混养方式对异枝江蓠的持续影响, 解析潜在的影响机制。
4 结论本研究利用16S rDNA高通量测序技术和Real-time qPCR技术揭示了两种不同混养方式对异枝江蓠生长性能、表面附生细菌群落及抗生素抗性基因的影响。发现“鱼-虾-藻”混养能够显著增强异枝江蓠的生长性能, 提高异枝江蓠表面附生细菌群落的多样性。“鱼-虾-藻”混养能够增加异枝江蓠ARGs和MGEs的相对丰度, 具有一定的生态风险, 因此在提高经济效益的同时也需要关注可能存在的对人类健康的危害。
王向辉, 刘又华, 李文丽, 等. 2021. 海藻生物炭对水溶液中甲基橙的吸附效果与机制. 水处理技术, 47(7): 42-47, 53 |
王春华. 2020. 水产养殖水体污染的生物修复技术. 渔业致富指南, (8): 34-36 |
甘华田. 2019. 加强养殖业抗生素监管织起食品安全网. 绿色中国, (14): 68-69 |
包樱钰, 李菲菲, 温东辉. 2021. 我国海水养殖业的抗生素污染现状. 海洋环境科学, 40(2): 294-302 |
朱光平, 薛晨晨, 范宏亮, 等. 2020. 环境中抗生素抗性基因研究进展. 预防医学, 32(11): 1121-1125, 1129 |
刘艳英. 2020. 抗生素抗性基因在环境中的分布及对人体的危害. 厦门科技, (6): 31-34 |
阮思甜. 2021. 我国水产养殖业需加快推进高质量发展. 农经, (S1): 46-50 |
芦科堃, 向文良, 卢倩文. 2019. 成都平原不同种植方式折耳根表面附生细菌群落结构与抗生素抗性基因分析. 微生物学报, 59(3): 489-498 |
李永涛. 2021. 水产品消费研究述评. 中国水产, (1): 70-73 |
李杰, 雷驰宙, 陈伟洲. 2012. 南澳贝藻混养互利机制的初步研究. 水产科学, 31(8): 449-453 DOI:10.3969/j.issn.1003-1111.2012.08.001 |
肖倩. 2020. 水产养殖中抗生素滥用问题研究. 养殖与饲料, 19(10): 46-48 DOI:10.3969/j.issn.1671-427X.2020.10.021 |
吴翔宇, 黄良夫, 杨守国, 等. 2021. 芋根江蓠对对虾养殖水体氮磷的去除效果. 现代农业科技, (8): 178-179 DOI:10.3969/j.issn.1007-5739.2021.08.076 |
余军楠, 2020. 江苏三种典型甲壳类动物水产养殖环境中抗生素及抗性基因污染特征研究[D]. 南京: 南京信息工程大学: 1-2.
|
迟小惠, 冯友军, 郑焙文. 2019. 耐药菌在人-动物-环境中的传播和遗传机制. 微生物学通报, 46(2): 311-318 |
陈聪聪, 杨玉艳. 2021. 水产养殖对水环境的影响和解决方法. 河北农机, (1): 75-76 |
罗洪添, 2019. 大型海藻龙须菜对重金属的生物修复效应[D]. 广州: 暨南大学: 34-40.
|
金明兰, 李华南, 赵鑫, 等. 2019. 养禽场周围水环境中磺胺类抗性菌、抗性基因分布. 科学技术与工程, 19(9): 266-270 |
周伟, 邓银银, 田翠翠, 等. 2021. 大型绿藻浒苔的产业化开发应用. 广西科学, 28(2): 113-118 |
秦文娟, 陆开宏, 郑忠明, 等. 2017. 龙须菜与缢蛏单养或混养对对虾集约化养殖尾水净化的效果. 宁波大学学报(理工版), 30(6): 35-41 |
郭文学, 尹家胜, 佟广香, 等. 2014. 养殖方式、光照强度对哲罗鱼稚鱼生长与存活的影响. 海洋与湖沼, 45(2): 265-273 |
廖秀睿, 李曦, 柳睿杰, 等. 2021. 曲褶刚毛藻在对虾养殖尾水无机氮盐净化中的应用. 海南热带海洋学院学报, 28(2): 1-5 |
BUCK B H, TROELL M F, KRAUSE G et al, 2018. State of the art and challenges for offshore integrated multi-trophic aquaculture (IMTA). Frontiers in Marine Science, 5: 165 DOI:10.3389/fmars.2018.00165 |
CAYROU C, RAOULT D, DRANCOURT M, 2010. Broad-spectrum antibiotic resistance of Planctomycetes organisms determined by Etest. Journal of Antimicrobial Chemotherapy, 65(10): 2119-2122 DOI:10.1093/jac/dkq290 |
DIAS E, OLIVEIRA M, MANAGEIRO V et al, 2019. Deciphering the role of cyanobacteria in water resistome: hypothesis justifying the antibiotic resistance (phenotype and genotype) in Planktothrix genus. Science of the Total Environment, 652: 447-454 DOI:10.1016/j.scitotenv.2018.10.167 |
EDWARDS P, 2015. Aquaculture environment interactions: past, present and likely future trends. Aquaculture, 447: 2-14 DOI:10.1016/j.aquaculture.2015.02.001 |
FANG H, HUANG K L, YU J N et al, 2019. Metagenomic analysis of bacterial communities and antibiotic resistance genes in the Eriocheir sinensis freshwater aquaculture environment. Chemosphere, 224: 202-211 DOI:10.1016/j.chemosphere.2019.02.068 |
GE H X, NI Q, CHEN Z et al, 2019. Effects of short period feeding polysaccharides from marine macroalga, Ulva prolifera on growth and resistance of Litopenaeus vannamei against Vibrio parahaemolyticus infection. Journal of Applied Phycology, 31(3): 2085-2092 DOI:10.1007/s10811-018-1663-3 |
GOECKE F, LABES A, WIESE J et al, 2010. Chemical interactions between marine macroalgae and bacteria. Marine Ecology Progress Series, 409: 267-299 DOI:10.3354/meps08607 |
HAN Y, WANG J, ZHAO Z L et al, 2018. Combined impact of fishmeal and tetracycline on resistomes in mariculture sediment. Environmental Pollution, 242: 1711-1719 DOI:10.1016/j.envpol.2018.07.101 |
HUO Y Z, WU H L, CHAI Z Y et al, 2012. Bioremediation efficiency of Gracilaria verrucosa for an integrated multi-trophic aquaculture system with Pseudosciaena crocea in Xiangshan harbor, China. Aquaculture, 326/329: 99-105 DOI:10.1016/j.aquaculture.2011.11.002 |
KANG Y H, KIM S, CHOI S K et al, 2021. A comparison of the bioremediation potential of five seaweed species in an integrated fish-seaweed aquaculture system: implication for a multi-species seaweed culture. Reviews in Aquaculture, 13(1): 353-364 DOI:10.1111/raq.12478 |
KANG Y H, SHIN J A, KIM M S et al, 2008. A preliminary study of the bioremediation potential of Codium fragile applied to seaweed integrated multi-trophic aquaculture (IMTA) during the summer. Journal of Applied Phycology, 20(2): 183-190 DOI:10.1007/s10811-007-9204-5 |
MANAGE P M, 2018. Heavy use of antibiotics in aquaculture: emerging human and animal health problems–a review. Aquatic Sciences, 23(1): 13-27 |
MARSHALL K, JOINT I, CALLOW M E et al, 2006. Effect of marine bacterial isolates on the growth and morphology of axenic plantlets of the green alga Ulva linza. Microbial Ecology, 52(2): 302-310 DOI:10.1007/s00248-006-9060-x |
MOUGET J L, DAKHAMA A, LAVOIE M C et al, 1995. Algal growth enhancement by bacteria: is consumption of photosynthetic oxygen involved?. FEMS Microbiology Ecology, 18(1): 35-43 DOI:10.1016/0168-6496(95)00038-C |
NARDELLI A E, CHIOZZINI V G, BRAGA E S et al, 2019. Integrated multi-trophic farming system between the green seaweed Ulva lactuca, mussel, and fish: a production and bioremediation solution. Journal of Applied Phycology, 31(2): 847-856 DOI:10.1007/s10811-018-1581-4 |
NICHOLAUS R, LUKWAMBE B, ZHAO L et al, 2019. Bioturbation of blood clam Tegillarca granosa on benthic nutrient fluxes and microbial community in an aquaculture wastewater treatment system. International Biodeterioration & Biodegradation, 142: 73-82 |
NIKOLAKOPOULOU T L, GIANNOUTSOU E P, KARABATSOU A A et al, 2008. Prevalence of tetracycline resistance genes in Greek seawater habitats. The Journal of Microbiology, 46(6): 633-640 DOI:10.1007/s12275-008-0080-8 |
NNADOZIE C F, ODUME O N, 2019. Freshwater environments as reservoirs of antibiotic resistant bacteria and their role in the dissemination of antibiotic resistance genes. Environmental Pollution, 254: 113067 DOI:10.1016/j.envpol.2019.113067 |
PRUDEN A, PEI R T, STORTEBOOM H et al, 2006. Antibiotic resistance genes as emerging contaminants: studies in northern Colorado. Environmental Science & Technology, 40(23): 7445-7450 |
RAWAT D, SHARMA U, PORIA P, et al, 2021. Iron-dependent mutualism between Chlorella sorokiniana and Ralstonia pickettii forms the basis for a sustainable bioremediation system[J]. BioRxiv, doi: 10.1101/2021.06.15.446916.
|
SUZUKI S, NAKANISHI S, TAMMINEN M et al, 2019. Occurrence of sul and tet(M) genes in bacterial community in Japanese marine aquaculture environment throughout the year: profile comparison with Taiwanese and Finnish aquaculture waters. Science of the Total Environment, 669: 649-656 DOI:10.1016/j.scitotenv.2019.03.111 |
TURCOTTE F, MOUGET J L, GENARD B et al, 2016. Prophylactic effect of Haslea ostrearia culture supernatant containing the pigment marennine to stabilize bivalve hatchery production. Aquatic Living Resources, 29(4): 401 DOI:10.1051/alr/2016032 |
WANG Z Y, CHEN Q W, ZHANG J Y et al, 2020. Critical roles of cyanobacteria as reservoir and source for antibiotic resistance genes. Environment International, 144: 106034 DOI:10.1016/j.envint.2020.106034 |
WANG L L, SU H C, HU X J et al, 2019. Abundance and removal of antibiotic resistance genes (ARGs) in the rearing environments of intensive shrimp aquaculture in South China. Journal of Environmental Science and Health, Part B, 54(3): 211-218 DOI:10.1080/03601234.2018.1550310 |
WU J J, SU Y L, DENG Y Q et al, 2019. Spatial and temporal variation of antibiotic resistance in marine fish cage-culture area of Guangdong, China. Environmental Pollution, 246: 463-471 DOI:10.1016/j.envpol.2018.12.024 |
XIAO X, LI C, HUANG H M, ET al, 2019. Inhibition effect of natural flavonoids on red tide alga Phaeocystis globosa and its quantitative structure-activity relationship. Environmental Science and Pollution Research, 26(23): 23763-23776 DOI:10.1007/s11356-019-05482-7 |
YAN W F, BAI R, WANG S Q et al, 2020. Antibiotic resistance genes are increased by combined exposure to sulfamethoxazole and naproxen but relieved by low-salinity. Environment International, 139: 105742 DOI:10.1016/j.envint.2020.105742 |
YANG Y F, CHAI Z Y, WANG Q et al, 2015. Cultivation of seaweed Gracilaria in Chinese coastal waters and its contribution to environmental improvements. Algal Research, 9: 236-244 DOI:10.1016/j.algal.2015.03.017 |
ZHANG Y R, YANG Z H, XIANG Y P et al, 2020. Evolutions of antibiotic resistance genes (ARGs), class 1 integron-integrase (intI1) and potential hosts of ARGs during sludge anaerobic digestion with the iron nanoparticles addition. Science of the Total Environment, 724: 138248 DOI:10.1016/j.scitotenv.2020.138248 |
ZHANG X X, ZHANG T, FANG H H P, 2009. Antibiotic resistance genes in water environment. Applied Microbiology and Biotechnology, 82(3): 397-414 DOI:10.1007/s00253-008-1829-z |
ZHENG Z Z, LI L F, MAKHALANYANE T P et al, 2021. The composition of antibiotic resistance genes is not affected by grazing but is determined by microorganisms in grassland soils. Science of the Total Environment, 761: 143205 DOI:10.1016/j.scitotenv.2020.143205 |
 2022, Vol. 53
2022, Vol. 53


