中国海洋湖沼学会主办。
文章信息
- 郭笳, 谢慧盈, 张振, 胡利莎, 刘玉盟, 马培振, 王海艳, 李翠. 2020.
- GUO Jia, XIE Hui-Ying, ZHANG Zhen, HU Li-Sha, LIU Yu-Meng, MA Pei-Zhen, WANG Hai-Yan, LI Cui. 2020.
- 中国常见贻贝基于28S rDNA序列的系统发育分析
- PHYLOGENETIC ANALYSIS BASED ON PARTIAL 28S RDNA SEQUENCE OF FAMILY MYTILIDAE IN CHINA
- 海洋与湖沼, 51(3): 613-621
- Oceanologia et Limnologia Sinica, 51(3): 613-621.
- http://dx.doi.org/10.11693/hyhz20200200038
文章历史
-
收稿日期:2020-02-10
收修改稿日期:2020-03-18
2. 中国科学院大学 北京 100049;
3. 中国海洋大学 青岛 266100;
4. 中国科学院海洋大科学研究中心 青岛 266071
2. University of Chinese Academy of Science, Beijing 100049, China;
3. Ocean University of China, Qingdao, 266100;
4. Center for Ocean Mega-Science, Chinese Academy of Sciences, Qingdao 266071, China
贻贝科在分类单元上属于软体动物门(Mollusca)、双壳纲(Bivalvia)、翼形亚纲(Pteriomorphia)、贻贝目(Mytilida), 中国记载在册的种有70余种(王祯瑞, 1997), 是我国重要的海洋经济作物。对贻贝科较为系统的分类始于Newell (1969), 他结合现生和化石双壳类特征, 将贻贝科Mytilidae并分为4个亚科, 分别为:贻贝亚科Mytilinae; 细齿蛤亚科Crenellinae; 偏顶蛤亚科Modiolinae; 石蛏亚科Lithophaginae。后来的分类学者在此基础上不断增加新的亚科, Bieler等(2010)在前人基础上将贻贝科分为8个亚科, 包括贻贝亚科Mytilinae、细齿蛤亚科Crenellinae、石蛏亚科Lithophaginae、偏顶蛤亚科Modiolinae、深海贻贝亚科Bathymodiolinae、亚科Septiferinae、亚科Dacrydiinae、亚科Limnoperninae; Morton (2015)将贻贝科提升到超科水平并重新划分为4个科13个亚科。目前, 国内外分类学者依据形态对贻贝科内各阶元的划分尚未达成一致结论。我国分类学者王祯瑞(1997)主要依据Newell (1969)的分类观点系统建立了中国海域贻贝分类系统(见表 1), 此后我国分类学者多沿用王祯瑞(1997)的分类系统(徐凤山等, 2008;杨文等, 2013)。
| 科 | 亚科 | 属 | 种数(种) |
| Mytilidae | Mytilinae | Mytilus | 2 |
| Perna | 1 | ||
| Trichomya | 1 | ||
| Septifer | 6 | ||
| Brachidontes | 3 | ||
| Hormomya | 2 | ||
| Crenellinae | Musculus | 8 | |
| Arvella | 1 | ||
| Solamen | 1 | ||
| Arcuatula | 1 | ||
| Gregariella | 2 | ||
| Trichomusculus | 2 | ||
| Lithophaginae | Lithophaga | 16 | |
| Modiolinae | Modiolus | 12 | |
| Idasola | 1 | ||
| Amygdalum | 3 | ||
| Botula | 1 | ||
| Xenostrobus | 1 | ||
| Stavelia | 1 | ||
| Limnoperna | 1 |
随着分子系统学研究的不断深入, DNA条形码技术(Hebert et al, 2003)、分子系统发育分析被用于解决贻贝分类问题, 如Owada(2007)通过分子数据构建系统发育关系, 发现Lithophaginae 为并系群, Leiosolenus和Lithophaga形态相似是由于趋同进化导致。Lee等(2004)通过比较DNA序列发现弗罗里达Brachidontes exustus群体存在4种隐存种。我国近年来仍旧沿用依据形态分类构建的贻贝分类系统, 亟需辅助分子手段重建贻贝科演化关系。28S rDNA是核糖体编码基因中最长的序列, 包含12个变异率较高的序列(D1-D12), 其中D2、D3区不仅变异率高且保守度高, 故而常作为物种间亲缘关系及系统进化的研究依据(Dietrich et al, 2001)。本研究将着眼于贻贝科这一重要海洋经济类群, 利用28S rDNA基因序列片段进行系统发育分析, 探讨中国常见贻贝物种之间的亲缘关系, 这一工作将对完善我国海洋贝类分类体系有重要意义, 为中国的海洋生物多样性开发与保护提供重要资料。
1 材料与方法 1.1 样本信息采集本研究所用样品涉及贻贝科6亚科12属28种, 其中10种为本次研究测序, 18种来自GenBank。测序样品采集于2011—2018年, 95%酒精固定保存并拍照记录其壳形(见图 1)。外群的合理选择直接影响系统发生分析的可靠性, 本研究选取与贻贝目贻贝科物种亲缘关系较近的帘蛤目(Veneroida)的沙筛贝(Mytilopsis sallei)作为系统发育树的外群, 在GenBank检索贻贝科常见物种以及外群沙筛贝共计31条28S rDNA基因片段序列, 各物种所属科属和登录号等信息见表 2。
 |
| 图 1 本实验测序样本照片 Fig. 1 Photos of sequenced samples in this study |
| 目 | 科 | 亚科 | 属 | 种 | GenBank序列号 |
| Mytilida | Mytilidae | Crenellinae | Arcuatula | Arcuatula senhousia | AB103122.1 |
| Arcuatula senhousia | * MN892536 | ||||
| Bathymodiolinae | Bathymodiolus | Bathymodiolus heckerae | MG520058.1 | ||
| Gigantidas childressi | MG520057.1 | ||||
| Gigantidas mauritanicus | MG520059.1 | ||||
| Lithophaginae | Leiosolenus | Leiosolenus lima | KY081354.1 | ||
| Leiosolenus lischkei | AB103123.1 | ||||
| Lithophaga | Lithophaga nigra | AB103127.1 | |||
| Lithophaga teres | KY081359.1 | ||||
| Modiolinae | Botula | Botula cinnamomea | AB103128.1 | ||
| Botula cinnamomea | * MN889038 | ||||
| Modiolus | Modiolus auriculatus | AJ307537.1 | |||
| Modiolus comptus | * MN892541 | ||||
| Modiolus modiolus | EF526455.1 | ||||
| Modiolus nipponicus | AB103125.1 | ||||
| Modiolus rufus | KC429423.1 | ||||
| Xenostrobus | Xenostrobus atratus | AB594397.1 | |||
| Xenostrobus atratus | * MN889242 | ||||
| Mytilinae | Mytilus | Mytilus edulis | KC429424.1 | ||
| Mytilus galloprovincialis | *MN892539 | ||||
| Mytilus galloprovincialis | AB103129.1 | ||||
| Mytilus unguiculatus | * MN889245 | ||||
| Mytilus unguiculatus | * MN892543 | ||||
| Brachidontes | Brachidontes exustus | KT757838.1 | |||
| Brachidontes mutabilis | KY081353.1 | ||||
| Brachidontes mutabilis | AB103124.1 | ||||
| Brachidontes setiger | * MN892542 | ||||
| Brachidontes variabilis | * MN892540 | ||||
| Brachidontes variabilis | KY081350.1 | ||||
| Perna | Perna canaliculus | MK419109.1 | |||
| Perna viridis | KY081366.1 | ||||
| Perna viridis | * MN892544 | ||||
| Septiferinae | Mytilisepta | Mytilisepta bifurcata | KJ453830.1 | ||
| Mytilisepta virgata | AB103126.1 | ||||
| Mytilisepta virgata | * MN892545 | ||||
| Mytilisepta virgata | KJ453833.1 | ||||
| Mytilisepta virgata | KJ453832.1 | ||||
| Septifer | Septifer bilocularis | * MN889243 | |||
| Septifer excisus | AB103130.1 | ||||
| Septifer excisus | KY081368.1 | ||||
| Septifer excisus | KY081367.1 | ||||
| Veneroida | Dreissenidae | Mytilopsis | Mytilopsis sallei | JX099497.1 | |
| 注: *表示本次研究测序的样本 | |||||
从贻贝样品中取出闭壳肌处肌肉20—30μg, 参照TIANamp Marine Animals DNA Kit试剂盒(离心柱型)操作说明提取DNA。通过聚合酶链式反应polymerase chain reac-tion(PCR)获得28S rDNA基因片段。实验扩增使用引物序列为: 28SF: 5′-GGGACTA CCCCCTGAATTTAAGCAT-3′, 28SR: 5′-CCAGCTA TCCTGAGGGAAACTTCG-3′。25μL的PCR反应体系, 其中引物0.2μmol/L, 50—70ng DNA模板, 2×PCR Taq mix12.5μL, ddH2O加至25μL。PCR仪循环参数设置如下: 94℃预变性5min, 30个扩增循环(94℃变性45s, 56℃退火1min, 72℃延伸1min), 72℃延伸10min, 4℃保存。PCR扩增产物送至上海生工有限公司进行测序。使用BLAST(http://blast.ncbi.nlm.nih.gov/Blast.cgi.)对所得序列进行同源性检索, 以检验物种鉴别的正确性, 为系统发育分析做准备。
1.3 系统发育分析与遗传距离本次研究获得的28S rDNA片段使用Clustal W(Thompson et al, 1994)进行多序列比对, 并进行人工校对, 删除结果中的空位及其他对比模糊区域, 调整部分空位位置。使用MEGA X(Kumar et al, 2018)分析序列的碱基组成, 计算物种两两之间的Kimura 2-parameter遗传距离。
本研究选用最大似然法(Maximum Likelihood, ML)和贝叶斯推论法(Bayesian inference, BI)进行系统发生分析。系统发育树的构建使用PhyloSuite v1.1.16 (Zhang et al, 2020)软件, 操作步骤如下:选择MAFFT (Katoh et al, 2013)自动模式进行序列常规对比; 应用ModelFinder (Kalyaanamoorthy et al, 2017)对28S rDNA部分序列筛选模型, 得到基于贝叶斯信息准则(Bayesian Information Criterion, BIC)选择核苷酸最佳替换模型, 并估计相关参数; 以沙筛贝为外群, 使用MrBayes 3.2.6 (Ronquist et al, 2012)构建贝叶斯树时, 采用ModelFinder推荐的最佳替换模型GTR+F+I+G4, 同时构建4条马尔代夫链, 以随机数为起始树, 共运行1000万代, 每10000代抽样一次, 初始25%的采样数据作为老化数据丢弃, 根据剩余样本构建一致树; ML树的构建使用软件IQ-TREE (Nguyen et al, 2015)自动选择模式, 对5000个超快样本进行引导, 并使用SH-aLRT branch test (Guindon et al, 2010), 经1000次自展法(bootstrap)确定拓扑结构的置信度。最终得到的系统发育树使用软件Figtree v 1.4.3(http://tree.bio.ed.ac.uk/software/figtree/)查看和编辑。
2 结果与分析 2.1 序列组成及遗传距离分析结果本研究共获得贻贝科40个个体的28S rDNA序列, 与作为外群的沙筛贝共同进行序列比对后, 去除引物段碱基并删除缺失位点后, 对773bp共有序列进行分析显示, 保守位点447个, 约占57.83%;变异位点326个, 简约信息位点233个, 自裔位点93个。T、C、A、G碱基平均含量为20.02%、27.24%、20.39%、32.35%。其中, A+T含量(40.41%)小于G+C含量(59.59%), 表现出一定的碱基组成偏倚。
通过计算物种28S rDNA基因片段序列两两之间遗传距离, 获得种内、属内、属间物种遗传距离分布箱线图(图 2)。研究发现:本研究贻贝科种内遗传距离的平均值为0.01;同属物种间的平均遗传距离值为0.034, 其中80%小于0.08, 但刻缘短齿蛤与短齿蛤属其他物种的遗传距离均大于0.1, 显著大于属内种间平均遗传距离0.034;不同属物种间的遗传距离平均值为0.115, 91%在0.08—0.20之间; 以上数据说明遗传距离随着分类阶元的不断提高而增大。由图可知, 一般情况下, 同属物种的遗传距离差异较小, 只有短齿蛤属的属内种间遗传距离波动范围较大; 此外, 原隔贻贝属(王祯瑞, 1997)在国际上已经划分为Mytilisepta属和Septifer属, 原石蛏属(王祯瑞, 1997)已经划分为Lithophaga属和Leiosolenus属, 本研究中, Mytilisepta属和Septifer属之间的平均遗传距离显著大于0.1, 明显位于属间物种平均遗传距离范围中, 本研究结果支持国际最新划分标准。总体来看, 贻贝种间、属间28S rDNA基因遗传距离差异显著, DNA间隙明显, 因此, 28S rDNA基因可用于贻贝系统分类。
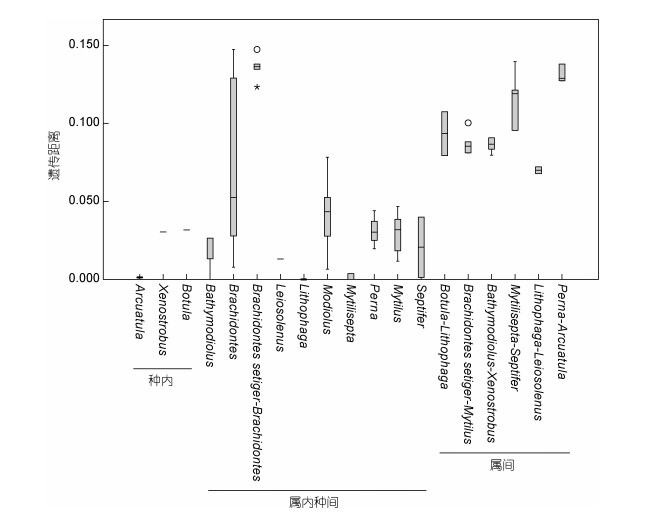 |
| 图 2 基于贻贝科6亚科28S rDNA基因片段序列构建的两两物种间遗传距离箱线图 Fig. 2 Boxplot of genetic distance between each two species for 28S rDNA gene fragments of 6 subfamilies of Mytilidae 注: ‘*’为极值; ‘o’为离群值; ‘-’为平均值; 横轴字母表示属名; 其中Brachidontes setiger-Brachidontes为刻缘短齿蛤与短齿蛤属其他物种间的遗传距离; Brachidontes setiger-Mytilus表示刻缘短齿蛤与贻贝属其他物种之间的遗传距离; Lithophaga-Leiosolenus、Mytilisepta-Septifer、Botula-Lithophaga、Bathymodiolus-Xenostrobus、Perna-Arcuatula均表示两两属间的遗传距离 |
基于28S rDNA重建贝叶斯树和最大似然树(见图 3)。从拓扑结构上来看, 贝叶斯树与最大似然树的结构保持一致, 部分分支的结构与支持率有所差异。两种方法获得的系统发育树均发现贻贝科为单系发生。本研究涉及了贻贝科的6个亚科, 其中深海贻贝亚科Bathymodiolinae和隔贻贝亚科Septiferinae分别为单系群; 贻贝亚科Mytilinae、偏顶蛤亚科Modiolinae、石蛏亚科Lithophaginae的单系性不被支持; 由于细齿蛤亚科Crenellinae仅有一个物种参与建树, 其单系性无法验证。贻贝亚科为多系群, 本亚科中的贻贝属Mytilus、股贻贝属Perna、短齿蛤属Brachidontes未聚集在一起。石蛏亚科的膜石蛏属Leiosolenus的短石蛏Leiosolenus lischkei和锉石蛏Leiosolenus lima单独聚集在一个分支(PP=1, BP=100), 并未与石蛏属Lithophaga的光石蛏Lithophaga teres和Lithophaga nigra聚集在同一枝系。其中石蛏属与偏顶蛤亚科的短壳肠蛤Botula cinnamomea 聚集在同一分支, 但贝叶斯后验概率不高; 膜石蛏属则与深海偏顶蛤亚科物种以及偏顶蛤亚科的Xenostrobus属物种聚集在一起。偏顶蛤亚科Modiolinae中Xenostrobus属、Botula属和偏顶蛤属也未聚为一支, 为多系群。
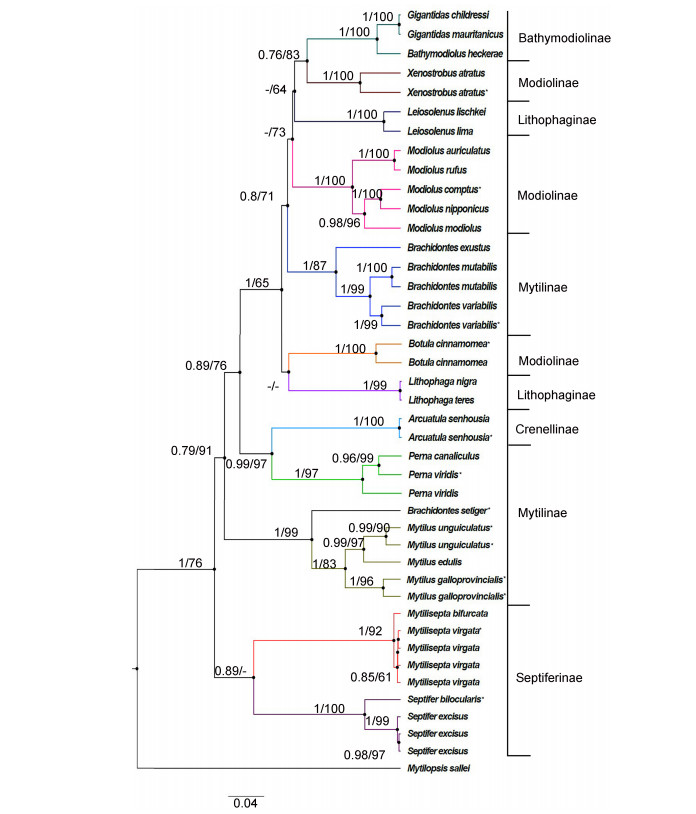 |
| 图 3 贻贝科28物种基于28S rDNA序列构建的贝叶斯树(BI树)和最大似然树(ML树) Fig. 3 Bayesian tree (BI tree) and maximum likelihood tree (ML tree) for 28 species of Mytilidae based on 28S rDNA gene 注:枝上数字代表ML树的自展值bootstraps(BP)和贝叶斯树的后验概率(PP), 颜色相同的分支表示同一属; “*”表示本次实验测序物种, “-”表示节点支持率小于50%;进化树右侧为各属对应亚科名 |
从属单系性来看, 本次研究中, 有8个属在系统发育树中表现为单系群并获得较高支持率, 即: Bathymodiolus属(PP=1, BP=100)、膜石蛏属Leiosolenus (PP=1, BP=100)、Modiolus属(PP=1, BP=100)、石蛏属Lithophaga(PP=1, BP=99)、Perna属(PP=1, BP=97)、Mytilus属(PP=1, BP=83)、Mytilisepta属(PP=1, BP=92)和Septifer属(PP=1, BP=100);短齿蛤属Brachidontes的单系性不被支持, 其中,刻缘短齿蛤Brachidontes setiger并未与短齿蛤属其他物种聚集在一处, 而是与贻贝属的物种聚集在同一分支且节点支持率较高(PP=1, BP=99);另外Xenostrobus属、Botula属和Arcuatula属在本次研究中只有一个物种参与构建系统发育树, 目前无法判断其单系性。
3 讨论 3.1 不同建树方法的比较ML法和BI法构建的系统发育树基本结构相同; ML树与BI树之间的差异主要表现在:隔贻贝属与Mytilisepta 属是否为姊妹群, 共同构成隔贻贝亚科; Lithophaga 属和Botula属是否为姊妹群; Xenostrobus属与深海偏顶蛤属是否为姊妹群; 此外, 最大似然树的部分节点置信度优于贝叶斯树的节点后验概率, 但贝叶斯树的拓扑结构明显优于最大似然树; 对于属阶元分支的节点支持率, 两种树基本保持一致。总的来说, 支持率较低或不一致的节点出现在大枝系节点, 多为属间或亚科之间关系的节点, 同样的问题也出现在线粒体基因构建的进化树(刘君等, 2011; Liu et al, 2018), 这种现象可能是由于贻贝早期辐射进化造成的。通过增加物种数目和多基因联合分析, 提供物种之间完整的演化信息, 会改善深部节点的支持率(Liu et al, 2018)。
3.2 我国贻贝系统分类存在的问题从系统发育分析结果来看, 贻贝在亚科阶元的分类存在诸多问题, 需要获得充足的物种样本以进行亚科阶元的研究。
在我国目前的贻贝分类系统中, 条纹隔贻贝属名依然采用属名Septifer。但在国际上Trovant等(2015)和Gerdol等(2017)已经根据分子数据将条纹隔贻贝和Septifer bifurcata划入Mytilisepta属, 并更名为Mytilisepta virgate Wiegmann, 1837和Mytilisepta bifurcata Conrad, 1837, 这一划分方法与本研究所得结论一致:通过系统发育进化树我们发现Mytilisepta属和Septifer属均为单系群, 且分支节点支持率较高; 此外, 通过贻贝科物种两两之间的遗传距离图可知, Mytilisepta属物种与Septifer属隆起隔贻贝和隔贻贝之间的平均遗传距离为0.114, 大于属内种间平均遗传距离。本研究所得系统发育关系与属内种间遗传距离均支持Mytilisepta属的有效性。此外, 从系统发育树我们发现Mytilisepta属与隔贻贝属Septifer的物种聚集在同一分支且表现为姊妹群, 亲缘关系较近, 目前隔贻贝亚科Septiferinae的有效性得到支持。
其次, 在我国目前的分类系统中, 膜石蛏属Leiosolenus为石蛏属Lithophaga的亚属。但国际上Owada(2007)根据分子系统发育结果将Leiosolenus提升到属的地位, 并将两属归为同一亚科Lithophaginae(Carter et al, 2011; Morton, 2015)。本研究中, 根据遗传距离(图 2)可知, Lithophaga属和Leiosolenus属之间的平均遗传距离为0.07, 小于属间平均遗传距离(0.115), 亲缘关系较近; 由系统发育进化树可知, Leiosolenus属与Lithophaga属分别为单系群但两属物种并未聚为一支, 石蛏亚科的单系性未获得系统发育关系的支持, 这与Owada(2007)研究结果一致。此外, 石蛏属Lithophaga与肠蛤属在系统发育树上聚集在同一分支, 遗传距离图显示石蛏属与肠蛤属之间的平均遗传距离为0.094, 小于属间平均遗传距离; 而膜石蛏属Leiosolenus则与偏顶蛤亚科物种亲缘关系较近, 但支持率均不高, 因此石蛏亚科的有效性需要获得更全面的物种以进一步验证。
在本次研究中, 刻缘短齿蛤并未与短齿蛤属其他物种聚集在一起, 而是与贻贝属的物种聚集在同一分支, 支持率较高; 通过遗传距离图可知, 刻缘短齿蛤与短齿蛤属其他物种之间的平均遗传距离远远大于属内种间平均遗传距离, 而它与贻贝属物种之间的平均遗传距离为0.087, 同样大于属内平均遗传距离(0.034), 故也不应归为贻贝属。刻缘短齿蛤Brachidontes setiger Dunker, 1857最初被命名为Volsella setigera Dunker, 1857, 因此我们建议恢复刻缘短齿蛤属名Volsella Dunker, 1857。
4 结论本次研究涉及贻贝科6亚科12属28种, 获得的系统发育关系在亚科阶元的节点支持率较低, 提示我们需要进一步获得更多样品进行系统发育关系构建。本次研究中只有深海贻贝亚科Bathymodiolinae和隔贻贝亚科Septiferinae为单系群, 贻贝亚科Mytilinae和偏顶蛤亚科Modiolinae, 以及石蛏亚科Lithophaginae的单系性不被支持。在属阶元, 深海偏顶蛤属Bathymodiolus 、贻贝属Mytilus和股贻贝属Perna为单系群。隔贻贝属(王祯瑞, 1997)的条纹隔贻贝Mytilisepta virgata和Mytilisepta bifurcata与隔贻贝属其余物种遗传距离较大, 建议接受将Mytilisepta属从原隔贻贝属Septifer划分出来。原石蛏属Lithophaga中的膜石蛏亚属Leiosolenus在系统发育树中并未与石蛏属其余物种聚集, 建议将膜石蛏亚属提升至属的地位。此外, 系统发育关系和遗传距离结果表明, 刻缘短齿蛤Brachidontes setiger与短齿蛤属其他物种亲缘关系较远, 因此建议恢复刻缘短齿蛤属名Volsella Dunker, 1857。对于我国常见贻贝的初步分析显示, 我国贻贝的种类组成需要进一步明确, 分类系统亟需重新修订, 以便为贻贝物种资源的开发利用与保护提供支持。
王兴霞, 2018.基于COI和18S rDNA基因序列的长腹剑水蚤属系统进化分析.上海: 上海海洋大学硕士学位论文 http://cdmd.cnki.com.cn/Article/CDMD-10264-1018849182.htm
|
王祯瑞. 1997. 中国动物志.软体动物门.双壳纲.贻贝目. 北京: 科学出版社
|
毛阳丽, 2011.贻贝属的系统发育及群体的形态学和遗传学研究.青岛: 中国海洋大学硕士学位论文
|
甘志彬, 2014.长臂虾科(甲壳动物亚门: 十足目: 真虾下目: 长臂虾总科)分子系统学研究.青岛: 中国科学院研究生院(海洋研究所)博士学位论文
|
申洁, 2016.中国瘿蜂科部分属种DNA条形码及系统发育研究.杭州: 浙江农林大学硕士学位论文 http://cdmd.cnki.com.cn/Article/CDMD-10341-1016282709.htm
|
朱明, 孟学平, 赵军, 等. 2015. 基于cox1基因的贻贝科遗传差异及系统发育关系. 江苏农业科学, 43(10): 23-27 |
刘君, 2012.双壳贝类DNA分类: 贻贝科和牡蛎科DNA条形码及栉江珧隐存种研究.青岛: 中国海洋大学博士学位论文
|
刘君, 李琪, 孔令锋, 等. 2011. 基于线粒体COⅠ的DNA条形码技术在贻贝科种类鉴定中的应用. 水生生物学报, 35(5): 874-881 |
杨文, 蔡英亚, 邝雪梅. 2013. 中国南海经济贝类原色图谱. 北京: 中国农业出版社
|
沈玉帮, 李家乐, 牟月军. 2009. 基于16S rRNA序列初步探讨贻贝属的系统发育. 海洋科学, 33(12): 50-55 |
张小静, 郑哲民, 赵玲, 等. 2012. 基于28SrDNA的蝗总科部分种类的分子系统发育研究. 陕西师范大学学报(自然科学版), 40(2): 67-70, 75 DOI:10.3969/j.issn.1672-4291.2012.02.015 |
赵庆, 吴彪, 刘志鸿, 等. 2018. 利用DNA条形码COI基因分析我国重要贝类系统进化关系. 中国水产科学, 25(4): 847-857 |
袁剑波, 2014.凡纳滨对虾系统发生地位及适应性进化分析.青岛: 中国科学院研究生院(海洋研究所)博士学位论文
|
徐凤山, 张素萍. 2008. 中国海产双壳类图志. 北京: 科学出版社
|
Bieler R, Carter J G, Coan E V, 2010. Classification of bivalve families. Malacologia, 52(2): 113-133 |
Carter J G, Altaba C R, Anderson L C et al, 2011. A synoptical classification of the Bivalvia (Mollusca). Paleontological Contributions, 4: 1-47 |
Dietrich C H, Rakitov R A, Holmes J L et al, 2001. Phylogeny of the major lineages of Membracoidea (Insecta:Hemiptera:Cicadomorpha) based on 28S rDNA sequences. Molecular Phylogenetics and Evolution, 18(2): 293-305 DOI:10.1006/mpev.2000.0873 |
Gerdol M, Fujii Y, Hasan I et al, 2017. The purplish bifurcate mussel Mytilisepta virgata gene expression atlas reveals a remarkable tissue functional specialization. BMC Genomics, 18: 590 DOI:10.1186/s12864-017-4012-z |
Guindon S, Dufayard J F, Lefort V et al, 2010. New algorithms and methods to estimate maximum-likelihood phylogenies:assessing the performance of PhyML 3.0. Systematic Biology, 59(3): 307-321 DOI:10.1093/sysbio/syq010 |
Hebert P D N, Cywinska A, Ball S L et al, 2003. Biological Identifications through DNA Barcodes. Proceedings of the Royal Society B:Biological Sciences, 270(1512): 313-321 DOI:10.1098/rspb.2002.2218 |
Kalyaanamoorthy S, Minh B Q, Wong T K F et al, 2017. ModelFinder:fast model selection for accurate phylogenetic estimates. Nature Methods, 14(6): 587-589 DOI:10.1038/nmeth.4285 |
Katoh K, Standley D M, 2013. MAFFT multiple sequence alignment software version 7:improvements in performance and usability. Molecular Biology and Evolution, 30(4): 772-780 DOI:10.1093/molbev/mst010 |
Kumar S, Stecher G, Li M et al, 2018. MEGA X:molecular evolutionary genetics analysis across computing platforms. Molecular Biology and Evolution, 35(6): 1547-1549 DOI:10.1093/molbev/msy096 |
Lee T, Foighil D Ó, 2004. Hidden Floridian biodiversity:mitochondrial and nuclear gene trees reveal four cryptic species within the scorched mussel, Brachidontes exustus, species complex. Molecular Ecology, 13(11): 3527-3542 DOI:10.1111/j.1365-294X.2004.02337.x |
Liu J, Liu H L, Zhang H B, 2018. Phylogeny and evolutionary radiation of the marine mussels (Bivalvia:Mytilidae) based on mitochondrial and nuclear genes. Molecular Phylogenetics and Evolution, 126: 233-240 DOI:10.1016/j.ympev.2018.04.019 |
Morton B, 2015. Evolution and adaptive radiation in the Mytiloidea (Bivalvia):clues from the pericardial-posterior byssal retractor musculature complex. Molluscan Research, 35(4): 227-245 DOI:10.1080/13235818.2015.1053167 |
Newell N D, 1969. Classification of bivalvia. In: Treatise on Invertebrate Paleontology. The Geological Society of America and The University of Kansas, (1): 205-224
|
Nguyen L T, Schmidt H A, von Haeseler A et al, 2015. IQ-TREE:a fast and effective stochastic algorithm for estimating maximum-likelihood phylogenies. Molecular Biology and Evolution, 32(1): 268-274 |
Owada M, 2007. Functional morphology and phylogeny of the rock-boring bivalves Leiosolenus and Lithophaga (Bivalvia:Mytilidae):a third functional clade. Marine Biology, 150(5): 853-860 DOI:10.1007/s00227-006-0409-y |
Ronquist F, Teslenko M, van der Mark P et al, 2012. MrBayes 3.2:efficient Bayesian phylogenetic inference and model choice across a large model space. Systematic Biology, 61(3): 539-542 DOI:10.1093/sysbio/sys029 |
Thompson J D, Higgins D G, Gibson T J, 1994. CLUSTAL W:improving the sensitivity of progressive multiple sequence alignment through sequence weighting, position-specific gap penalties and weight matrix choice. Nucleic Acids Research, 22(22): 4673-4680 DOI:10.1093/nar/22.22.4673 |
Trovant B, Orensanz J M, Ruzzante D E et al, 2015. Scorched mussels (BIVALVIA:MYTILIDAE:BRACHIDONTINAE) from the temperate coasts of South America:Phylogenetic relationships, trans-Pacific connections and the footprints of Quaternary glaciations. Molecular Phylogenetics and Evolution, 82: 60-74 |
Wood A R, Apte S, MacAvoy E S et al, 2007. A molecular phylogeny of the marine mussel genus Perna (Bivalvia:Mytilidae) based on nuclear (ITS1&2) and mitochondrial (COI) DNA sequences. Molecular Phylogenetics and Evolution, 44(2): 685-698 DOI:10.1016/j.ympev.2006.12.019 |
Zhang D, Gao F L, Jakovlić I et al, 2020. PhyloSuite:an integrated and scalable desktop platform for streamlined molecular sequence data management and evolutionary phylogenetics studies. Molecular Ecology Resources, 20(1): 348-355 DOI:10.1111/1755-0998.13096 |
 2020, Vol. 51
2020, Vol. 51


