中国海洋湖沼学会主办。
文章信息
- 吴仁协, 张浩冉, 牛素芳, 苗奔奔, 翟云. 2019.
- WU Ren-Xie, ZHANG Hao-Ran, NIU Su-Fang, MIAO Ben-Ben, ZHAI Yun. 2019.
- 东海近岸带鱼(Trichiurus japonicus)线粒体控制区序列的群体遗传变异研究
- STUDY ON POPULATION GENETIC VARIATION OF TRICHIURUS JAPONICUS IN NEARSHORE OF THE EAST CHINA SEA IN MITOCHONDRIAL CONTROL REGION SEQUENCES
- 海洋与湖沼, 50(6): 1318-1327
- Oceanologia et Limnologia Sinica, 50(6): 1318-1327.
- http://dx.doi.org/10.11693/hyhz20190400077
文章历史
-
收稿日期:2019-04-21
收修改稿日期:2019-07-08
带鱼(Trichiurus japonicus)隶属鲈形目Perciformes、带鱼科Trichiuridae、带鱼属Trichiurus, 为暖温性近底层重要经济鱼类, 广泛分布于东亚大陆架海域, 包括中国沿海、朝鲜半岛西部及南部沿海以及日本北海道以南沿海(吴仁协等, 2018)。带鱼是我国海洋四大渔业之一, 也是东海区最重要的海洋渔业捕捞对象。东海区的带鱼渔获量自2000年(90.99×104 t)达到顶峰后, 一直呈波动下降趋势, 近10年渔获量维持在70×104 t左右(农业部渔业渔政管理局, 2009-2018; 张魁等, 2015), 但仍是东海区资源量最大的鱼种。随着捕捞压力加大和近海渔业生态环境的不断恶化, 东海区的带鱼在年龄与生长、繁殖和发育、种群组成、食性等方面均出现了一系列的变化(林新濯, 1987; 罗秉征, 1991), 包括生殖期间对外界环境适应能力提高、产卵场范围扩大等(徐兆礼等, 2015), 其种群生物学趋向低龄化、小型化、性早熟及简单化(张其永等, 2017), 资源已处于衰退状态。目前虽有一些报道涉及东海区的带鱼群体遗传背景研究, 但这些研究较为零散, 且在采样范围、样品数量或是标记灵敏度等方面存在明显不足(王可玲等, 1994; 杨天燕等, 2007; He et al, 2014; Xiao et al, 2014; 郑文娟等, 2015), 其研究结果还难以全面和深入认识东海区的带鱼群体遗传变异和种群结构特征, 不利于其种群的遗传资源保护和管理。
有关东海近海的带鱼洄游分布和种群划分问题, 已有学者进行了广泛的研究和探讨。研究表明, 东海区的带鱼具有沿东海大陆边缘作南北移动的长距离洄游规律(朱元鼎, 1959), 其越冬场主要位于30°N以南的浙江中南部外海水深60—100m海域, 越冬期1—3月(郑元甲等, 2003); 其产卵场分布广阔, 遍及东海近海海域, 生殖期从3月一直延续至10月(盛期为5—7月)(徐兆礼等, 2015)。同时, 东海区的带鱼还与黄渤海带鱼和粤东带鱼存在一定程度的混栖(张其永等, 1966; 徐兆礼等, 2015)。根据洄游分布、渔期资料、形态测量等资料, 多数学者认为东海近海带鱼为一个地方种群(朱元鼎, 1959; 林新濯等, 1965; 张其永等, 1966; 郑元甲等, 2003; 徐兆礼等, 2016), 但也有部分学者将东海区的带鱼划分为东海北部群和东海南部群(罗秉征等, 1981; 卢继武等, 1983)。在群体遗传学方面, 王可玲等(1994)分析了中国近海5个带鱼群体的同工酶遗传分化, 得出舟山海域群体和石狮近海群体为同一个种群的结论, 但所分析的东海带鱼群体数量较少, 难以为种群鉴别提供确切的遗传学证据。同样, He等(2014)基于线粒体Cyt b序列研究了东海带鱼11个群体的分子系统地理学和群体历史动态, 但所用标记的分辨力较低, 难以有效揭示群体遗传结构和遗传多样性水平。因此, 有必要采用高分辨力的分子标记和扩大采样范围来进一步研究东海近海的带鱼群体遗传变异特征和种群划分问题。
线粒体控制区序列因进化速率较快, 具有较高的种内多态性和分辨力, 且序列中多态的数量和分布模式可以为推测群体历史动态提供有效的遗传信息(Liu et al, 2006a, b), 已成为鱼类群体水平遗传变异研究的理想分子标记(Xiao et al, 2014; 郑文娟等, 2015)。本研究利用线粒体控制区序列对东海近岸6个带鱼群体的遗传变异和历史动态进行分析, 从种群历史和现代基因流作用两个方面来揭示群体遗传结构和遗传多样性水平, 阐明东海近岸带鱼群体现有系统地理格局及其形成机制, 以期全面认识东海近岸带鱼群体遗传变异特征和评估其种质资源状况, 为今后制定合理的渔业保护和管理策略提供科学依据。
1 材料与方法 1.1 样品采集实验所用的191尾带鱼于2008年7月至2018年4月采自东海近岸6个海域, 包括上海市崇明岛近岸(CM)、浙江省嵊泗列岛近岸(SS)、浙江省宁波近岸(NB)、浙江省台州近岸(TZ)、福建省平潭近岸(PT)和福建省东山岛近岸(DS)(表 1)。鱼体经形态学鉴定后取其背部肌肉浸入含有95%酒精溶液的2mL离心管中, 于–20℃条件下保存。
| 采样点(群体简称) | 采样时间 | 样品数量 | 单倍型数量 | 多态位点数 | 单倍型多样性 | 核苷酸多样性 | 平均核苷酸差异数 |
| 上海市崇明岛近岸(CM) | 2010.04 | 32 | 28 | 36 | 0.9919±0.0099 | 0.0086±0.0048 | 4.9252±2.4625 |
| 浙江省嵊泗列岛近岸(SS) | 2008.07 | 35 | 33 | 43 | 0.9966±0.0078 | 0.0089±0.0049 | 5.1782±2.5683 |
| 浙江省宁波近岸(NB) | 2016.04 | 50 | 39 | 47 | 0.9861±0.0077 | 0.0095±0.0051 | 5.4588±2.6722 |
| 浙江省台州近岸(TZ) | 2018.04 | 31 | 26 | 43 | 0.9839±0.0160 | 0.0097±0.0053 | 5.5826±2.7576 |
| 福建省平潭近岸(PT) | 2014.09 | 20 | 17 | 29 | 0.9789±0.0245 | 0.0103±0.0057 | 5.9125±2.9460 |
| 福建省东山岛近岸(DS) | 2013.07 | 23 | 21 | 28 | 0.9921±0.0154 | 0.0084±0.0047 | 4.8376±2.4496 |
| 合计 | 191 | 121 | 70 | 0.9911±0.0020 | 0.0092±0.0050 | 5.3357±2.5863 |
采用传统的苯酚-氯仿法提取样品基因组DNA。根据带鱼线粒体DNA基因组全序列(GenBank登录号: EU339148, 下同), 用Primer Premier5软件设计一对引物CR-F553 (5′-ATACCAGGACTCAACATC-3′)和CR-Phe (5′-CGGGCCTATCTTACCATCTT-3′)扩增本研究的带鱼线粒体控制区部分序列。PCR的反应体系为50µL, 包括5µL 10×反应缓冲液(含Mg2+), 正反引物(10µmol/L)各1µL, 1µL dNTPs (10mmol/L), 0.5µL Taq DNA聚合酶(2.5U/µL) (北京全式金生物技术有限公司), 2—3µL DNA模板(约50ng/µL), ddH2O补齐总体积。PCR扩增在Veriti96 Well热循环仪上进行, 反应程序为: 94℃预变性5min; 94℃变性45s, 54℃退火45s, 72℃延伸45s, 循环35次; 最后再72℃延伸7min。PCR产物用1.2% TBE琼脂糖凝胶电泳检测, 检测合格的PCR产物送至广州天一辉远生物科技有限公司进行测序, 测序引物同PCR反应。
1.3 数据分析对所得序列用Dnastar Lasergene v7.1软件包(Burland, 2000)进行编辑、校对和比对, 并以DnaSP 5.10软件(Librado et al, 2009)确定单倍型。采用Arlequin Ver 3.5.1.2软件(Excoffier et al, 2010)计算单倍型数目、多态位点、转换、颠换、插入/缺失等序列多态性参数, 以及单倍型多样性(h)、核苷酸多样性(π)、两两序列间的平均核苷酸差异数(k)等遗传多样性指数。采用Network 5.0.0.1软件(Bandelt et al, 1999)构建基于中介邻接法(Median-joning)的单倍型网络关系图, 以分析单倍型的谱系结构。
在Arlequin Ver 3.5.1.2软件中, 采用分化固定指数Fst评估两两群体间的遗传分化程度, 并以Exact检验检测单倍型在群体间分布频率的差异。群体间的基因交流值由DnaSP 5.10软件估算。通过设定3种分子变异分析(AMOVA)方式来评估群体遗传结构。一是将6个群体都归为一个组群以检验群体间是否存在显著的遗传分化。二是将6个群体划分为东海北部组群(CM、SS、NB、TZ)和东海南部组群(PT、DS)以检验是否存在显著的地理结构。三是考虑到东海近岸江河入海径流(如长江口、钱塘江口、瓯江口、闽江口)可能对其近海动物线粒体DNA谱系结构和群体间基因交流产生影响(阎雪岚等, 2009), 将本研究的6个群体划分为3个组群(CM; SS、NB、TZ; PT、DS)以检测东海近岸径流是否对带鱼群体间的基因交流形成地理屏障。AMOVA运算由Arlequin Ver 3.5.1.2软件运行, 单倍型间的遗传距离采用由jModelTest V2.1.10软件(Darriba et al, 2012)运算出的Kimura双参数模型, 其余参数为软件设置的默认参数。
群体中性检验、核苷酸不配对分布分析和群体扩张时间计算见吴仁协等(2009)报道, 由Arlequin Ver 3.5.1.2软件运算。根据线粒体DNA的种群统计公式θ = 2Nef u(Liu et al, 2006b)估算出总群体的母系有效群体(Nef )大小, 其中θ的估计值即是Arlequin Ver 3.5.1.2软件运算出的θs和θл数值。参照Xiao等(2014)报道的带鱼线粒体控制区序列4%每百万年的突变速率来估算本研究群体发生扩张的时间和有效母系群体大小。运用Beast1.7.5 (Drummond et al, 2007)中贝叶斯天际线(Bayesian skyline plots, BSP)来检测群体历史和有效群体大小变化。基于最适进化模型和严格的分子钟(控制区序列分歧速率为4%—10%每百万年), 运算3×108迭代并用Log Combiner合并运行, 用Tracer1.5软件读取结果, 以获取检测Beast模拟数值(ESS)大于200的BSP分析结果。
2 结果与分析去掉两端引物和tRNAPhe序列后, 所测定的191条带鱼线粒体控制区序列长度为576—578bp, 其A、T、C、G平均含量分别为26.67%、31.34%、22.63%、19.37%, A+T含量(58%)高于C+G含量(42%), 表现出明显的T偏倚和反G偏倚。在比对的577 bp控制区序列内共检测到70个多态位点, 其中有47个是简约信息位点。这些多态位点共定义了73处核苷酸替换, 包括63处替换和10处颠换, 同时检测到6处插入/缺失。在191个个体中, 共检测到121个单倍型(MN031014—MN031134), 单倍型间的核苷酸差异数范围为1—16。
表 1显示, 6个群体的单倍型多样性(h)都很高且相差无几(0.9789—0.9966);核苷酸多样性(π)以平潭近岸群体最高(0.0103)、东山岛近岸群体最低(0.0084), 但群体间差异不大, 并未显示出明显的地理趋势。所有个体的平均单倍型多样性、核苷酸多样性和核苷酸差异数分别为0.9911、0.0092、5.3357, 其遗传多样性表现出高h、中低等π模式。基于中介邻接法构建的带鱼控制区单倍型网络关系图呈星状结构(图 1), 存在一个主体单倍型(H19)位于网络图的中心, 该单倍型被东海近岸5个群体所共享, 提示东海近岸带鱼经历了群体扩张。同时, 单倍型网络关系图结构比较简单, 未检测到与采样点相对应的类群, 表明东海近岸带鱼控制区序列不存在明显的地理谱系结构。
 |
| 图 1 东海近岸带鱼线粒体控制区序列单倍型的中介邻接网络关系图(圆圈面积与单倍型频率成正比) Fig. 1 The median-joining network of haplotypes in T. japonicus mitochondrial control region from nearshore of the East China Sea (the sizes of circles are proportional to haplotype frequency) |
两两群体间的Fst值均较低(–0.0135—0.0212), 且统计检验均不显著(P=0.080—0.833 > 0.05) (表 2), 表明6个群体具有高度的遗传同质性。部分群体间的Fst值为负值, 说明部分群体内个体间的遗传差异大于群体间的遗传差异水平。Exact检验分析显示, 单倍型在群体间的分布频率差异并不显著(P=0.071— 0.812 > 0.05), 可认为东海近岸带鱼6个群体间是随机交配的。同时, 群体间的基因交流值为23.1至无穷大(表 2), 提示群体间存在非常广泛的基因交流。表 3的三种AMOVA分析结果均显示东海近岸带鱼的遗传变异绝大部分来自群体内部(占总变异的99.37%— 99.37%), 来自群体间的遗传变异仅占极少量(0.12%—0.43%), 并且统计检验均不显著(P=0.202—0.204 > 0.05)。此外, AMOVA分析结果不支持根据取样范围将6个群体划分为东海北部和南部2个组群(Fct=0.0035, P=0.276), 也不支持东海沿岸径流对带鱼群体的遗传分化产生明显作用的假设(Fct=0.0045, P=0.261)。
| 群体 | CM | SS | NB | TZ | PT | DS |
| CM | ∞ | 41.2 | 32.2 | 30.6 | ∞ | |
| SS | –0.0069 | ∞ | 46.7 | 23.1 | ∞ | |
| NB | 0.0120 | –0.0033 | 50.5 | 68.9 | ∞ | |
| TZ | 0.0153 | 0.0106 | 0.0098 | 81.5 | ∞ | |
| PT | 0.0161 | 0.0212 | 0.0072 | 0.0061 | ∞ | |
| DS | –0.0134 | –0.0039 | –0.0028 | –0.0006 | –0.0135 |
| 变异来源 | 自由度 | 方差总和 | 变异组成 | 变异百分比 | 分化固定指数 | P值 |
| 1个组群(CM、SS、NB、TZ、PT、DS) | ||||||
| 群体间 | 5 | 15.074 | 0.0115 | 0.43 | Fst=0.0043 | 0.204 |
| 群体内 | 184 | 489.136 | 2.6583 | 99.57 | ||
| 2个组群(CM、SS、NB、TZ)(PT、DS) | ||||||
| 组群间 | 1 | 3.476 | 0.0095 | 0.35 | Fct=0.0035 | 0.276 |
| 组群内群体间 | 4 | 11.598 | 0.0074 | 0.28 | Fsc=0.0028 | 0.287 |
| 群体内 | 184 | 489.136 | 2.6584 | 99.37 | Fst=0.00637 | 0.203 |
| 3个组群(CM)(SS、NB、TZ)(PT、DS) | ||||||
| 组群间 | 2 | 6.780 | 0.0120 | 0.45 | Fct=0.0045 | 0.261 |
| 组群内群体间 | 3 | 8.294 | 0.0033 | 0.12 | Fsc=0.0013 | 0.395 |
| 群体内 | 184 | 489.136 | 2.6584 | 99.43 | Fst=0.0057 | 0.202 |
由于群体间遗传分化不显著, 将6个群体合并分析群体的历史动态。中性检验结果表明, Tajima’s D检验(D=–1.689, P=0.012 < 0.05)和Fu’s FS检验(FS=–25.073, P=0.000 < 0.05)均为显著的负值, 提示东海近岸带鱼可能经历了群体扩张事件。图 2显示, 群体数量扩张和空间扩张的核苷酸不配对分布均呈单峰分布, 与群体扩张模型下的预期分布非常吻合。拟合优度检验也显示(表 4), 数量扩张和空间扩张的SSD值(均为0.001)和Hri指数(均为0.013)均较小, 且统计检验不显著(P=0.166—0.366 > 0.05), 表明所观测的两种核苷酸不配对分布均未显著偏离扩张模型, 揭示了东海近岸带鱼在历史上可能发生过群体数量增长和栖息地扩大事件。根据群体数量和空间扩张模型所估算的参数τ 95%置信区间值分别为4.21—6.22和4.22—6.06(表 4), 推算出带鱼群体发生扩张时间约在13.5—9万年前(晚更新世)。群体数量扩张后、前的θ1/θ0比值为无穷大(表 4), 提示东海近岸带鱼在晚更新世发生群体扩张后其有效群体大小急剧增大。BSP分析也表明(图 3), 约11.5万年前至今, 带鱼有效群体大小一直呈快速增长(约增大了48倍); 而约在30—13万年前期间带鱼有效群体大小却逐渐下降(约下降了43%)。根据所得θs值(11.16)和θл值(5.41), 推算出东海近岸带鱼现存的有效母系群体大小约为0.67×107—1.40×108, 这与BSP分析估算的有效群体大小在107数量级相接近。
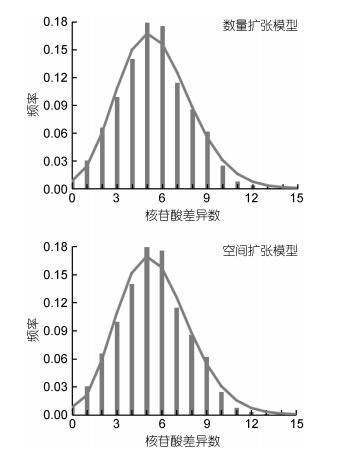 |
| 图 2 东海近岸带鱼线粒体控制区单倍型的核苷酸不配对分布, 柱状图为观测值, 曲线为群体扩张模型下的预期分布 Fig. 2 The observed pairwise differences (bars) and the expected mismatch distribution under the population expansion model (solid line) for the mitochondrial control region haplotypes of T. japonicus from nearshore of the East China Sea |
| 样品 | 数量扩张参数 | 拟合优度检验 | 空间扩张参数 | 拟合优度检验 | ||||||||||
| τ (95%CI) | θ0 | θ1 | SSD | PSSD | Hri | PHri | τ (95%CI) | θ | M | SSD | PSSD | Hri | PHri | |
| 合计 | 4.21—6.22 | 0.000 | 228.1 | 0.001 | 0.207 | 0.013 | 0.350 | 4.22—6.06 | 0.024 | 240.7 | 0.001 | 0.166 | 0.013 | 0.366 |
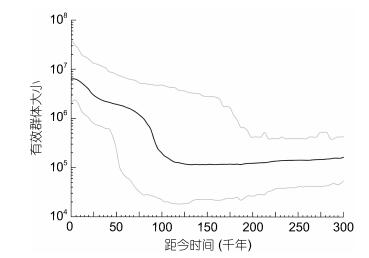 |
| 图 3 东海近岸带鱼群体的贝叶斯天际线分析 Fig. 3 The Bayesian skyline plots (BSP) analysis of T. japonicus populations from nearshore of the East China Sea |
本研究的Fst值和AMOVA分析均显示东海近岸带鱼6个群体间的遗传分化不显著, Exact检验分析也提示群体间可随机交配产生基因流, 单倍型遗传关系分析表明群体不存在明显的地理谱系结构。这种缺乏显著的系统地理种群结构与种群历史进程和现代基因流的作用有较大关系。更新世全球气候周期性波动引起海平面剧变而导致边缘海海洋生物的分布范围和生物量都发生了巨大的变化(Hewitt, 2000; Lambeck et al, 2002)。这种与气候波动相关的种群历史进程必然极大地影响了现有海洋生物的种内系统地理格局(Avise, 2000; Liu et al, 2006a, b; He et al, 2014)。此外, 物种扩散、洄游及繁殖行为、海洋环流以及对环境的适应性等对种群基因流产生影响的进化机制也是形成海洋生物种群遗传结构的一个重要因素(Schulte, 2001; Han et al, 2012)。
本研究的群体历史动态分析结果表明东海近岸带鱼经历了晚更新世的群体扩张事件, 包括群体数量增长和栖息地扩大。单倍型网络关系图也符合群体发生扩张的谱系特征。在更新世, 因每次冰盛期海平面下降(比现在海平面约低110—130m)(Waelbroeck et al, 2002), 渤海和黄海完全露出海面成为陆地, 东海缩剩为一狭长的冲绳海槽, 南海则变为袋状海湾, 二者水深颇大(约1000—3000m)、均无陆架(Wang, 1999), 成为中国近海海洋生物的两个冰期避难所(Liu et al, 2007; He et al, 2015)。在冰期海洋环境恶化和沿岸栖息地消失的情况下, 包括带鱼在内的众多海洋动物在中国大陆架区应该都已灭绝, 而在冰期避难所的部分带鱼孑留群体则幸存下来(Liu et al, 2006a; Han et al, 2008; 吴仁协等, 2009, 2012)。同时, 大陆和台湾岛之间所形成的陆桥以及台湾-琉球群岛陆桥阻碍了东海和南海两个冰期避难所的海洋生物的基因交流(He et al, 2015), 从而促进异域群体的遗传分化(Liu et al, 2007; Cheang et al, 2010)。随着冰后期海平面上升和环境好转, 自晚更新世之后, 由于群体扩张, 来自东海和南海冰期避难所的带鱼孑留群体均有可能在东海陆架区发生重新殖化的事件。从线粒体DNA序列遵循严格的母系遗传特征来讲, 若是南海冰期避难所的孑留带鱼群体在东海陆架区发生过重新殖化, 那么本研究应该能检测到不止一个单倍型谱系或是群体间的遗传差异明显。然而, 本研究只检测到一个单倍型谱系, 且群体间的遗传多样性水平差异较小、无明显的地理趋势, 提示目前东海近岸带鱼应该只来自东海冰期避难所的孑留群体在晚更新世后重新殖化的群体。由于群体扩张发生的时间比较晚, 扩散后新建立的群体尚未在迁移和遗传漂变之间取得平衡(Slatkin, 1993; Liu et al, 2006a, 2007), 这可能是东海近岸带鱼群体间遗传差异不明显的主要历史原因。这种因经历了近期的群体扩张而在中国大陆架区缺乏显著的群体遗传变异现象在许多海洋鱼类中已有报道(Ni et al, 2014)。
在东海近海, 主要海流流系包括东海沿岸流、台湾暖流和黑潮。东海沿岸流是由近岸江河入海径流与周围海水混合形成的一股沿岸水, 夏季向北流延伸至长江口附近, 冬季自长江口向南流进入粤东沿岸; 台湾暖流为常年存在的一股高温、高盐水, 流向总趋势是沿闽浙近海北上; 东海黑潮除了流速和流量有季节性变化外, 流向和流幅变动不大, 流轴比较稳定, 主要为东北流向(陈新军等, 2004)。带鱼产浮性卵, 受精卵在水温25—29.5℃、盐度35条件下历经81h 30min孵化(柴学军等, 2015)。虽然目前对带鱼的幼体浮游期缺乏了解, 但研究表明带鱼受精卵在培养水温21℃时的仔鱼期有5天(沙学绅等, 1981)。因此, 带鱼至少一周长的早期发育浮游期可借助东海近海夏季海流向北流的趋势, 进行从南向北的长距离扩散, 有利于东海近岸带鱼群体间产生广泛的基因交流。Slatkin(1987)认为群体的每一个世代只要有一个迁移者能够有效进入另一个群体的繁育群体, 就可以阻止群体间因为遗传漂变或选择作用而产生的遗传分化。本研究估算的群体间基因流最小值为23.1, 如此频繁的基因交流足以促使东海近岸带鱼群体间形成高度的遗传同质性。
带鱼产卵地区主要位于沿岸的河口附近水深7—22m, 在水质呈白浆状的浅海中排卵(朱元鼎, 1959), 这提示沿岸的江河入海径流可能会促进东海近岸带鱼群体间的遗传分化。然而本研究结果并不支持这种假设, 表明东海近岸的江河入海径流并不构成带鱼扩散的有效屏障。这可能是由于带鱼卵和幼体确实具有较强的潜在扩散能力外, 也可能与东海区的带鱼群体长期存在“北带南游”的洄游规律有密切相关。带鱼每年沿东海和台湾海峡大陆边缘作南北移动, 即11—3月上旬自北往南进行越冬洄游, 3—6月自南往北进行生殖洄游(朱元鼎, 1959; 张其永等, 1966)。这种有规律性的集群洄游可促使东海近岸带鱼群体间有充分的基因交流机会, 导致群体遗传的同质化, 形成了一个随机交配的种群。可见, 有利的海洋环流加上较强的浮游期幼体扩散能力以及长期的洄游规律是导致东海近岸带鱼群体间遗传分化不明显的重要现时原因。
基于线粒体控制区序列分析, 本研究的东海近岸带鱼缺乏显著的系统地理种群结构。这与之前的带鱼同工酶标记研究结论相一致, 即舟山海礁群体与石狮梅林群体间遗传距离(D=0.004)以及青岛胶南近海群体与舟山海域群体间遗传距离(D=0.0015)均较小, 没有明显的遗传分化, 分别可视为一个种群(王可玲等, 1994; 杨天燕等, 2007)。同样, Xiao等(2014)报道了温州、珠海、北海近海等3个带鱼群体线粒体控制区序列缺乏显著的谱系结构。本研究和王可玲等(1994)研究从分子水平上确认了朱元鼎(1959)、林新濯等(1965)、张其永等(1966)、郑元甲等(2003)、徐兆礼等(2016)对东海近海带鱼归为一个种群的结论, 不支持罗秉征等(1981)、卢继武等(1983)将东海近海带鱼划分为东海北部群和东海南部群的观点。
3.2 东海近岸带鱼群体遗传多样性水平在本研究中, 东海近岸带鱼群体总的h值虽较高(0.9911), 但群体总的π值较低(π=0.0092)。由于h指数仅考虑单倍型的数目和频率, π指数还包括单倍型间的突变差异(Arnason et al, 1996)。特别是在h接近1的情况下, 该指数不能有效提供群体遗传变异信息, 而π指数更适用于评判群体的遗传多样性水平(Liu et al, 2006a)。因此, 从π指数来看, 东海近岸带鱼群体遗传多样性总体处于中低等水平。这种较低的核苷酸多样性在温州、珠海、北海等3个近海带鱼群体(π=0.008)和舟山海域带鱼群体(π=0.012)的线粒体控制区序列变异分析中均有相同报道(Xiao et al, 2014; 郑文娟等, 2015)。同样, 基于9种同工酶分析也显示青岛胶南近海和舟山海域2个带鱼群体的多态位点比例(分别为0.1538和0.0769)和平均杂合度(分别为0.0173和0.0037)均较低(杨天燕等, 2007); 随机扩增DNA(RAPD)分析还表明黄海带鱼的多态性比小带鱼(Eupleurogrammus muticus)的低(蒙子宁等, 2003)。然而, 王可玲等(1994)报道的1976—1992年中国近海5个带鱼群体11种同工酶的多态位点比例(0.208— 0.417)和平均杂合度(0.067—0.103)比一般鱼类的高。这些研究结论的差异除了与分析样品的采样年份和所用标记位点不同有关外, 可能还与群体间的遗传分化程度相关。研究表明种群存在显著分化的遗传结构或谱系可导致高水平的核苷酸多样度(Liu et al, 2006a, 2007; He et al, 2010)。在上述研究中, 王可玲等(1994)检测的中国近海5个带鱼群体因遗传分化较大可划分为4个种群, 而其余研究报道的带鱼群体均无明显的谱系分化或是遗传差异。
Frankham(1996)指出群体的遗传多样性与有效群体大小有直接的关系, 只有当群体数量很小时其遗传多样性才会显著下降(Nei et al, 1975)。Ryman等(1995)认为与中等大小群体经历严重瓶颈效应相比, 在大群体降到中等大小群体过程中会丧失更多的稀有等位基因。尤其是当群体因生存压力而导致有效群体大小降低时, 其遗传多样性积累的速度会降低, 而遗传多样性丧失的速度会增加(Frankham et al, 2004)。本研究的BSP分析显示约在30—13万年前的更新世中晚期, 东海近岸带鱼的有效群体大小数量发生了持续性的下降, 约下降了43%, 表明群体可能经历了瓶颈效应。在此期间, 全球因冰期气候变冷大约发生了5次较为明显的海平面下降(Waelbroeck et al, 2002), 这对西北太平洋边缘海的海水温度、海流模式、涌升流密度、海岸栖息地以及海洋生产力都产生了很大影响(Wang, 1999; Lambeck et al, 2002; Liu et al, 2006b; He et al, 2014)。这种周期性的气候剧烈变迁和环境变化可能对东海近岸带鱼群体造成了较大的生存压力, 引起有效群体大小的大幅度下降, 从而导致遗传多样性的丢失。虽然自晚更新世起, 东海近岸带鱼发生群体扩张, 有效群体大小一直呈快速增长(约增大了48倍), 形成目前资源量庞大的东海带鱼种群。此过程可产生许多新的突变, 虽积累了单倍型的多态性, 但因缺乏足够的时间难以积累核苷酸序列的多样化(Grant and Bowen, 1998), 从而导致东海近岸带鱼的群体遗传多样性呈现高h、中低等π的模式。综上可知, 东海近岸带鱼群体在更新世中晚期可能经历了瓶颈效应和随后的群体快速扩张是导致其较低遗传多样性的主要原因。
自20世纪70年代中后期起, 东海区的带鱼一直遭受过度捕捞以致资源目前仍处于衰退状态, 种群生物学特征出现明显的退化现象(张其永等, 2017)。蒙子宁等(2003)认为过度捕捞可能促使黄海带鱼群体定向选择加强而导致其遗传多样性的丢失。类似的解释和推测也见于其他商业捕捞鱼类的群体遗传变异研究中(Allendorf et al, 2008; 牛素芳等, 2011, 2018)。然而, 目前仅在少数海洋鱼类中发现过度开发与其低的群体遗传多样性之间有直接的相关性, 如金赤鲷(Pagrus auratus)(Hauser et al, 2002)、大西洋鳕(Gadus morhua)(Hutchinson et al, 2003)、克氏平鲉(Sebastes crameri)(Gomez-Uchida et al, 2006)等。虽然目前因缺乏东海区的带鱼在过度捕捞前的群体遗传学数据, 难以通过比较渔业开发前后的遗传学数据来直接判断捕捞效应对东海区的带鱼群体遗传多样性的影响程度。但是, 本研究的BSP分析表明自晚更新世以来东海近岸带鱼的有效群体大小呈持续快速增长态势, 现存有效群体大小数量庞大。He等(2014)基于线粒体Cyt b序列分析结果认为捕捞压力并未对南海北部的带鱼种群动态产生重要影响。基于微卫星DNA标记研究也表明宁波近海带鱼群体未检测到近期的遗传瓶颈效应(张浩冉等, 2019)。据此, 我们认为近40来年的过度捕捞对东海区的带鱼资源量和渔获量的波动有明显的影响(王跃中等, 2011), 但对其有效群体大小的影响可能很有限, 当前的过度捕捞并不是导致东海近岸带鱼群体遗传多样性较低的主要原因。
4 结论本研究基于线粒体控制区序列的分析结果表明, 更新世气候变迁对东海近岸带鱼群体的遗传变异特征产生了重要影响。更新世中晚期可能经历的瓶颈效应和随后的群体快速扩张是导致东海近岸带鱼群体遗传多样性较低的主要原因。虽然当前的捕捞效应并未明显作用于群体的遗传多样性, 但从长远的进化角度来看, 较低的遗传多样性水平意味着东海近岸带鱼资源延续能力和进化潜力所有下降, 资源的长期可持续利用可能会受到影响, 应当进一步加强对东海近岸带鱼群体遗传资源的有效保护。东海近岸带鱼缺乏显著的系统地理种群结构, 一方面是因为自晚更新世后东海带鱼发生近期的群体扩张所引起的, 另一方面是带鱼浮游期幼体借助海流进行广泛扩散以及群体具有固定洄游行为加强了群体间的基因交流。研究结果提示, 在线粒体DNA水平上, 东海近岸带鱼群体可作为一个遗传资源管理单元进行管理。此外, 考虑到已有研究对中国近海带鱼群体遗传多样性水平的评估结论存在明显不一致, 有必要在后续的研究中采用综合分析手段和分辨力更高的分子标记(如微卫星DNA、SNP标记等)来进一步解析东海及中国近海的带鱼群体遗传结构和重新评估其种质资源状况。
王可玲, 张培军, 刘兰英, 等. 1994. 中国近海带鱼种群生化遗传结构及其鉴别的研究. 海洋学报, 16(1): 93-104 DOI:10.3321/j.issn:0253-4193.1994.01.012 |
王跃中, 贾晓平, 林昭进, 等. 2011. 东海带鱼渔获量对捕捞压力和气候变动的响应. 水产学报, 35(12): 1881-1889 |
牛素芳, 苏永全, 王军, 等. 2011. 福建近海竹荚鱼线粒体DNA控制区和细胞色素b遗传多态性. 中国水产科学, 18(1): 66-74 |
牛素芳, 吴仁协, 张丽艳, 等. 2018. 基于Cyt b基因序列的南海北部蓝圆鲹群体遗传多样性研究. 应用海洋学学报, 37(2): 263-273 DOI:10.3969/J.ISSN.2095-4972.2018.02.014 |
卢继武, 罗秉征, 黄颂芳. 1983. 台湾浅滩海域带鱼种群的探讨. 海洋与湖沼, 14(4): 377-387 |
朱元鼎, 1959.中国主要海洋渔业生物学基础的参考资料.见: 太平洋西部渔业研究委员会第二次全体会议论文集.北京: 科学出版社, 122-127
|
农业部渔业渔政管理局, 2009-2018.2009-2018年中国渔业统计年鉴.北京: 中国农业出版社
|
杨天燕, 高天翔. 2007. 黄海和东海带鱼群体同工酶分析. 海洋水产研究, 28(3): 44-49 |
吴仁协, 张浩冉, 郭刘军, 等. 2018. 中国近海带鱼Trichiurus japonicus的命名和分类学地位研究. 基因组学与应用生物学, 37(9): 3782-3791 |
吴仁协, 柳淑芳, 庄志猛, 等. 2009. 基于线粒体Cyt b基因的黄海、东海小黄鱼(Larimichthys polyactis)群体遗传结构. 自然科学进展, 19(9): 924-930 DOI:10.3321/j.issn:1002-008X.2009.09.005 |
吴仁协, 梁秀何, 庄志猛, 等. 2012. 中国近海银鲳线粒体COI基因序列变异分析. 动物分类学报, 37(3): 480-488 |
沙学绅, 阮洪超, 何桂芬. 1981. 带鱼卵子和仔、稚鱼的形态特征. 水产学报, 5(2): 155-160 |
张魁, 陈作志. 2015. 应用贝叶斯状态空间建模对东海带鱼的资源评估. 中国水产科学, 22(5): 1015-1026 |
张其永, 林双淡, 杨高润. 1966. 我国东南沿海带鱼种群问题的初步研究. 水产学报, 3(2): 106-118 |
张其永, 洪万树, 陈仕玺. 2017. 中国近海大黄鱼和日本带鱼群体数量变动及其资源保护措施探讨. 应用海洋学学报, 36(3): 438-445 DOI:10.3969/J.ISSN.2095-4972.2017.03.018 |
张浩冉, 梁镇邦, 吴仁协, 等. 2019. 利用SLAF-seq技术开发带鱼(Trichiurus japonicus)微卫星标记以及跨物种扩增检测. 基因组学与应用生物学, 38(2): 574-585 |
陈新军, 俞存根, 卢伙胜. 2004. 渔业资源与渔场学. 北京: 海洋出版社, 224-226
|
林新濯. 1987. 中国近海三种主要经济鱼类的生物学特性与资源现状. 水产学报, 11(3): 187-194 |
林新濯, 王福刚, 潘家模, 等. 1965. 中国近海带鱼Trichiurus haumela (Forskål)种族的调查. 水产学报, 2(4): 11-23 |
罗秉征, 1991.带鱼.见: 邓景耀, 赵传絪.海洋渔业生物学.北京: 农业出版社, 111-163
|
罗秉征, 卢继武, 黄颂芳, 1981.中国近海带鱼耳石生长的地理变异与地理种群的初步探讨.见: 海洋与湖沼论文集.北京: 海洋出版社, 181-191
|
郑元甲, 陈雪忠, 程家骅, 等. 2003. 东海大陆架生物资源与环境. 上海: 上海科学技术出版社, 451-453
|
郑文娟, 杜一超, 林洁, 等. 2015. 基于线粒体DNA D-loop区部分序列分析舟山海域带鱼种群遗传结构. 水生生物学报, 39(2): 408-413 |
柴学军, 朱云海, 王跃斌, 等. 2015. 东海带鱼的胚胎发育研究. 浙江海洋学院学报(自然科学版), 34(5): 429-432 DOI:10.3969/j.issn.1008-830X.2015.05.006 |
徐兆礼, 陈佳杰. 2015. 东、黄渤海带鱼的洄游路线. 水产学报, 39(6): 824-835 |
徐兆礼, 陈佳杰. 2016. 再议东黄渤海带鱼种群划分问题. 中国水产科学, 23(5): 1185-1196 |
阎雪岚, 唐文乔, 杨金权. 2009. 基于线粒体控制区的序列变异分析中国东南部沿海凤鲚种群遗传结构. 生物多样性, 17(2): 143-150 |
蒙子宁, 庄志猛, 金显仕, 等. 2003. 黄海带鱼、小带鱼RAPD和线粒体16S rRNA基因序列变异分析. 自然科学进展, 13(11): 1170-1176 DOI:10.3321/j.issn:1002-008X.2003.11.009 |
Allendorf F W, England P R, Luikart G et al, 2008. Genetic effects of harvest on wild animal populations. Trends in Ecology & Evolution, 23(6): 327-337 |
Arnason E, Pálsson S, 1996. Mitochondrial cytochrome b DNA sequence variation of Atlantic cod Gadus morhua, from Norway. Molecular Ecology, 5(6): 715-724 DOI:10.1111/j.1365-294X.1996.tb00368.x |
Avise J C, 2000. Phylogeography:The History and Formation of Species. Cambridge, USA: Harvard University Press, 1-447
|
Bandelt H J, Forster P, Röhl A, 1999. Median-joining networks for inferring intraspecific phylogenies. Molecular Biology and Evolution, 16(1): 37-48 |
Burland T G, 2000. DNASTAR's Lasergene sequence analysis software. In: Misener S, Krawetz S A eds. Bioinformatics Methods and Protocols. Totowa, USA: Humana Press, 71-91
|
Cheang C C, Chu K H, Ang P O Jr, 2010. Phylogeography of the marine macroalga Sargassum hemiphyllum (Phaeophyceae, Heterokontophyta) in northwestern Pacific. Molecular Ecology, 19(14): 2933-2948 DOI:10.1111/j.1365-294X.2010.04685.x |
Darriba D, Taboada G L, Doallo R et al, 2012. jModelTest 2:more models, new heuristics and parallel computing. Nature Methods, 9(8): 772 |
Drummond A J, Rambaut A, 2007. BEAST:Bayesian evolutionary analysis by sampling trees. BMC Evolutionary Biology, 7: 214 DOI:10.1186/1471-2148-7-214 |
Excoffier L, Lischer H E L, 2010. Arlequin suite ver 3.5:a new series of programs to perform population genetics analyses under Linux and Windows. Molecular Ecology Resources, 10(3): 564-567 DOI:10.1111/j.1755-0998.2010.02847.x |
Frankham R, 1996. Relationship of genetic variation to population size in wildlife. Conservation Biology, 10(6): 1500-1508 DOI:10.1046/j.1523-1739.1996.10061500.x |
Frankham R, Ballou J D, Briscoe D A et al, 2004. A Primer of Conservation Genetics. Cambridge, UK: Cambridge University Press, 1-220
|
Gomez-Uchida D, Banks M A, 2006. Estimation of effective population size for the long-lived darkblotched rockfish Sebastes crameri. Journal of Heredity, 97(6): 603-606 DOI:10.1093/jhered/esl042 |
Grant W A S, Bowen B W, 1998. Shallow population histories in deep evolutionary lineages of marine fishes:insights from sardines and anchovies and lessons for conservation. Journal of Heredity, 89(5): 415-426 DOI:10.1093/jhered/89.5.415 |
Han Z Q, Gao T X, Yanagimoto T et al, 2008. Deep phylogeographic break among white croaker Pennahia argentata (Sciaenidae, Perciformes) populations in North-western Pacific. Fisheries Science, 74(4): 770-780 DOI:10.1111/j.1444-2906.2008.01588.x |
Han Z Q, Yanagimoto T, Zhang Y P et al, 2012. Phylogeography study of Ammodytes personatus in northwestern pacific:Pleistocene isolation, temperature and current conducted secondary contact. PLoS One, 7(5): e37425 DOI:10.1371/journal.pone.0037425 |
Hauser L, Adcock G J, Smith P J et al, 2002. Loss of microsatellite diversity and low effective population size in an overexploited population of New Zealand snapper (Pagrus auratus). Proceedings of the National Academy of Sciences of the United States of America, 99(18): 11742-11747 DOI:10.1073/pnas.172242899 |
He L J, Mukai T, Chu K H et al, 2015. Biogeographical role of the Kuroshio Current in the amphibious mudskipper Periophthalmus modestus indicated by mitochondrial DNA data. Scientific Reports, 5: 15645 DOI:10.1038/srep15645 |
He L J, Zhang A B, Weese D et al, 2010. Late Pleistocene population expansion of Scylla paramamosain along the coast of China:a population dynamic response to the Last Interglacial sea level highstand. Journal of Experimental Marine Biology and Ecology, 385(1-2): 20-28 DOI:10.1016/j.jembe.2010.01.019 |
He L J, Zhang A B, Weese D et al, 2014. Demographic response of cutlassfish (Trichiurus japonicus and T. nanhaiensis) to fluctuating palaeo-climate and regional oceanographic conditions in the China seas. Scientific Reports, 4: 6380 |
Hewitt G, 2000. The genetic legacy of the Quaternary ice ages. Nature, 405(6789): 907-913 DOI:10.1038/35016000 |
Hutchinson W F, van Oosterhout C, Rogers S I et al, 2003. Temporal analysis of archived samples indicates marked genetic changes in declining North Sea cod (Gadus morhua). Proceedings of the Royal Society B:Biological Sciences, 270(1529): 2125-2132 DOI:10.1098/rspb.2003.2493 |
Lambeck K, Esat T M, Potter E K, 2002. Links between climate and sea levels for the past three million years. Nature, 419(6903): 199-206 DOI:10.1038/nature01089 |
Librado P, Rozas J, 2009. DnaSP v5:a software for comprehensive analysis of DNA polymorphism data. Bioinformatics, 25(11): 1451-1452 DOI:10.1093/bioinformatics/btp187 |
Liu J X, Gao T X, Wu S F et al, 2007. Pleistocene isolation in the Northwestern Pacific marginal seas and limited dispersal in a marine fish, Chelon haematocheilus (Temminck & Schlegel, 1845). Molecular Ecology, 16(2): 275-288 |
Liu J X, Gao T X, Yokogawa K et al, 2006a. Differential population structuring and demographic history of two closely related fish species, Japanese sea bass (Lateolabrax japonicus) and spotted sea bass (Lateolabrax maculatus) in Northwestern Pacific. Molecular Phylogenetics and Evolution, 39(3): 799-811 DOI:10.1016/j.ympev.2006.01.009 |
Liu J X, Gao T X, Zhuang Z M et al, 2006b. Late Pleistocene divergence and subsequent population expansion of two closely related fish species, Japanese anchovy (Engraulis japonicus) and Australian anchovy (Engraulis australis). Molecular Phylogenetics and Evolution, 40(3): 712-723 DOI:10.1016/j.ympev.2006.04.019 |
Nei M, Maruyama T, Chakraborty R, 1975. The bottleneck effect and genetic variability in populations. Evolution, 29(1): 1-10 |
Ni G, Li Q, Kong L F et al, 2014. Comparative phylogeography in marginal seas of the northwestern Pacific. Molecular Ecology, 23(3): 534-548 DOI:10.1111/mec.12620 |
Ryman N, Utter F, Laikre L, 1995. Protection of intraspecific biodiversity of exploited fishes. Reviews in Fish Biology and Fisheries, 5(4): 417-446 DOI:10.1007/BF01103814 |
Schulte P M, 2001. Environmental adaptations as windows on molecular evolution. Comparative Biochemistry and Physiology Part B:Biochemistry and Molecular Biology, 128(3): 597-611 DOI:10.1016/S1096-4959(00)00357-2 |
Slatkin M, 1987. Gene flow and the geographic structure of natural populations. Science, 236(4803): 787-792 DOI:10.1126/science.3576198 |
Slatkin M, 1993. Isolation by distance in equilibrium and non-equilibrium populations. Evolution, 47(1): 264-279 |
Waelbroeck C, Labeyrie L, Michel E et al, 2002. Sea-level and deep water temperature changes derived from benthic foraminifera isotopic records. Quaternary Science Reviews, 21(1-3): 295-305 DOI:10.1016/S0277-3791(01)00101-9 |
Wang P X, 1999. Response of Western Pacific marginal seas to glacial cycles:paleoceanographic and sedimentological features. Marine Geology, 156(1-4): 5-39 DOI:10.1016/S0025-3227(98)00172-8 |
Xiao Y S, Ren G J, Song N et al, 2014. Genetic diversity and population structure of largehead hairtail, Trichiurus japonicus, based on mtDNA control region. Mitochondrial DNA, 25(6): 473-481 DOI:10.3109/19401736.2013.809427 |
 2019, Vol. 50
2019, Vol. 50

