中国海洋湖沼学会主办。
文章信息
- 李迎, 米铁柱, 乔玲, 甄毓. 2019.
- LI Ying, MI Tie-Zhu, QIAO Ling, ZHEN Yu. 2019.
- 基于转录组测序对两种氮素营养条件下多形微眼藻氮代谢途径的解析
- NITROGEN METABOLISM PATHWAY OF MINUTOCELLUS POLYMORPHUS UNDER TWO NITROGEN NUTRITION CONDITIONS BASED ON TRANSCRIPTOME
- 海洋与湖沼, 50(6): 1241-1251
- Oceanologia et Limnologia Sinica, 50(6): 1241-1251.
- http://dx.doi.org/10.11693/hyhz20190400073
文章历史
-
收稿日期:2019-04-17
收修改稿日期:2019-07-01
2. 青岛海洋科学与技术试点国家实验室 海洋生态与环境科学功能实验室 青岛 266071;
3. 中国海洋大学 环境科学与工程学院 青岛 266100;
4. 中国水产科学研究院黄海水产研究所 青岛 266071
2. Laboratory for Marine Ecology and Environmental Science, Pilot National Laboratory for Marine Science and Technology(Qingdao), Qingdao 266071, China;
3. College of Environmental Science and Engineering, Ocean University of China, Qingdao 266100, China;
4. Yellow Sea Fisheries Research Institute, Chinese Academy of Fishery Sciences, Qingdao 266071, China
氮是海洋浮游生物生长所必需的营养元素之一, 是海洋生物的新陈代谢和生化组成的重要参与成分。海水中氮营养元素的化学形态和浓度直接影响海洋微藻的生长繁殖, 也是引发有害藻华的原因之一(Dham et al, 2005; Taylor et al, 2006)。藻类对不同氮素营养的吸收利用机制不同, 并因分子层面的响应而表现出不同的生理活动和生长状况, 以更好地适应环境(Allen et al, 2006; Taylor et al, 2006)。微藻氮代谢一般是指硝酸盐在硝酸盐还原酶等一系列催化反应下, 还原为氨, 然后在谷氨酸酶和谷氨酰胺酶的作用下最终转化成各种常用的氨基酸, 进而参与蛋白质等大分子的生物合成的过程(Muro-Pastor et al, 2005)。有研究表明, 氮代谢还与藻细胞内油脂含量的变化有密切关系。当环境中氮的浓度和形态发生变化时会导致微藻氮代谢通路改变, 细胞内油脂含量和脂肪酸的组成也随之发生变化(Cha et al, 2011)。
多形微眼藻(Minutocellus polymorphus)隶属于硅藻纲(Bacillariophyceae)、微眼藻属(Minutocellus) (Walsh et al, 1988)。自2009年以来在我国秦皇岛海域频繁暴发并给当地养殖业带来巨大经济损失的抑食金球藻(Aureococcus anophagefferens)褐潮中, 本研究团队发现抑食金球藻暴发过程中伴随着多形微眼藻的大量繁殖(Qiao et al, 2017)。而目前有关多形微眼藻对氮营养盐利用特征的研究非常少, 研究多形微眼藻利用不同氮素营养的氮代谢途径有助于进一步了解该藻与抑食金球藻的相互关系及种群演替过程, 可为进一步研究海洋微藻碳代谢、脂质合成调控等机制研究提供理论基础。
本研究以多形微眼藻为实验对象, 测定了其在两种氮素营养(硝酸钠和尿素)条件下的转录组, 并基于转录组数据构建其氮代谢通路, 分析氮代谢相关酶的编码基因的差异表达, 以期从分子水平阐明该藻对不同氮源的利用机制, 为研究硅藻在不同环境中的适应机制奠定基础。
1 材料与方法 1.1 藻种与培养本实验所用藻种多形微眼藻(M. polymorphus)来自于近海海洋环境科学国家重点实验室(厦门大学)高亚辉教授课题组。培养于室内光照培养箱中, 用f/2培养基进行培养, 盐度为33, pH为7.8—8.0, 培养温度为(20±1)℃, 光暗周期比为12h:12h, 光照条件为80—100μmol/(m2·s)。
1.2 测序样品准备选取硝酸钠和尿素两种氮源进行实验培养。为获得多形微眼藻在每种氮源下的最适生长浓度, 分别设置100、200、400和882μmol/L(以N计, 下同)四个不同浓度梯度的氮源进行预实验, 每日从培养基中均匀取出1mL样品, 加10μL鲁格试剂固定, 待细胞充分沉降后, 用光学显微镜进行细胞计数, 并绘制生长曲线图(图 1), 后选取882μmol/L的硝酸钠和400μmol/L的尿素作为氮源, 进行下一步实验, 其余营养盐条件参照f/2培养基。实验进行前, 将藻细胞转入实验浓度的氮源下, 二次驯化后, 再移入含有对应氮源的改良f/2培养基中。培养体积均为300mL, 每组处理为3个平行样, 置于光照培养箱中进行培养。取指数生长期的藻细胞, 过膜收集后洗脱至离心管中用台式高速冷冻离心机(HealForce Neofuge 15R)离心(4000r/min, 5min), 弃上清液后速冻于液氮中保存。样品收集后送交北京诺禾致源生物信息科技有限公司进行总RNA的提取及Illumina PE 150上机测序。
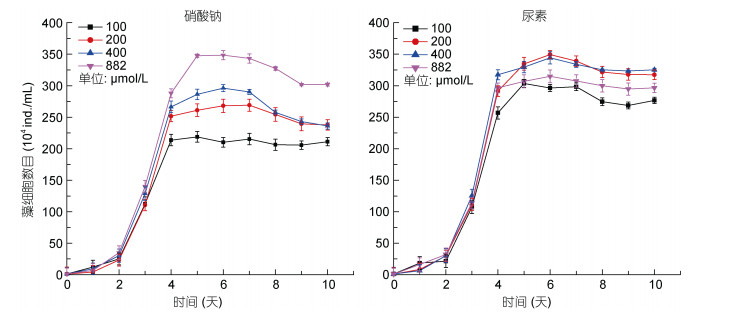 |
| 图 1 多形微眼藻在不同氮素营养条件下的生长曲线 Fig. 1 Growth curve of M. polymorphus in different nitrogen concentrations |
通过CASAVA碱基识别(base calling)分析将高通量测序得到的原始图像数据文件转化为原始测序序列(raw reads), 以FASTQ文件格式保存, 对测序的数据进行错误率分布检查和A/T/G/C含量分布检查以保证后续分析数据的质量。同时对测序得到的原始序列进行过滤, 得到的可用序列(clean reads)用于后续分析。原始数据过滤的步骤包括去除其中含接头的序列、去除N (N表示无法确定碱基信息)的比例大于10%的序列、去除低质量的序列(质量值Qphred≤20的碱基数占整个序列50%以上的序列)三个步骤。通过Trinity (Grabherr et al, 2011)软件对可用序列进行拼接, 获得一组转录本序列, 作为后续分析的参考序列(ref)。
1.4 基因功能注释及代谢途径分析为了得到全面的基因功能信息, 使用NCBI blast 2.2.28+算法(Camacho et al, 2009)和diamond v0.8.22软件在NR、NT、KOG、Swiss-prot四大数据库中对拼接后的转录组进行基因功能注释, 对应的e-value阈值分别是10-5、10-5、10-3、10-5; 通过HMMER3程序对Gene进行Pfam蛋白结构域预测, Pfam(Protein family)将蛋白质的结构域分为不同的蛋白家族, 通过蛋白序列的比对建立每个家族的氨基酸序列的隐马尔科夫(HMM)统计模型; 通过软件Blast2Gov2.5 (Götz et al, 2008)和自写脚本基于NR与PFAM两部分的注释结果进行GO注释(Gene Ontology annotation); 利用KAAS服务器对基因做KO(KEGG Ontology)功能注释和信号通路注释, 根据其参与的KEGG (Kyoto Encyclopedia of Genes and Genomes)代谢通路进行分类分析(Moriya et al, 2007)。
1.5 基因差异表达分析及差异基因KEGG富集采用DESeq2对基因表达水平中的read count数据进行分析, read count值代表比对到某个基因的序列数(Love et al, 2014), 用以区分三个生物学重复之间以及对照组和处理组之间的差异, 并对三个生物学重复的样品进行筛选, 筛选阈值为padj < 0.05且|log2FoldChange| > 1。
KEGG将不同的基因与生物体内的分子作用链接起来构成一个网络图, 从而系统的分析其基因功能(Kanehisa et al, 2008)。通过Pathway显著性富集可以找到不同比较组合的差异基因可能位于哪些代谢通路, 也可以用来确定差异表达基因主要参与的主要信号转导途径, 从而能够快速发现特定生物学现象与引起这种现象基因的关联。
2 结果与讨论 2.1 转录组测序信息及拼接将得到的高质量有效序列进行de novo拼接, 共获得58354个转录本, 43341个非重复序列基因(unigene), 长度范围为200—40000bp, 拼接长度分布如表 1所示。
| 转录长度区间(bp) | 转录本数 | Unigene数 |
| 200—500 | 23255 | 8405 |
| 500—1000 | 9190 | 9034 |
| 1000—2000 | 11295 | 11288 |
| >2000 | 14614 | 14614 |
| 总数 | 58354 | 43341 |
通过对43341个unigene在上述基因库中比对发现, 一共有36109个unigene被注释, 占总unigene的83.31%, 其中同时在所有基因数据库中注释到的unigene共有3186个, 占总unigene数的7.35%。通过在NR库的比对注释, 获得了多形微眼藻的基因功能信息。对多形微眼藻的基因序列与近缘物种基因序列进行相似性分析, 发现与另外两种硅藻假微型海链藻(Thalassiosira pseudonana)和三角褐指藻(Phaeodactylum tricornutum)具有较高的同源性, 其比例分别为24.7%和20.9%;对所有转录本在GO数据库进行比对后, 将注释成功的基因按照基因功能分类系统中生物过程、细胞组分和分子功能三大类的下一层级进行分类, 共获得56个基因功能类别(GO term); 将注释成功的基因按真核生物直系同源关系(KOG, euKaryotic Ortholog Groups)的26个组别进行分类, 可从中获得相关基因所调节的功能信息及不同氮源对生物功能影响的情况。通过基因的功能注释和KEGG代谢途径分析可以得到细胞中生物分子参与的通路信息, 其中能匹配到EC编号的基因转录本有11694条, 将有EC编号的基因产物与相关确定基因组中的基因结合起来, 构建了126条不同的参考代谢通路, 包括氮代谢、谷氨酸代谢和氰基氨基酸代谢等与氮代谢有关的通路。由于本研究涉及多形微眼藻在两种不同氮源条件下的生长特征, 因此我们重点分析了氮代谢途径的变化情况。
2.3 氮代谢途径的解析通过对多形微眼藻转录本的基因功能注释并对其进行氮代谢途径分析, 共发现15种与氮代谢相关的酶, 对应76个编码基因的转录本, 每个酶有1—9个转录本(表 2)。通过BLASTx与NCBI数据库进行对比, 并列出与其具有较高同源性的NCBI登录号。结果表明, 参与氮代谢过程的41个功能基因与已完成基因组测序的假微型海链藻和三角褐指藻具有较高同源性, 未比对到藻类的基因序列用##进行标记。
| 酶(蛋白)名称 | 英文名称(简称) | EC编号 | 基因ID (本转录组中) | 与NCBI数据库比对一致性% (NCBI登录号) |
| 硝酸盐转运蛋白 | nitrate/nitrite transporter (NRT) | - | *Cluster-473.28229 | 71(XP_002288802) |
| Cluster-473.5624 | 60(XP_002287398.1) | |||
| Cluster-473.5623 | 60(XP_002287398.1) | |||
| Cluster-473.21089 | 55(XP_002295904.1) | |||
| Cluster-473.26691 | 62(XP_002295904.1) | |||
| 硝酸盐还原酶 | nitrate reductase (NAD(P)H) (NR) | 1.7.1.1 1.7.1.2 1.7.1.3 | *Cluster-473.3694 | 69(XP_002294410) |
| *Cluster-473.6176 | 70(XP_002183599) | |||
| *Cluster-473.6174 | 70(XP_002183599) | |||
| Cluster-473.6175 | 69(XP_002183599) | |||
| 亚硝酸盐还原酶 | ferredoxin-nitrite reductase (nirA) | 1.7.7.1 | *Cluster-473.12039 | 79(XP_002289265.1) |
| 谷氨酸脱氢酶 | glutamate dehydrogenase (NADP+) (GDH2) | 1.4.1.4 | *Cluster-473.24972 | 67(XP_002289225.1) |
| Cluster-8284.0 | 53(XP_002181513) | |||
| Cluster-473.24657 | 57(XP_002181513) | |||
| Cluster-473.6155 | 60(XP_002297200) | |||
| Cluster-473.24969 | 64(XP_002289225.1) | |||
| Cluster-9792.1 | 52(XP_002181513) | |||
| ##Cluster-7691.1 | 100(WP_012187234.1) | |||
| ##Cluster-9792.0 | 86(PTM05628.1) | |||
| 谷氨酸合成酶 | glutamate synthase (NADPH/NADH) (GLT1) | 1.4.1.13 1.4.1.14 | Cluster-473.18900 | 77(XP_002290024.1) |
| Cluster-473.13314 | 78(XP_002184279) | |||
| Cluster-473.17107 | 77(XP_002180120) | |||
| Cluster-473.17112 | 77(XP_002180120) | |||
| Cluster-473.17111 | 77(XP_002180120) | |||
| 谷氨酸合成酶(铁氧还蛋白) | glutamate synthase (ferredoxin) (GLTD) | 1.4.7.1 | *Cluster-473.23208 | 77(XP_002294462) |
| Cluster-473.23209 | 77(XP_002294462) | |||
| Cluster-473.6754 | 77(XP_002294462) | |||
| 谷氨酰胺合成酶 | glutamine synthetase (GS) | 6.3.1.2 | *Cluster-473.10865 | 78(XP_002295274.1) |
| Cluster-473.27303 | 69(XP_002294945) | |||
| Cluster-473.15350 | 77(XP_002295274.1) | |||
| ##Cluster-473.4799 | 73(PTL97059.1) | |||
| ##Cluster-473.9257 | 93(PWL24251.1) | |||
| 氰酸裂解酶 | cyanate lyase (CynS) | 4.2.1.104 | *Cluster-473.576 | 60(XP_002295609) |
| *Cluster-473.577 | 60(XP_002295609) | |||
| 尿氨酸甲酰胺酶 | formamidase (FM) | 3.5.1.49 | Cluster-473.22192 | 82(XP_002180029) |
| Cluster-473.22193 | 83(XP_002180029.1) | |||
| ##Cluster-11815.0 | 53(WP_095527559.1) | |||
| 精氨琥珀酸合成酶 | argininosuccinate synthase (ASS1) | 6.3.4.5 | Cluster-473.16795 | 80(XP_002185721.1) |
| Cluster-473.13641 | 80(XP_002185721.1) | |||
| Cluster-473.16789 | 80(XP_002185721.1) | |||
| Cluster-473.16794 | 80(XP_002185721.1) | |||
| Cluster-473.16793 | 80(XP_002185721.1) | |||
| ##Cluster-10869.2 | 100(WP_012180129.1) | |||
| ##Cluster-10869.3 | 100(WP_012180129.1) | |||
| ##Cluster-10869.1 | 100(WP_012180129.1) | |||
| ##Cluster-6269.0 | 90(WP_022701197.1) | |||
| 精氨琥珀酸裂解酶 | argininosuccinate lyase (ASL) | 4.3.2.1 | Cluster-473.13551 | 85 (XP_002291698.1) |
| Cluster-473.8701 | 84(XP_002291698.1) | |||
| 脲酶 | Urease (URE) | 3.5.1.5 | Cluster-473.9393 | 71(XP_002296690.1) |
| ##Cluster-5008.0 | 100(WP_012179037.1) | |||
| 氨甲酰转移酶 | ornithine carbamoyltransferase (OTC) | 2.1.3.3 | Cluster-5052.0 | 72(XP_009041368.1) |
| Cluster-473.18958 | 73(XP_009041368.1) | |||
| 谷氨酸脱氢酶 | glutamate dehydrogenase (NAD(P)+) (DGH1) | 1.4.1.3 | ##Cluster-473.15161 | 98(WP_044228175.1) |
| ##Cluster-473.19548 | 99(WP_044228175.1) | |||
| ##Cluster-473.7956 | 99(WP_044228175.1) | |||
| 碳酸酐酶 | carbonic anhydrase (CA) | 4.2.1.1 | ##Cluster-3279.0 | 100(WP_012179896.1) |
| ##Cluster-473.26029 | 94(WP_071691136.1) | |||
| 注: *Cluster表示尿素组与硝酸组相比有显著差异表达的编码基因; ##表示未比对到藻类序列, 列出其同源性较高的物种 | ||||
为了较清楚地解释多形微眼藻中硝酸钠、尿素等氮源的转运和同化利用分子机制, 我们对相关基因参与的KEGG代谢途径进行分析, 构建了多形微眼藻的氮代谢通路(图 2), 其中包括硝酸盐同化、氨同化、尿素循环三部分。
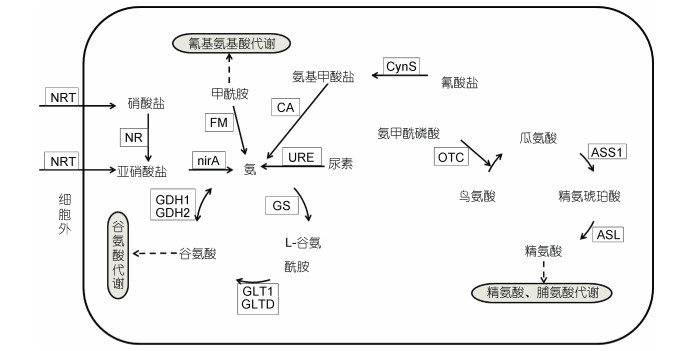 |
| 图 2 基于多形微眼藻转录组数据构建的氮代谢通路图 Fig. 2 Nitrogen metabolism pathway reconstructed based on M. polymorphus transcriptome 注: NRT:硝酸盐转运蛋白; NR:硝酸盐还原酶; nirA:亚硝酸盐还原酶; GLT1:谷氨酸合成酶(NADPH/NADH); GLTD:谷氨酸合成酶(铁氧还蛋白); GDH1:谷氨酸脱氢酶(NAD(P)+); GDH2:谷氨酸脱氢酶(NADP+); GS:谷氨酰胺合成酶; FM:尿氨酸甲酰胺酶; CA:碳酸酐酶; CynS:氰酸裂解酶; URE:脲酶; OTC:氨甲酰转移酶; ASS1:精氨琥珀酸合成酶; ASL:精氨琥珀酸裂解酶 |
在多形微眼藻中具有(亚)硝酸盐转运蛋白(NRT)和氮同化相关酶, 包括硝酸盐还原酶(NR)、亚硝酸盐还原酶(nirA)、谷氨酰胺合成酶(GS), 这些基因是多形微眼藻吸收和同化NO3-的重要基因组分。谷氨酰胺合成酶将氨转化成谷氨酰胺后参与谷氨酸循环, 在谷氨酸合成酶(GLT1/GLTD)的作用下接受来自α-酮戊二酸的碳骨架生成谷氨酸(Muro-Pastor et al, 2005), 最后通过谷氨酸脱氢酶(GDH1/GDH2)的作用转化成氨, 形成完整的GS-GOGAT循环, 谷氨酸合成酶是该循环的限速酶。如表 2所示, 在多形微眼藻中存在两种形式的谷氨酸合成酶, 它们分别以铁氧还蛋白和NADPH、NADH为辅酶。多形微眼藻氮代谢过程中氨的来源有五种, 除了硝酸盐同化和谷氨酸-谷氨酰胺循环过程中生成的氨外还存在其他三种氨的内源补充途径, 如尿素分解、氰酸盐分解、甲酰胺的催化转化都可以为氮代谢过程补充氨的消耗。细胞内的氰酸盐不但影响生物分子的生理功能, 还会对氨基酸的合成形成一定的干扰。氰酸盐裂解酶(cyanase)将氰酸盐分解为铵和二氧化碳, 不仅可以为氨同化补充氮源, 还可以为细胞的生理活动提供碳源。碳酸酐酶(carbonic anhydrase, CA)也可以催化氰酸盐分解为氨, 同时还能催化二氧化碳的可逆水合反应, 为酶反应过程提供CO2/HCO3-; 除此之外, 碳酸酐酶还可以通过移去CO2/HCO3-来推动反应产生能量(吴沿友等, 2006)。
在多形微眼藻中还存在着不完整的尿素-鸟氨酸代谢途径。在线粒体中, 鸟氨酸接受氨甲酰磷酸的氨甲酰基后生成瓜氨酸, 瓜氨酸从线粒体中脱出进入细胞溶胶中, 并接受来自天冬氨酸的碳骨架缩合成精氨琥珀酸, 精氨琥珀酸在精氨琥珀酸裂解酶(argininosuccinate lyase, ASL)的作用下脱去碳骨架, 生成精氨酸和延胡索酸。延胡索酸可作为中间代谢产物将尿素循环和TCA循环连接起来。有研究表明, 在已知基因组的假微型海链藻(T. pseudonana)、三角褐指藻(P. tricornutum)以及圆柱拟脆杆藻(Fragilariopsis cylindrus)中存在完整的鸟氨酸-尿素循环途径(Allen et al, 2011; Bender et al, 2012)。在荆晓丽等(2016)对玛氏骨条藻(Skeletonema marinoi)的氮代谢通路研究中也发现了从氨同化到尿素循环的完整代谢通路:玛氏骨条藻中的氨可以通过线粒体的氨甲酰磷酸合成酶Ⅰ与CO2合成氨甲酰磷酸, 然后将氨甲酰基转移给鸟氨酸合成瓜氨酸, 从而进入尿素循环。玛氏骨条藻细胞内的鸟氨酸-尿素循环是为了维持蛋白质的合成而对特殊条件作出的响应, 其细胞具有完整的尿素循环不仅有助于提高分解代谢过程中的氮的再分配效率, 还有利于在长期氮限制后迅速恢复, 也为玛氏骨条藻在海洋中占据一定的生态位提供了优势。而在多形微眼藻转录组中并未发现从氨同化过程到鸟氨酸代谢的关键酶基因——氨甲酰磷酸合成酶Ⅰ (CPSⅠ)和氨基甲酸激酶(ARCC)。硅藻尿素循环中的C、N通量可能是由线粒体蛋白氨甲酰磷酸合成酶Ⅰ (CPSⅠ)驱动和调控的, 在假微型海链藻和三角褐指藻中, 氨甲酰磷酸合成酶Ⅰ(CPSⅠ)在线粒体中作用于NH4+而不是谷氨酰胺, 从而参与尿素-鸟氨酸的循环的第一步(Allen et al, 2006)。同样氨基甲酸激酶(ARCC)也可以催化氨甲酰磷酸的合成, 而多形微眼藻中氨甲酰磷酸合成酶Ⅰ(线粒体)的缺乏则会阻碍细胞从氮限制中的恢复速率。参与鸟氨酸-尿素循环的精氨酸酶(arginase)在本转录组中也并未发现其参考基因, 精氨酸酶是尿素循环中生成尿素和鸟氨酸的关键酶, 缺少这一种酶就不能形成一个完整的鸟氨酸-尿素循环, 而这三种酶基因在多形微眼藻中未表达或是根本不存在尚不可知, 接下来我们会对这几种关键基因进行深入研究, 通过实时荧光定量分析方法来探索多形微眼藻和玛氏骨条藻中的尿素循环机制。
不同的硅藻种类对氮元素的利用有不同的响应, 每个种类对氮水平的变化敏感度也不同, 反映了不同硅藻种类氮吸收动力学的差异。相比玛氏骨条藻的氮代谢途径, 多形微眼藻的尿素循环并不完整, 缺少三种关键酶基因。同时在本转录组中, 不同氮源的脲酶基因表达量差异并不明显, 但实验结果表明多形微眼藻仍可以在尿素培养基中生长, 我们推测多形微眼藻可能具有特殊的尿素利用途径。鉴于浮游植物的异质性, 需要进一步研究不同硅藻细胞内的尿素循环是如何受环境因素调节。这些结果为研究玛氏骨条藻和多形微眼藻在营养胁迫下的不同响应机制提供了依据, 有利于我们从整体上把握硅藻的氮代谢途径。
2.4 氮代谢途径中差异表达基因分析 2.4.1 尿素培养下氮代谢的差异表达基因根据每百万序列片段中来自某一基因每千个碱基长度中包含的片段数计算出的FPKM (fragments per kilobase per million mapped reads)值, 比较了硝酸钠和尿素培养下的样品中参与氮代谢途径的差异表达基因。与硝酸钠培养组相比, 尿素培养下上调表达的编码基因有9个, 下调表达的编码基因有1个(图 3, 表 2中带*的为差异基因)。其中, 上调的基因对应的酶和蛋白包括硝酸盐同化的硝酸盐转运蛋白(NRT)、硝酸盐还原酶(NR)、亚硝酸盐还原酶(nirA)和参与氨同化的谷氨酸脱氢酶(GDH1/GDH2)、谷氨酸合成酶(GLT1/ GLTD)、氰酸裂解酶(cyanate lyase, CynS); 下调基因对应的酶是在氨态氮转化为谷氨酸过程中起重要作用的谷氨酰胺合成酶(GS)。
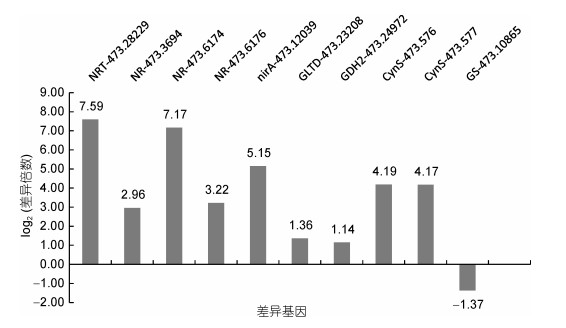 |
| 图 3 多形微眼藻氮代谢途径中差异表达的基因 Fig. 3 Genes expressed differentially in nitrogen metabolism of M. polymorphus 注: NRT:硝酸盐转运蛋白; NR:硝酸盐还原酶; nirA:亚硝酸盐还原酶; GLTD:谷氨酸合成酶(铁氧还蛋白); GDH2:谷氨酸脱氢酶(NADP+); CynS:氰酸裂解酶; GS:谷氨酰胺合成酶; NRT-473.28229等数字代表对应酶的基因编号; 硝酸钠组表达倍数视为1, 只列出尿素组的差异倍数, 图中数值代表log2(差异倍数) |
对两种氮源条件下多形微眼藻转录组中参与硝酸盐同化过程的(亚)硝酸盐转运蛋白、硝酸盐还原酶和亚硝酸盐还原酶的表达量比较发现:尿素组(亚)硝酸盐转运蛋白的基因表达量相对于硝酸钠作为氮源时显著上调, 尿素组三个编码硝酸盐还原酶的基因表达量均高于硝酸盐组, 编码亚硝酸盐还原酶的基因表达量也显著上调, 说明尿素培养下, 促进了硝酸盐同化基因的表达上调。硅藻中的氮转运蛋白具有较强的灵活性和较快的反应速度, 而且可以在高浓度下迅速吸收氮营养物质, 这种特性使得硅藻在富含氮的环境中具有竞争优势(Collos et al, 2005)。在许多真核微藻中, 硝酸盐转运蛋白的基因在硝酸盐缺乏时具有较高的表达量, 在硝酸盐充足时中度表达, 在铵盐条件下表达量最低(Hildebrand et al, 2000; Kang et al, 2009)。Ashworth等(2013)的研究报道了假微型海链藻中硝酸盐转运蛋白的基因在营养物质耗竭时表达最强, 增长最快。而在Mock等(2008)、Bender等(2014)的研究中提到硝酸盐转运蛋白基因是在NO3-的限制下诱导的(< 0.2mmol/L)。Dong等(2014)在抑食金球藻的对尿素和硝酸利用特征的研究中也表明, 抑食金球藻的亚硝酸盐转运蛋白在氮限制条件下表现出了强烈上调, 而在短期氮恢复过程中突然下调, 表明抑食金球藻的亚硝酸盐转运体基因受氮浓度的调节。根据这些结果, 我们推测在尿素条件下, 多形微眼藻会偏向于选择硝酸盐, 无论硝酸盐是否可以利用, 都会诱导硝酸盐转运蛋白的表达, 通过增加硝酸盐转运体的数量来调节硝酸盐的转运。
Joseph等(1998)对根管藻(Rhizosolenia)的研究发现, 当细胞外硝酸盐含量很低时(< 5nmol/L), 细胞内部存在硝酸盐的累积, 而且可以检测到硝酸盐还原酶的活性, 这个结果提示, 硝酸盐还原酶(NR)的活性是由细胞内部的硝酸盐诱导的, 与细胞外的硝酸盐浓度无关。同时在对小球藻和纤维藻的研究中发现, 在硝酸盐限制条件下, 细胞内的氮化合物被氧化成硝酸盐和亚硝酸盐, 进而诱导硝酸盐还原酶基因的转录(Kessler et al, 1970)。在Raven等(1987)的研究中提出, 浮游硅藻进化出一种营养储存液泡, 它能保存高浓度的硝酸盐和磷酸盐。储存液泡可以使硅藻适应脉冲式无机营养物质, 它的储存能力在没有外部营养来源的情况下足以使细胞分裂两到三次(Katz et al, 2004)。因此, 当多形微眼藻在尿素培养基中, 虽然硝酸盐利用受到了限制, 但其内部可能会存在储存硝酸盐的液泡, 进而诱导了硝酸盐还原酶基因的表达, 但是否会被翻译成蛋白质需要进一步验证。我们推测, 当在尿素培养基中生长时, 由于尿素的添加, 对硝酸盐吸收转化起到了正向反馈作用, 多形微眼藻需要通过提高相关基因的表达促进细胞的硝酸盐同化过程以维持更好的生命活动。
谷氨酸合成酶在氮代谢过程中起着中心作用, 在氨同化过程中可以把氨提供给氨基酸。谷氨酰胺合成酶是氮代谢过程中的多功能酶, 参与到多种氮代谢的调节, 且受光照、碳含量、氮素营养等环境因素的影响较大(Oliveira et al, 2001)。通过KEGG富集分析图发现参与谷氨酸代谢的三种酶的编码基因都表现出了较大的差异(图 3)。氮源的不同对这些氨同化酶表达的影响也有所差别, 其中, 尿素组谷氨酰胺合成酶的基因表达量较硝酸钠培养条件下显著下降, 表明无机氮源和有机氮源对多形微眼藻谷氨酰胺合成酶的影响有差异, 这可能是由于细胞内尿素含量过多引起体内pH值变化影响了谷氨酰胺合成酶活性导致的(王福源等, 1984)。谷氨酸脱氢酶(NADP+)(GDH2)基因在尿素组的表达量显著上调, 另一种谷氨酸脱氢酶(NAD(P)+)(GDH1)基因表达量则不明显, 这与Treberg等(2010)研究结果一致, NAD(P)H+谷氨酸脱氢酶在催化生化反应时既可利用NAD(H)+也可利用NADP(H)+为辅酶, 当两种辅酶均存在时, NADP(H)+的存在会抑制谷氨酸脱氢酶对NAD(H)+的利用率。另外, 参与谷氨酸合成的谷氨酸合成酶(GLTD)在尿素组的基因表达量则明显高于在硝酸钠培养下的基因表达量; 而另一种谷氨酸合成酶(GLT1)基因表达量并不显著。这两种酶属于不同的蛋白质, 其依赖还原力的专一性和抗原性及细胞定位等方面不尽相同, 这可能就导致了两种酶基因的表达差异。
在不同的氮元素下, 细胞的氮代谢调控有所不同, 当尿素作为唯一氮源时, 转化为氨的三种途径中所涉及到的酶基因表达量都呈现了上调表达, 而将氨转化为谷氨酰胺的谷氨酰胺合成酶基因表达量呈下调趋势。这可能与多形微眼藻对两种氮源的利用有关:当硝酸作为唯一氮源时, 多形微眼藻从(亚)硝酸盐还原为氨并参与氨基酸代谢的过程不受到氮元素的限制; 而当尿素作为唯一氮源时, 将尿素分解为氨的脲酶基因表达量并没有升高, 反而硝酸盐转化为氨的硝酸盐还原酶基因量高表达, 这表明多形微眼藻相比尿素可能优先选择硝酸盐作为氮源。在尿素培养组氰酸盐裂解酶和谷氨酸脱氢酶两种生成氨的酶基因表达量也有所提高, 这有可能是因为多形微眼藻对尿素的利用率不高导致的, 从而需要将更多的硝酸盐或氰酸盐转化为氨, 为氮代谢过程补充氨的消耗, 以维持正常的生命活动。这种代谢反应可以使细胞将能量转移到产生必要的化合物上, 以此来适应外部环境变化对细胞带来的压力。硝酸盐转运蛋白、硝酸盐还原酶基因在不同氮源条件下的差异表达, 表明它们在氮的利用方面起着重要作用, 为我们进一步研究硅藻细胞中的氮同化和氮分配提供了依据。
多形微眼藻常在褐潮期间伴随着抑食金球藻大量繁殖, 有研究表明:抑食金球藻通常在溶解有机氮浓度较高和溶解无机氮浓度相对较低的海域暴发(Gobler et al, 2004); 在秦皇岛海域营养盐与浮游植物群落结构的研究中也证实了这一观点: 2013年3月至6月, 随着秦皇岛海域尿素、溶解游离态氨基酸、溶解结合态氨基酸浓度的增加, 硝酸盐和亚硝酸盐浓度的降低, 抑食金球藻的密度呈现增长趋势(Qiao et al, 2017)。其他研究表明, 抑食金球藻对尿素的吸收能力高于硝酸盐、谷氨酸和铵, 当尿素与硝酸盐共存时, 以尿素作为主要氮源, 而非硝酸盐(Dong et al, 2014)。在对多形微眼藻的研究中发现, 多形微眼藻相比尿素更喜欢利用硝酸盐, 其他研究也表明, 硅藻会优先利用硝酸盐(Lomas et al, 2001; 张云, 2013)。在抑食金球藻暴发的海域, 这种对硝酸盐的优先利用特点可能会增加多形微眼藻相较于其他浮游植物的竞争力。
2.4.3 尿素循环差异表达基因分析根据基因预测和注释的结果分析发现, 多形微眼藻中只含有1种脲酶基因(urease), 且在以尿素为氮源培养条件下, 并未发现多形微眼藻的脲酶基因有差异表达, 参与尿素循环的关键酶基因氨甲酰转移酶(OTC)、精氨琥珀酸合成酶(ASS1)、精氨琥珀酸裂解酶(ASL)的表达量也未发现显著差异, 表明氮源对多形微眼藻细胞内的尿素循环途径影响不明显。有研究表明, 脲酶活性可能受到细胞内尿素的诱导作用, 也可能随着尿素循环活动或者氨基酸和嘌呤分解代谢尿素的速率的变化而变化(Fan et al, 2003; Solomon et al, 2010)。多形微眼藻中脲酶基因的存在, 可以将尿素水解为NH4+和CO2, 这说明该藻可以利用尿素作为氮源。无论在尿素组或者硝酸盐组, 均检测到了脲酶基因的转录, 这表明脲酶的转录不仅受外源尿素的调控, 且在这两种氮源条件下, 脲酶的转录结果并未发现明显差异, 这证实了Allen等(2011)的研究结果:无论哪种氮源形式, 细胞内尿素的生成和循环都具有相似的、稳定的途径, 这一结论在对三角褐指藻的研究中也得到了证实:脲酶基因在氮限制和营养盐充足时的表达差异亦不明显。同时我们发现, 参与氮代谢的另一通路, 氰酸盐的裂解中氰酸盐裂解酶的表达量在尿素组显著上调, 由于氰酸盐在细胞内是氨甲酰磷酸和尿素的分解产生的, 这可能是由于尿素含量较多对它们基因表达的正调控作用; 而氰酸盐裂解的产物氨甲酰磷酸又可以通过谷氨酰胺的合成产生(Harano et al, 1997), 因此我们推测细胞内氨的含量和尿素的含量可能会影响氰酸裂解酶基因的表达。
微藻对氮素营养的吸收利用过程涉及与氮代谢相关的多个基因, 通过对多形微眼藻转录组序列的分析, 分析了多形微眼藻在应对不同氮源时所做的分子响应, 为研究硅藻在利用不同氮源时氮代谢中关键基因的表达调控模式奠定了基础。不同营养条件不仅对氮素营养的吸收和循环具有显著影响, 而且对藻细胞的大多数代谢通路和调控系统也会产生不同程度的影响。硅藻对氮的吸收和同化过程与其碳代谢具有十分密切的关系, 氮代谢过程需要来自光合作用的ATP和还原当量(Falkowski et al, 1975)和来自TCA循环的碳骨架(Hockin et al, 2012)。在本转录组中, 我们发现了碳代谢通路在两种培养条件下也有不同的响应: C4过程中的关键酶和限速酶丙酮酸磷酸双激酶(PPDK)的基因在尿素组表达显著上调, 参与催化羧化和脱羧反应的磷酸烯醇式丙酮酸羧化酶(PEPC)和磷酸烯醇式丙酮酸羧激酶(PEPCK)的编码基因在两种氮源下表达差异比较显著, 光合作用暗反应过程中的转羟乙醛酶(TKT)、核酮糖磷酸激酶(PRK)对应的转录本在尿素和硝酸钠条件下也具有显著差异的表达。这些编码基因的表达变化不仅直接影响碳代谢相关的酶活性和代谢产物, 而且可能会对氮代谢产生一定的影响。这一现象为后续要开展的实验明确了方向, 接下来我们会根据转录组数据和荧光定量PCR实验深入分析在两种不同氮源条件下碳代谢的分子响应机制。
3 结论微藻在受到外界环境变化刺激后, 会启动其自身的内在调控机制来适应新环境, 如通过调控细胞内的代谢途径来改变相关基因的表达水平。本实验基于多形微眼藻的转录组数据, 分析了其在不同氮源处理下的氮代谢通路, 进一步分析了不同硅藻种类的氮代谢途径差异。参与氮代谢过程的硝酸盐还原酶、亚硝酸盐还原酶等在基因表达水平的变化体现了多形微眼藻在利用不同氮素营养时做出的分子响应。同时其在尿素培养基中也可正常生存, 表明多形微眼藻在进化过程中产生了利用多种形式氮源的适应性, 这在一定程度上解释了多形微眼藻为何会成为秦皇岛海域褐潮暴发时的第二大优势种。本研究通过探讨多形微眼藻在不同氮素营养条件下的转录响应, 从分子生物学角度阐述了其对海域中营养盐的利用特征, 可为监测和预防有害藻华的暴发提供有利依据。
致谢 感谢厦门大学国家重点实验室高亚辉教授课题组提供的多形微眼藻藻种。
王福源, 王妙虎, 王锦华, 等. 1984. 尿素对生成L-谷氨酰胺的影响. 微生物学杂志, 4(2): 6-8 |
吴沿友, 李西腾, 郝建朝, 等. 2006. 不同植物的碳酸酐酶活力差异研究. 广西植物, 26(4): 366-369 DOI:10.3969/j.issn.1000-3142.2006.04.007 |
张云, 2013.不同类群代表性浮游植物对尿素的生理生态响应.广州: 暨南大学硕士学位论文, 108-118 http://cdmd.cnki.com.cn/Article/CDMD-10559-1013027635.htm
|
荆晓丽, 米铁柱, 甄毓, 等. 2016. 基于玛氏骨条藻(Skeletonema marinoi)转录组的氮代谢途径解析. 海洋环境科学, 35(5): 703-711 |
Allen A E, Dupont C L, Oborník M et al, 2011. Evolution and metabolic significance of the urea cycle in photosynthetic diatoms. Nature, 473(7346): 203-207 DOI:10.1038/nature10074 |
Allen A E, Vardi A, Bowler C, 2006. An ecological and evolutionary context for integrated nitrogen metabolism and related signaling pathways in marine diatoms. Current Opinion in Plant Biology, 9(3): 264-273 DOI:10.1016/j.pbi.2006.03.013 |
Ashworth J, Coesel S, Lee A et al, 2013. Genome-wide diel growth state transitions in the diatom Thalassiosira pseudonana. Proceedings of the National Academy of Sciences of the United States of America, 110(18): 7518-7523 DOI:10.1073/pnas.1300962110 |
Bender S J, Durkin C A, Berthiaume C T et al, 2014. Transcriptional responses of three model diatoms to nitrate limitation of growth. Frontiers in Marine Science, 1: 3 |
Bender S J, Parker M S, Armbrust E V, 2012. Coupled effects of light and nitrogen source on the urea cycle and nitrogen metabolism over a diel cycle in the marine diatom Thalassiosira pseudonana. Protist, 163(2): 232-251 DOI:10.1016/j.protis.2011.07.008 |
Camacho C, Coulouris G, Avagyan V et al, 2009. BLAST+: architecture and applications. BMC Bioinformatics, 10(1): 421 |
Cha T S, Chen J W, Goh E G et al, 2011. Differential regulation of fatty acid biosynthesis in two Chlorella species in response to nitrate treatments and the potential of binary blending microalgae oils for biodiesel application. Bioresource Technology, 102(22): 10633-10640 DOI:10.1016/j.biortech.2011.09.042 |
Collos Y, Vaquer A, Souchu P, 2005. Acclimation of nitrate uptake by phytoplankton to high substrate levels. Journal of Phycology, 41(3): 466-478 DOI:10.1111/j.1529-8817.2005.00067.x |
Dham V V, Wafar M, Heredia A M, 2005. Nitrogen uptake by size-fractionated phytoplankton in mangrove waters. Aquatic Microbial Ecology, 41(3): 281-291 |
Dong H P, Huang K X, Wang H L et al, 2014. Understanding strategy of nitrate and urea Assimilation in a Chinese Strain of Aureococcus anophagefferens through RNA-Seq Analysis. PLoS One, 9(10): e111069 DOI:10.1371/journal.pone.0111069 |
Falkowski P G, Stone D P, 1975. Nitrate uptake in marine phytoplankton: energy sources and the interaction with carbon fixation. Marine Biology, 32(1): 77-84 DOI:10.1007/BF00395161 |
Fan C, Glibert P M, Alexander J et al, 2003. Characterization of urease activity in three marine phytoplankton species, Aureococcus anophagefferens, Prorocentrum minimum, and Thalassiosira weissflogii. Marine Biology, 142(5): 949-958 DOI:10.1007/s00227-003-1017-8 |
Götz S, García-Gómez J M, Terol J et al, 2008. High-throughput functional annotation and data mining with the Blast2GO suite. Nucleic Acids Research, 36(10): 3420-3435 DOI:10.1093/nar/gkn176 |
Grabherr M G, Haas B J, Yassour M et al, 2011. Full-length transcriptome assembly from RNA-Seq data without a reference genome. Nature Biotechnology, 29(7): 644-652 DOI:10.1038/nbt.1883 |
Harano Y, Suzuki I, Maeda S et al, 1997. Identification and nitrogen regulation of the cyanase gene from the cyanobacteria Synechocystis sp. strain PCC 6803 and Synechococcus sp. strain PCC 7942. Journal of Bacteriology, 179(18): 5744-5750 |
Hildebrand M, Dahlin K, 2000. Nitrate transporter genes from the diatom Cylindrotheca fusiformis (bacillariophyceae): mRNA levels controlled by nitrogen source and by the cell cycle. Journal of Phycology, 36(4): 702-713 DOI:10.1046/j.1529-8817.2000.99153.x |
Hockin N L, Mock T, Mulholland F et al, 2012. The response of diatom central carbon metabolism to nitrogen starvation is different from that of green algae and higher plants. Plant Physiology, 158(1): 299-312 DOI:10.1104/pp.111.184333 |
Joseph L, Villareal T A, 1998. Nitrate reductase activity as a measure of nitrogen incorporation in Rhizosolenia formosa (H. Peragallo): Internal nitrate and diel effects. Journal of Experimental Marine Biology and Ecology, 229(2): 159-176 DOI:10.1016/S0022-0981(98)00047-1 |
Kanehisa M, Araki M, Goto S et al, 2008. KEGG for linking genomes to life and the environment. Nucleic Acids Research, 36(S1): D480-D484 |
Kang L K, Hwang S P L et al, 2009. Establishment of minimal and maximal transcript levels for nitrate transporter genes for detecting nitrogen deficiency in the marine phytoplankton Isochrysis galbana (prymnesiophyceae) and Thalassiosira pseudonana (bacillariophyceae). Journal of Phycology, 45(4): 864-872 DOI:10.1111/j.1529-8817.2009.00698.x |
Katz M E, Finkel Z V, Grzebyk D et al, 2004. Evolutionary trajectories and biogeochemical impacts of marine eukaryotic phytoplankton. Annual Review of Ecology, Evolution, and Systematics, 35: 523-556 DOI:10.1146/annurev.ecolsys.35.112202.130137 |
Kessler E, Oesterheld H, 1970. Nitrification and Induction of Nitrate Reductase in Nitrogen-deficient Algae. Nature, 228(5268): 287-288 DOI:10.1038/228287a0 |
Lomas M W, Glibert P M, Clougherty D A et al, 2001. Elevated organic nutrient ratios associated with brown tide algal blooms of Aureococcus anophagefferens (Pelagophyceae). Journal of Plankton Research, 23(12): 1339-1344 DOI:10.1093/plankt/23.12.1339 |
Love M I, Huber W, Anders S, 2014. Moderated estimation of fold change and dispersion for RNA-seq data with DESeq2. Genome Biology, 15(12): 550 DOI:10.1186/s13059-014-0550-8 |
Mock T, Samanta M P, Iverson V et al, 2008. Whole-genome expression profiling of the marine diatom Thalassiosira pseudonana identifies genes involved in silicon bioprocesses. Proceedings of the National Academy of Sciences of the United States of America, 105(5): 1579-1584 DOI:10.1073/pnas.0707946105 |
Moriya Y, Itoh M, Okuda S et al, 2007. KAAS: an automatic genome annotation and pathway reconstruction server. Nucleic Acids Research, 35(S2): W182-W185 |
Muro-Pastor M I, Reyes J C, Florencio F J, 2005. Ammonium assimilation in cyanobacteria. Photosynthesis Research, 83(2): 135-150 |
Oliveira I C, Brenner E, Chiu J et al, 2001. Metabolite and light regulation of metabolism in plants: lessons from the study of a single biochemical pathway. Brazilian Journal of Medical and Biological Research, 34(5): 567-575 DOI:10.1590/S0100-879X2001000500003 |
Qiao L, Chen Y, Mi T et al, 2017. Quantitative PCR analysis of the spatiotemporal dynamics of Aureococcus anophagefferens and Minutocellus polymorphus and the relationship between brown tides and nutrients in the coastal waters of Qinhuangdao, China. Journal of Applied Phycology, 29(1): 297-308 DOI:10.1007/s10811-016-0959-4 |
Raven J A, 1987. The role of vacuoles. New Phytologist, 106(3): 357-422 DOI:10.1111/j.1469-8137.1987.tb00149.x |
Solomon C M, Collier J L, Berg G M et al, 2010. Role of urea in microbial metabolism in aquatic systems: a biochemical and molecular review. Aquatic Microbial Ecology, 59(1): 67-88 |
Taylor G T, Gobler C J, Sañudo-Wilhelmy S A, 2006. Speciation and concentrations of dissolved nitrogen as determinants of brown tide Aureococcus anophagefferens bloom initiation. Marine Ecology Progress Series, 312: 67-83 DOI:10.3354/meps312067 |
Treberg J R, Brosnan M E, Brosnan J T, 2010. The simultaneous determination of NAD(H) and NADP(H) utilization by glutamate dehydrogenase. Molecular and Cellular Biochemistry, 344(1-2): 253-259 DOI:10.1007/s11010-010-0549-8 |
Walsh G E, McLaughlin L L, Yoder M J et al, 1988. Minutocellus polymorphus: a new marine diatom for use in algal toxicity tests. Environmental Toxicology and Chemistry, 7(11): 925-929 |
 2019, Vol. 50
2019, Vol. 50


