中国海洋湖沼学会主办。
文章信息
- 王坤, 王沛, 张浩, 张树峰, 王大志. 2019.
- WANG Kun, WANG Pei, ZHANG Hao, ZHANG Shu-Feng, WANG Da-Zhi. 2019.
- 米氏凯伦藻细胞表面膜蛋白质组及其对温度变化的响应研究
- THE CELL SURFACE PROTEOME OF KARENIA MIKIMOTOI AND ITS RESPONSE TO TEMPERATURE CHANGE
- 海洋与湖沼, 50(3): 652-663
- Oceanologia et Limnologia Sinica, 50(3): 652-663.
- http://dx.doi.org/10.11693/hyhz20180700189
文章历史
-
收稿日期:2018-07-30
收修改稿日期:2018-11-05
2. 中国科学院海洋生态与环境科学重点实验室 青岛 266071
2. CAS Key Laboratory of Marine Ecology and Environmental Sciences, Institute of Oceanology, Chinese Academy of Sciences, Qingdao 266071, China
米氏凯伦藻(Karenia mikimotoi Hansen)是一种单细胞浮游植物, 属于裸甲藻目, 可以产生鱼毒素和溶血性毒素, 近年来常在我国近岸尤其是浙江、福建和广东沿海形成藻华, 给渔业养殖造成了巨大的损失。米氏凯伦藻没有细胞壁, 其细胞膜直接和外界环境相接触, 因此细胞表面的膜蛋白可能在营养盐的跨膜转运、细胞通讯、信号应答和能量传递等方面起着非常重要作用(Musil et al, 1990; Snyders et al, 1999; Hicke et al, 2003), 对调控细胞的基本生理功能甚至藻华的形成可能有重要作用, 但目前这方面的研究非常缺乏, 一定程度上也限制了我们对米氏凯伦藻藻华形成机制的认识。
细胞表面膜蛋白质的研究一直是生命科学领域的重点。目前国内外已建立了多种研究方法, 直接对细胞表面的膜蛋白进行标记是一种常用的方法。早期的研究是对整个细胞表面膜蛋白进行生物素化(Palenik et al, 1995)和矢量标记(Bertomeu et al, 2003), 但是这样的处理方法会导致膜蛋白溶解度降低, 只能鉴定到少数蛋白质。近来有学者利用荧光染料在体内和体外标记无细胞壁的人类细胞株系(Mayrhofer et al, 2006)和细菌表面膜蛋白(Anaya et al, 2007)。通过直接标记人类单核细胞表面膜蛋白的方法, 推测出单核细胞的活性和氧化压力受病毒影响, 但不影响病毒感染的动力学(Kadiu et al, 2009)。Li等(2012)也将荧光染料标记方法应用于藻亚历山大藻细胞表面蛋白研究, 通过直接标记, 鉴定到38个细胞表面蛋白。这些研究表明, 荧光染料可直接用于标记细胞表面蛋白, 开展细胞表面膜蛋白研究。
研究表明, 东海海域的藻华演替与温度具有很好的相关性(张传松, 2008)。我国闽浙沿海的米氏凯伦藻藻华通常发生在每年的5、6月份, 此时海水温度升高迅速, 因此推测米氏凯伦藻藻华暴发与海水升温存在一定的关联性。沈盎绿等(2014)发现在东海海水温度较常年高时, 米氏凯伦藻藻华暴发几率会增大; 而当东海海区海水温度较常年下降时, 东海原甲藻赤潮暴发几率会增大。因此, 研究米氏凯伦藻对环境温度变化的响应对可能有助于揭示其藻华形成机制。
本文利用荧光染料标记技术, 建立了米氏凯伦藻细胞表面膜蛋白标记、分离和鉴定方法, 并且比较了不同温度培养条件下米氏凯伦藻细胞表面膜蛋白表达的差异, 探讨了这些膜蛋白在米氏凯伦藻适应环境温度变化时的作用。
1 材料与方法 1.1 藻种及培养条件本实验所用米氏凯伦藻(Karenia mikimotoi)由厦门大学近海海洋环境科学国家重点实验室海洋藻类收集中心(CCMA)所提供, 编号CCM-083, 于2012年分离于中国福建苏澳海区(25°37′N, 119°42'E)。藻细胞培养在K培养基中(Keller et al, 1987), 培养条件为:海水盐度30, 光照强度100μE/(m2·s), 温度(20±1)℃, 光暗周期为14h:10h。培养用海水采自厦门湾, 使用前用0.22μm滤膜过滤后灭菌。温度实验设置五组(12℃, 16℃, 20℃, 12℃→16℃, 12℃→20℃), 每组6个平行, 变温组在第五天变温, 藻细胞在实验前经过相应温度驯化。
1.2 生长参数测定每天在固定时间(光照周期第8小时左右)采集细胞计数样1mL加入鲁格试剂后在光学显微镜下进行细胞计数, 计算各培养液中的细胞密度。另取5mL藻液, 在相应培养温度下暗处理5—10min后读取Fv/Fm值, 测定PSII最大光化学量子产量。
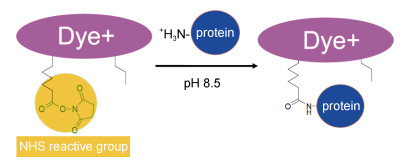
1.3 细胞样品收集及处理各组的藻液在指数期时通过离心(1000g, 5min)收集藻细胞。细胞表面膜蛋白质标记所用的染料为Amershamatm CyDye DIGE Fluors (mimimal dyes)荧光染料, 通过蛋白质分子上的赖氨酸残基与染料N-hydroxy-sulfosuccinimide (NHS)的酯基团共价结合, 如图 1所示。
将收集的藻细胞用1mL磷酸盐缓冲溶液PBS (0.02mol/L phosphate, 0.45mol/L NaCl, pH 7.5)洗涤, 加入Hank’s平衡缓冲溶液HBSS (5.33mmol/L KCl, 413.79mmol/L NaCl, 1.26mmol/L CaCl2, 4.17mmol/L NaHCO3, 0.407mmol/L MgSO4, 0.493mmol/L MgCl2, 0.338mmol/L Na2HPO4, 5.56mmol/L D-Glucose, 0.441mmol/L KH2PO4)后于4℃, 800g离心5min, 沉淀重悬于200μL HBSS (含1mol/L urea, pH 8.5)缓冲溶液中, 加入600pmol的cy5染料(温度实验标记方案如表 1所示), 于4℃暗处孵育20min进行标记处理。反应完后加入20μL 10mmol/L的lysine, 4℃暗处反应10min后4℃, 800g离心去上清液。用500μL HBSS (pH 7.4, 盐度30)的缓冲液洗涤2次, 4℃, 800g离心5min去上清液后–80℃保存。
| gel | cy2 | cy5 |
| 1 | 内标 | 16℃ |
| 2 | 内标 | 20℃ |
| 3 | 内标 | 12℃→20℃ |
| 4 | 内标 | 12℃ |
| 5 | 内标 | 12℃→16℃ |
| 6 | 内标 | 12℃→16℃ |
| 7 | 内标 | 12℃ |
| 8 | 内标 | 12℃→20℃ |
将标记的藻细胞样品通过trizol提取法提取大部分蛋白, 然后结合顺序提取法提取膜蛋白。第一步向样品中加入trizol试剂(Invitrogen, Carlsbad, CA, USA), 按照之前报道的方法进行提取(Wang et al, 2013); 第二步参照Molloy等人的方法进行提取(Molloy et al, 1998), 并向得到的上清液中加入20% TCA/丙酮(W/V), 震荡充分混匀后放置于–20℃过夜。4℃, 20000g离心30min后向沉淀中加入1mL含有20mm DTT的100%丙酮, 4℃静止30min, 相同条件下离心, 取沉淀并重复此步骤3次。向沉淀中加入适量重溶胀液使蛋白溶解。提取的蛋白使用GE healthcaretm公司的2-D quant kit试剂盒进行蛋白定量, 在480nm吸收波长下用酶标仪测定BSA标准品和样品的吸光值, 绘制BSA浓度标准曲线, 以标准曲线计算每个样品的蛋白浓度。
1.5 蛋白质分离与鉴定使用2-D DIGE方法对标记的细胞表面膜蛋白进行分离, 双向电泳的等点聚焦条件如表 2所示。蛋白样品溶液分别加入1% DTT和0.5% IPG buffer, 用重溶液(RB)补齐至450μL, 混匀后15000g离心20min取上清液。使用pH 4—7的IPG胶条, 按照表 2设置的等电聚焦程序进行蛋白质一向分离。等电聚焦结束之后立即将胶条在平衡液Ⅰ(6mol/L urea, 50mmol/L Tris, pH 8.8, 30% V/V glycerol, 2% SDS, a trace of bromophenol blue, 1% DTT)和平衡液Ⅱ(6mol/L urea, 50mmol/L Tris, pH 8.8, 30% V/V glycerol, 2% SDS, a trace of bromophenol blue, 2.5%碘乙酰胺)各平衡17min, 之后进行第二向SDS-PAGE电泳, 分离胶浓度12%, 浓缩胶浓度5%。
| 步骤 | 电压 | 时间(h)/伏.小时(V.h) | 升压类型 | 温度(℃) | 电流(μA) |
| 1 | 100 | 6h | step | 20 | < 75 |
| 2 | 500 | 30min | grad | 20 | < 75 |
| 3 | 1000 | 30min | grad | 20 | < 75 |
| 4 | 2000 | 1h | grad | 20 | < 75 |
| 5 | 10000 | 1h | grad | 20 | < 75 |
| 6 | 10000 | 60kVh | step | 20 | < 75 |
| 合计 | 20h/78kVh | ||||
使用typHoon 9500荧光扫描仪对得到的胶图进行扫描, 参数设为扫描滤光片波长为633nm, 发射波长为670nm。温度实验部分中cy2内参样品参数设为扫描滤光片波长为488nm, 发射波长为520nm。扫描的图像用decydertm 2D 7.2软件进行分析, 利用decyder 7.2软件中的differential in-gel analysis (DIA)模块对胶内蛋白点进行识别检测, 同时以cy2内参样品的荧光强度对胶内另一个蛋白荧光点进行归一化分析。设置参数为:点数为2500, 最大容量小于30000。对不同温度条件下蛋白质数据进行t-test和ANOVA方差分析, 找出相互之间差异表达的蛋白质点并生成差异列表。蛋白质丰度差异值大于1.5倍并且统计学P值小于0.5认为是可信的, 后进行下一步的酶解和质谱鉴定。将凝胶保存在3%乙酸之中, 4℃保存以备后续挖点鉴定。
1.6 蛋白质酶解与分析根据荧光扫描结果, 切下荧光对应的蛋白质点胶粒, 脱色后加入适量酶液(10ng/μL, 用10mmol/L的碳酸氢铵溶解胰蛋白酶)覆盖胶粒, 然后置于4℃下30min使胶粒充分溶胀。加入10mmol/L碳酸氢铵覆盖胶粒, 50℃空气中放置2h。酶解完的肽段样品采用AB SCIEX 5800 MALDI TOF/TOFTM MS质谱仪进行鉴定分析。同时使用Sequenzyme peptide标准试剂盒(AB SCIEX)对质谱仪进行校准, AB SCIEX自带的TOF/TOFTM series explorertm软件做一级质谱同时自动选择母离子进行二级质谱。一级模式设定:激光脉冲频率为400Hz; 并进行1000次激光打击形成一级图谱; 质荷比扫描范围选择850—4000Da; 信噪比(S/N)为10;其局部的噪音窗口宽度的质荷比为250, 最小半峰宽为2.9bins。二级模式之下:激光脉冲频率为1000Hz; 进行2000次激光打击, 并累积成图。每一个分析的蛋白点选择15个一级质谱的谱峰S/N大于50的母离子进行MS/MSC串联二级质谱分析。MALDI TOF/TOF质谱鉴定的结果采用GPS Explorer 3.5软件包以及MASCOT 2.2数据库搜索软件对蛋白质谱一级、二级质谱结果进行联合搜库(Wang et al, 2011)。将鉴定到的蛋白质用iProClass数据库和Toulza等(2010)报道的结果进行GO功能注释。
2 结果 2.1 细胞表面膜蛋白标记及鉴定米氏凯伦藻细胞表面膜蛋白经过荧光染料cy5标记后用trizol提取法结合顺序提取法提取, 蛋白质双向凝胶电泳分离结果如图 2所示。通过软件分析对比, 共检测到44个蛋白质荧光点, 它们可能是细胞表面蛋白。
 |
| 图 2 米氏凯伦藻细胞表面膜蛋白三步提取法的双向凝胶电泳荧光图 Fig. 2 The 2-D map of cell surface membrane proteins extracted using sequential extraction |
将44个荧光蛋白质点酶解后用MALDI- TOF/TOF质谱鉴定到27个可信蛋白, 其中在甲藻纲蛋白质数据库中鉴定到11个可信蛋白(表 3); 通过de novo结合MS-blast鉴定到16个可信蛋白(表 4), 其他17个蛋白质点未能被鉴定。在所鉴定的蛋白质中存在蛋白异构体, 在胶图上的分子量和等电点存在差异, 但都被鉴定为同一类蛋白, 这可能是同一个蛋白家族的异构体或只是同一个基因表达产物的不同修饰体(Hockin et al, 2012)。
| 胶点 | 蛋白名称 | 物种 | NCBI登录号 | 现论分子量(Da) | 理论等电点pI | 匹配肽段数 | 蛋白质得分 | 得分罝信度(%) | 总离子得分 | 总离子罝信度(%) |
| 4 | 3; short=pcp | Kryptoperidin ium foliaceum | 297385107 | 44544.5 | 4.78 | 5 | 60 | 99.345 | 44 | 99.906 |
| 5 | bip /heat shock protein 70 | Crypthecodinium cohnii | 20198944 | 74439.3 | 5.15 | 18 | 558 | 100 | 485 | 100 |
| 7 | light-harvesting protein | Symbioditiium sp. c3 | 306430545 | 66010.2 | 7.6 | 3 | 78 | 99.989 | 72 | 100 |
| 8 | light-harvesting protein | Symbiodinium sp. c3 | 306430563 | 54070.4 | 7.74 | 4 | 53 | 96.866 | 44 | 99.916 |
| 9 | chloroplast light harvesting complex protein | Heterocapsa triquetra | 58613555 | 26741.7 | 6.17 | 3 | 57 | 98.534 | 46 | 99.939 |
| 11 | putative chloroplast light harvesting protein | Lepidodinium chlorophorum | 338746086 | 20253.2 | 5.8 | 3 | 59 | 99.137 | 46 | 99.952 |
| 16 | chloroplast light harvesting protein isoform 12 | Karlodinium veneficum | 77024193 | 42318.4 | 5.4 | 4 | 100 | 100 | 89 | 100 |
| 20 | chloroplast light harvesting protein isoform 8 | Karlodinium veneficum | 77024185 | 42628.5 | 5.27 | 5 | 96 | 100 | 82 | 100 |
| 25 | chloroplast light harvesting protein isoform 5-1 ike protein | Karlodinium veneficum | 157093103 | 18701.6 | 9.25 | 3 | 52 | 95.257 | 42 | 99.836 |
| 41 | chlorophyll a/b-binding protein isoforin 4 /actin-related protein - arp5p | Karlodinium veneficum | 255965856 | 24033.4 | 5.85 | 4 | 60 | 99.248 | 43 | 99.881 |
| 44 | photosystem ⅱ d2 apoprotein (chloroplast) | karenia mikimotoi | 422034799 | 16031.8 | 4.8 | 4 | 94 | 100 | 70 | 100 |
| 胶点 | 蛋白名称 | 物种 | 登录号 | 总卨得分片段数 | 匹配肽段数 | 匹配肽段数>62 | 匹配肽段且高得分片段>104 | 两个独立肽段且高得分片段>134 |
| 1 | membrane protein | Prevote Ha sp. oral taxon 317 | 496523478 | 64 | 1 | 1 | ||
| 2 | hypothetical protein dappudraft_302881 | DapHnia pulex | 321470999 | 159 | 4 | |||
| 6 | hypothetical protein, conserved | Toxoplasma GOndii me49 | 237837237 | 207 | 4 | 1 | y | y |
| 10 | light-harvesting protein, partial | Symbiodinium sp. c3 | 306430557 | 173 | 3 | 1 | y | y |
| 14 | hypothetical protein | Oribacterium sp. oral | 496506261 | 67 | 1 | 1 | ||
| 15 | efflux transporter, rnd family, mfp subunit | Bradyrhizobium sp. dfci-l | 540135867 | 149 | 2 | 2 | y | |
| 17 | nodulation receptor kinase | Melilotus albus | 21698802 | 95 | 2 | 1 | ||
| 19 | ABC transporter substrate-binding protein | Streptomyces himastatinicus | 497403020 | 71 | 1 | 1 | ||
| 27 | membrane protein | Achromobacter piechaudii | 493248717 | 129 | 3 | 1 | ||
| 28 | predicted: keratin, type ii cuticular hb2 | Equus caballus | 194212013 | 171 | 3 | |||
| 30 | protein hum-10 | Caenorhabditis elegans | 392925914 | 110 | 2 | 1 | y | |
| 32 | ABC transporter substrate-binding protein | Subdoligranulum sp. | 496683461 | 105 | 2 | 1 | y | |
| 33 | traO-interacting protein 1 | Myotis brandtii | 521024389 | 147 | 3 | y | ||
| 35 | light harvesting protein | Emiliania huxleyi ccmpI5l6 | 551555397 | 254 | 5 | 1 | y | y |
| 38 | hypothetical protein trividraft_l 38892 | Trichoderma virens gv29-8 | 358387764 | 68 | 1 | 1 | ||
| 39 | rhodanese-like domain-containing protein | Colletotrichum gram inicola m 1.001 | 310795854 | 100 | 2 | 1 |
对鉴定到的27个蛋白质进行GO功能注释分类, 结果如图 3所示。其中膜蛋白共9个占33%, 叶绿体的类囊体蛋白7个占26%, 未得到分类的蛋白质7个占26%, 其他少数蛋白质被注释到胞内蛋白。
 |
| 图 3 基于细胞成份组成的GO注释分类 Fig. 3 The GO classifications of the annotated proteins |
米氏凯伦藻在20℃、16℃和12℃三个温度条件下经过4个月的温度驯化后生长情况如图 4所示。20℃条件下米氏凯伦藻始终处于较快生长状态, 在16℃、12℃条件下米氏凯伦藻生长缓慢。在第4天将12℃条件下的米氏凯伦藻分别转移到20℃和16℃条件下, 转到20℃条件下的藻细胞密度出现了短暂降低的现象, 之后细胞进入快速生长状态, 而转到16℃条件下的藻细胞生长较缓慢。
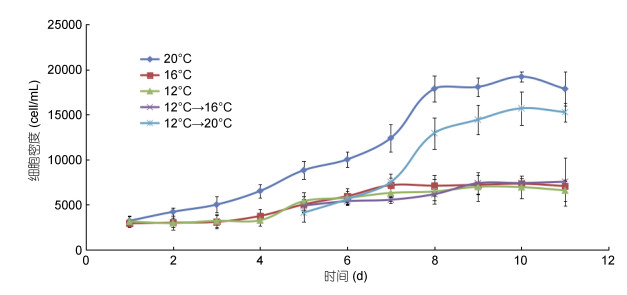 |
| 图 4 米氏凯伦藻在不同温度梯度条件下的生长曲线 Fig. 4 Growth curves of K. mikimotoi at different growth temperatures |
五个温度梯度条件下米氏凯伦藻的最大光化学量子产量(Fv/Fm)如图 5。20℃条件下最大。当第4天变温时, 在第5天12℃→20℃, 12℃→16℃两组的Fv/Fm降低, 特别是在12℃→20℃组, 之后迅速恢复正常。
 |
| 图 5 米氏凯伦藻5个温度梯度下Fv/Fm变化 Fig. 5 Variations of Fv/Fm of K. mikimotoi at different growth temperatures |
对五种温度处理条件下的米氏凯伦藻表面蛋白进行2D-DIGE分离, 并用Decyder 7.0软件对各组样品间差异蛋白进行识别分析。两两温度之间差异表达的蛋白满足数据统计学意义且蛋白丰度变化大于1.5倍的蛋白共有6个, 如图 6和表 5所示。
 |
| 图 6 米氏凯伦藻细胞表面蛋白质组凝胶电泳图 Fig. 6 The 2D map of differential expression membrane proteins of K. mikimotoi |
| 温度对比方案* | 上调蛋白个数 | 下调蛋白个数 |
| 12℃/20℃ | 2 | 0 |
| 16℃/20℃ | 1 | 0 |
| 12℃/16℃ | 0 | 0 |
| 12℃→20℃/20℃ | 1 | 2 |
| 12℃→16℃/16℃ | 1 | 4 |
| 12℃→16℃/12℃ | 1 | 3 |
| 12℃→20℃/12℃ | 1 | 4 |
| 12℃→16℃/12℃→20℃ | 0 | 0 |
| 注: *温度对比方案以“/”后为参照基准 | ||
用AB SCIEX 5800 MALDI-TOF/TOFTM MS质谱对6个差异表达蛋白质进行鉴定, 均为可信的蛋白且其表达变化如表 6所示。注释到4个可信蛋白。其中2个蛋白质(点135, 点165)注释为transport, 1个蛋白质(点158)注释为cellular protein modification process, 1个蛋白质(点292)被注释到generation of precursor metabolites and energy。另外两个蛋白未得到有效注释。
| 胶点 | NCBIb | 蛋白名称 |
| 132 | 496523478 | TonB-linked outer membrane protein |
| 158 | 77024185 | L-cystine transport system permease protein TcyB |
| 165 | 505821775 | Photosystem Ⅱ D2 apoprotein (Fragment) |
| 222 | 519111539 | Hypothetical protein |
| 270 | 548247975 | Hypothetical protein |
| 292 | 422034799 | Chloroplast light harvesting protein isoform 8 |
用2D-DIGE染料标记细胞表面膜蛋白技术已经被应用于研究人类细胞(Mayrhofer et al, 2006; Kadiu et al, 2009)、浮游藻类(Li et al, 2012)以及细菌(Anaya et al, 2007)等。这种方法克服了以往生物素和同位素标记方法(Palenik et al, 1995)导致的膜蛋白疏水性增加和影响后续蛋白质鉴定等问题, 在生命科学研究领域具有较好的应用前景。
本文运用蛋白质荧光标记技术、顺序提取法和凝胶电泳分离技术对米氏凯伦藻细胞表面膜蛋白质进行了研究。从凝胶电泳的荧光图可以看出, 三步提取方法中第一步用trizol试剂能将细胞内大部分蛋白提取出来, 说明大部分是未被荧光染料标记的胞内蛋白。实验中所得到胶图的荧光点只有几十个, 根据基因组开放阅读框预测的膜蛋白可以占到其整个基于组的20%—30% (Wallin et al, 1998), 而胶图上的荧光点只有几十个, 这可能是这些膜蛋白丰度较低且被胞内高丰度蛋白掩盖(Mitra et al, 2007), 因而即使基于高灵敏度的荧光染料标记蛋白法也很难被检测到(Marouga et al, 2005)。第二步及第三步的荧光点和第一步的荧光点基本上重合, 表明第一步包含了后续两步的大部分膜蛋白。在上样量相同的情况下, 第二、三两步凝胶电泳所得到的荧光点很少, 可能是由于膜蛋白比较难溶, 用基于凝胶的方法分离, 在第一向等电聚焦时不能完全溶解, 导致蛋白质的丢失(Luche et al, 2003)。
对已鉴定到的蛋白进行细胞成分GO注释, 发现了与底物转运有关的ABC transporter substrate -binding protein。ABC转运蛋白作为细胞膜蛋白非常重要的组成成分, 其蛋白质超家族是目前已知的从原核生物到人类最古老的蛋白家族之一(Verrier et al, 2008), 在大肠杆菌中ABC转运蛋白占整个基因组的5%(Higgins et al, 2004)。所有ABC转运蛋白都含有非常保守的跨膜结构域和ATP结合结构域, 具有执行物质运输、胁迫响应和细胞间通讯等多种功能(Rea, 2007)。本研究还鉴定到了藻细胞表面heat shock protein 70 (HSP70)分子伴侣蛋白。Li等(2012)在利用2D-DIGE荧光染料标记链状亚历山大藻细胞表面膜蛋白时, 同样鉴定到HSP70c和HSP70两个热休克蛋白。研究表明, HSP70可以存在于细胞表面(Kornilovs et al, 2002; Jang et al, 2003; Shin et al, 2003)。在氧化胁迫和热休克情况下, 细胞中的HSP70可以稳定溶酶体膜结构的稳定性, 阻止溶酶体的裂解(Nylandsted et al, 2004), 通过阻止细胞色素c和组织蛋白酶的释放去控制细胞凋亡过程(Bivik et al, 2007)。然而HSP70可能并非通过典型的分泌途径到达细胞膜表面(李成, 2011), 而具体是如何转移到细胞膜上未知。efflux transporter, RND family, MFP subunit是跨膜结构的协同转运蛋白超家族(MFS transporter)以及相关的nodulation receptor kinase、MFS transporter这类蛋白质是通过膜内外底物浓度不同来转运的次级蛋白转运超家族(Maloney, 1990, 1992; Holmes et al, 2012)。研究中还鉴定到两个membrane protein, 这可能是由于利用de novo从头测序结合MS-blast同源搜索只是鉴定到符合膜蛋白结构的部分肽段, 具体是何种蛋白质还不能确定。这些结果表明米氏凯伦藻的细胞膜具有较强的物质转运能力和抗逆能力。
本研究中, 除了鉴定到与表面膜相关的蛋白质以外, 还鉴定到一些在叶绿体类囊体上参与光合作用的捕光蛋白的不同亚基。一般认为捕光蛋白存在细胞内, 然而在本研究中被大量鉴定, 这可能一方面是它占类囊体膜上叶绿素总量的40%—60%, 一方面在利用荧光染料进行细胞表面标记时, 由于米氏凯伦藻特殊的形态结构, 叶绿体与细胞膜紧密接触以及鞭毛与细胞膜相连导致捕光蛋白和细胞骨架蛋白被荧光染料标记。米氏凯伦藻的捕光蛋白存在跨膜特性并锚定在类囊体上, 用来捕捉更多的光能用于光合作用或其他生理功能, 这可能是米氏凯伦藻对光利用的一种特殊机制。同时鉴定到组成光合系统Ⅱ的photosystemⅡd2 apoprotein和chlorophyll a/b-binding protein isoform 4 /actin-related protein - arp5p, d2蛋白质具有5个跨膜区域, 这两个蛋白被荧光染料标记上的原因尚不清楚。
此外, 本研究中还鉴定到protein hum-10、Traf3-interacting protein 1和Rhodanese-like domain- containing protein以及一些假想蛋白质, 它们的功能尚不明确, 这些蛋白质可能是目前未知的细胞表面膜蛋白质, 有待后续继续深入研究。
3.2 米氏凯伦藻对温度变化的响应米氏凯伦藻在20℃、16℃和12℃三个温度经长期驯化后, 20℃条件下第2天细胞即进入快速生长期, 而16℃和12℃条件下细胞生长缓慢。在第4天进行快速变温后, 12℃→20℃和12℃→16℃两组的米氏凯伦藻细胞密度短期迅速减少, PSII最大光化学量子产量(Fv/Fm)也迅速降低, 这可能与短时间内温度骤变导致米氏凯伦藻细胞生理不适有关, 这与现场海水温度逐渐升高(郭伟其等, 2005)的情况有所不同。但随后两组变温的细胞逐渐恢复并适应新的温度环境, 表明米氏凯伦藻对于温度变化的适应能力较强。因此我们猜想:在环境温度缓慢升高时, 米氏凯伦藻生长速率较慢而无法竞争过硅藻等其他藻类, 不利于其形成藻华; 但当环境温度快速升高时, 米氏凯伦藻较其他藻类适应性更快, 可以率先占据营养盐等资源, 从而有利于其藻华的形成。
通过比较20℃、16℃和12℃三个温度梯度以及12℃→20℃, 12℃→16℃膜蛋白质组的变化, 发现了6个蛋白质的表达发生显著变化。在16℃和12℃之间, 膜蛋白质组水平上两者之间没有显著差异。这可能是由于12℃和16℃与米氏凯伦藻最适生长温度范围20—24℃相差较远, 低温抑制了细胞活力, 导致两者的差异较小。
TonB-linked outer membrane protein (点132)是一种重要的质子驱动膜转运蛋白, 主要参与环境中铁(Lundrigan et al, 1986; Pressler et al, 1988; Koedding et al, 2004)以及维生素B12 (Heller et al, 1985)等底物的跨膜转运, 在细胞各种底物的转运方面起着重要作用, 当米氏凯伦藻的培养温度从12℃转运到16℃和20℃时, 细胞内TonB-linked outer membrane protein的表达下调, 说明在低温条件下细胞可能需要更多转运载体, 维持基本的物质需要。L-cystine transport system permease protein tcyb (点158)被定位在细胞质膜上的多跨膜的转运胱氨基酸的膜蛋白, 属于ABC transporter permease, 它广泛分布在细胞膜上, 通过水解ATP释放能量从而将底物从膜低浓度一侧转运到高浓度一侧(Liu et al, 1999)。在12℃和16℃低温培养条件下该蛋白显著高表达, 可能是补偿由于温度降低而导致蛋白质活性降低的影响(Varkey et al, 2015), 同时也可保持细胞内正常的物质代谢以及提高胞内渗透压抵御低温。此外, 还鉴定到类囊体上的光捕捉蛋白质(点292)和photosystem Ⅱ d2 apoprotein (点165), 这可能是是在利用荧光进行细胞表面标记时, 由于米氏凯伦藻较为特殊的细胞形态结构, 即叶绿体与细胞膜紧密接触有关。光捕捉蛋白质在培养温度16℃与12℃之间没有变化, 在20℃条件下表达上调, 而photosystem Ⅱ d2 apoprotein与之相反, 这表明低温胁迫导致细胞基本代谢功能的转变(Cui et al, 2005), 同时也表明米氏凯伦藻光系统Ⅰ对低温较为敏感, 细胞通过调整光系统Ⅰ和光系统Ⅱ之间的电子转移平衡以避免细胞受损; 而当温度升高时, 米氏凯伦藻需要较多的捕光蛋白以获取更多能量维持细胞的快速增长。由于米氏凯伦藻基因和蛋白质数据库的不完善以及关于膜蛋白信息的缺乏, 未能得到hypothetical protein (点222和270)的具体功能信息。
4 结论本文建立了基于荧光染料的细胞表面膜蛋白标记方法, 为甲藻细胞表面膜蛋白研究提供了技术支撑。米氏凯伦藻表面膜蛋白主要为转运蛋白、HSP70家族蛋白和光合作用相关蛋白等, 这表明米氏凯伦藻具有较强的物质转运能力、光合作用能力和抗胁迫能力, 这些膜蛋白可能在米氏凯伦藻适应环境变动方面具有重要作用。
低温会抑制米氏凯伦藻细胞的生长, 但迅速升温可刺激细胞生长, 并且米氏凯伦藻可快速适应温度的变化, 这可能是米氏凯伦藻藻华形成的一个重要原因。注释到的4个与环境温度变化相关的细胞表面膜蛋白主要为转运和捕光蛋白, 表明物质转运和光合作用对于米氏凯伦藻响应环境的温度变化有着重要作用。
目前由于米氏凯伦藻基因组数据库的缺乏以及膜蛋白提取方法方面面临的挑战, 本文仅鉴定到数量极少的膜蛋白, 后续应加强米氏凯伦藻基因组和转录组的研究, 完善膜蛋白提取方法, 以获得更多的膜蛋白信息, 提升我们对米氏凯伦藻基本生命活动规律以及赤潮形成机制的认识。
李成, 2011.海洋甲藻蛋白质组学研究: 以亚历山大藻为例.厦门: 厦门大学博士学位论文, 36-96 https://dspace.xmu.edu.cn/handle/2288/53021
|
沈盎绿, 2014.东海原甲藻和米氏凯伦藻种间竞争对海水温度变化的响应机制.上海: 华东师范大学博士学位论文, 102-113 http://cdmd.cnki.com.cn/Article/CDMD-10269-1015543010.htm
|
张传松, 2008.长江口及邻近海域赤潮生消过程特征及其营养盐效应分析.青岛: 中国海洋大学博士学位论文, 96-125 http://cdmd.cnki.com.cn/article/cdmd-10423-2009162527.htm
|
郭伟其, 沙伟, 沈红梅, 等. 2005. 东海沿岸海水表层温度的变化特征及变化趋势. 海洋学报, 27(5): 1-8 |
Anaya C C, Church N, Lewis J P, 2007. Detection and identification of bacterial cell surface proteins by fluorescent labeling. Proteomics, 7(2): 215-219 DOI:10.1002/(ISSN)1615-9861 |
Bertomeu T, Hastings J W, Morse D, 2003. Vectorial labeling of dinoflagellate cell surface proteins. Journal of Phycology, 39(6): 1254-1260 DOI:10.1111/j.0022-3646.2003.03-066.x |
Bivik C, Rosdahl I, Öllinger K, 2007. Hsp70 protects against UVB induced apoptosis by preventing release of cathepsins and cytochrome c in human melanocytes. Carcinogenesis, 28(3): 537-544 |
Cui S X, Huang F, Wang J et al, 2005. A proteomic analysis of cold stress responses in rice seedlings. Proteomics, 5(12): 3162-3172 DOI:10.1002/(ISSN)1615-9861 |
Dančík V, Addona T A, Clauser K R et al, 1999. De novo peptide sequencing via tandem mass spectrometry. Journal of Computational Biology, 6(3-4): 327-342 |
Heller K, Kadner R J, 1985. Nucleotide sequence of the gene for the vitamin B12 receptor protein in the outer membrane of Escherichia coli. Journal of Bacteriology, 161(3): 904-908 |
Hicke L, Dunn R, 2003. Regulation of membrane protein transport by ubiquitin and ubiquitin-binding proteins. Annual Review of Cell and Developmental Biology, 19(1): 141-172 DOI:10.1146/annurev.cellbio.19.110701.154617 |
Higgins C F, Linton K J, 2004. The ATP switch model for ABC transporters. Nature Structural & Molecular Biology, 11(10): 918-926 |
Hockin N L, Mock T, Mulholland F et al, 2012. The response of diatom central carbon metabolism to nitrogen starvation is different from that of green algae and higher plants. Plant Physiology, 158(1): 299-312 DOI:10.1104/pp.111.184333 |
Holmes A R, Keniya M V, Ivnitski-Steele I et al, 2012. The monoamine oxidase A inhibitor clorgyline is a broad-spectrum inhibitor of fungal ABC and MFS transporter efflux pump activities which reverses the azole resistance of Candida albicans and Candida glabrata clinical isolates. Antimicrobial Agents and Chemotherapy, 56(3): 1508-1515 DOI:10.1128/AAC.05706-11 |
Jang J H, Hanash S, 2003. Profiling of the cell surface proteome. Proteomics, 3(10): 1947-1954 DOI:10.1002/(ISSN)1615-9861 |
Kadiu I, Wang T, Schlautman J D et al, 2009. HIV-1 transforms the monocyte plasma membrane proteome. Cellular Immunology, 258(1): 44-58 |
Keller M D, Selvin R C, Claus W et al, 1987. Media for the culture of oceanic ultraphytoplankton. Journal of Phycology, 23(4): 633-638 |
Koedding J, Howard P, Kaufmann L et al, 2004. Dimerization of TonB is not essential for its binding to the outer membrane siderophore receptor FhuA of Escherichia coli. Journal of Biological Chemistry, 279(11): 9978-9986 |
Kornilovs'ka I, Nilsson I, Utt M et al, 2002. Immunogenic proteins of Helicobacter pullorum, Helicobacter bilis and Helicobacter hepaticus identified by two‐dimensional gel electrophoresis and immunoblotting. Proteomics, 2(6): 775-783 DOI:10.1002/1615-9861(200206)2:6<>1.0.CO;2-O |
Li C, Wang D Z, Dong H P et al, 2012. Proteomics of a toxic dinoflagellate Alexandrium catenella DH01: detection and identification of cell surface proteins using fluorescent labeling. Chinese Science Bulletin, 57(25): 3320-3327 DOI:10.1007/s11434-012-5156-5 |
Liu P Q, Liu C E, Ames G F L, 1999. Modulation of ATPase activity by physical disengagement of the ATP-binding domains of an ABC transporter, the histidine permease. Journal of Biological Chemistry, 274(26): 18310-18318 DOI:10.1074/jbc.274.26.18310 |
Luche S, Santoni V, Rabilloud T, 2003. Evaluation of nonionic and zwitterionic detergents as membrane protein solubilizers in two‐dimensional electrophoresis. Proteomics, 3(3): 249-253 DOI:10.1002/pmic.200390037 |
Lundrigan M D, Kadner R J, 1986. Nucleotide sequence of the gene for the ferrienterochelin receptor FepA in Escherichia coli. Homology among outer membrane receptors that interact with TonB. Journal of Biological Chemistry, 261(23): 10797-10801 |
Maloney P C, 1990. Microbes and membrane biology. FEMS Microbiology Letters, 87(1-2): 91-102 DOI:10.1111/fml.1990.87.issue-1-2 |
Maloney P C, 1992. The molecular and cell biology of anion transport by bacteria. BioEssays, 14(11): 757-762 DOI:10.1002/(ISSN)1521-1878 |
Marouga R, David S, Hawkins E, 2005. The development of the DIGE system: 2D fluorescence difference gel analysis technology. Analytical and Bioanalytical Chemistry, 382(3): 669-678 DOI:10.1007/s00216-005-3126-3 |
Mayrhofer C, Krieger S, Allmaier G et al, 2006. DIGE compatible labelling of surface proteins on vital cells in vitro and in vivo. Proteomics, 6(2): 579-585 DOI:10.1002/(ISSN)1615-9861 |
Mitra S K, Gantt J A, Ruby J F et al, 2007. Clouse and Michael B.Goshe. Membrane proteomic analysis of Arabidopsis thaliana using alternative solubilization techniques. Journal of Proteome Research, 6(5): 1933-1950 |
Molloy M P, Herbert B R, Walsh B J et al, 1998. Extraction of membrane proteins by differential solubilization for separation using two‐dimensional gel electrophoresis. Electrophoresis, 19(5): 837-844 DOI:10.1002/(ISSN)1522-2683 |
Musil L S, Cunningham B A, Edelman G M et al, 1990. Differential phosphorylation of the gap junction protein connexin43 in junctional communication-competent and -deficient cell lines. The Journal of Cell Biology, 111(5): 2077-2088 DOI:10.1083/jcb.111.5.2077 |
Nylandsted J, Gyrd-Hansen M, Danielewicz A et al, 2004. Heat shock protein 70 promotes cell survival by inhibiting lysosomal membrane permeabilization. The Journal of Experimental Medicine, 200(4): 425-435 DOI:10.1084/jem.20040531 |
Palenik B, Koke J A, 1995. Characterization of a nitrogen- regulated protein identified by cell surface biotinylation of a marine phytoplankton. Applied and Environmental Microbiology, 61(9): 3311-3315 |
Pressler U, Staudenmaier H, Zimmermann L et al, 1988. Genetics of the iron dicitrate transport system of Escherichia coli. Journal of Bacteriology, 170(6): 2716-2724 DOI:10.1128/jb.170.6.2716-2724.1988 |
Rea P A, 2007. Plant ATP-binding cassette transporters. Annual Review of Plant Biology, 58: 347-375 |
Shin B K, Wang H, Yim A M et al, 2003. Global profiling of the cell surface proteome of cancer cells uncovers an abundance of proteins with chaperone function. Journal of Biological Chemistry, 278(9): 7607-7616 DOI:10.1074/jbc.M210455200 |
Snyders S, Kohorn B D, 1999. TAKs, thylakoid membrane protein kinases associated with energy transduction. Journal of Biological Chemistry, 274(14): 9137-9140 DOI:10.1074/jbc.274.14.9137 |
Toulza E, Shin M S, Blanc G et al, 2010. Gene expression in proliferating cells of the dinoflagellate Alexandrium catenella (Dinophyceae). Applied and Environmental Microbiology, 76(13): 4521-4529 DOI:10.1128/AEM.02345-09 |
Varkey D, Mazard S, Ostrowski M et al, 2015. Effects of low temperature on tropical and temperate isolates of marine Synechococcus. The ISME Journal, 10(5): 1252-1263 |
Verrier P J, Bird D, Burla B et al, 2008. Plant ABC proteins–a unified nomenclature and updated inventory. Trends in Plant Science, 13(4): 151-159 DOI:10.1016/j.tplants.2008.02.001 |
Wallin E, von Heijne G, 1998. Genome‐wide analysis of integral membrane proteins from eubacterial, archaean, and eukaryotic organisms. Protein Science, 7(4): 1029-1038 |
Wang D Z, Li C, Xie Z X et al, 2011. Homology-driven proteomics of dinoflagellates with unsequenced genomes using MALDI-TOF/TOF and automated De novo sequencing. Evidence-Based Complementary and Alternative Medicine: 471020 |
Wang D Z, Zhang Y J, Zhang S F et al, 2013. Quantitative proteomic analysis of cell cycle of the dinoflagellate Prorocentrum donghaiense (Dinophyceae). PLoS One, 8(5): e63659 DOI:10.1371/journal.pone.0063659 |
 2019, Vol. 50
2019, Vol. 50