中国海洋湖沼学会主办。
文章信息
- 崔宗梅, 黄津伟, 王海艳, 胡利莎, 刘玉盟. 2018.
- CUI Zong-Mei, HUANG Jin-Wei, WANG Hai-Yan, HU Li-Sha, LIU Yu-Meng. 2018.
- 广东雷州半岛东部沿岸潮间带常见牡蛎的种类及其分布
- CLASSIFICATION AND DISTRIBUTION OF OYSTERS IN EASTERN COAST OF LEIZHOU PENINSULA, CHINA
- 海洋与湖沼, 49(6): 1350-1357
- Oceanologia et Limnologia Sinica, 49(6): 1350-1357.
- http://dx.doi.org/10.11693/hyhz20180300054
-
文章历史
- 收稿日期:2018-03-15
- 收修改稿日期:2018-06-22
2. 中国科学院大学 北京 100049;
3. 天津师范大学 天津 300387
2. University of Chinese Academy of Sciences, Beijing 100049, China;
3. Tianjin Normal University, Tianjin 300387, China
牡蛎属于软体动物门、双壳纲、牡蛎目、牡蛎总科、牡蛎科, 分布区域面积广, 而且肉味鲜美, 具有很高的经济价值。由于牡蛎是营固着生活的软体动物, 栖息环境复杂, 贝壳形态随栖息环境变化极大, 单纯依靠贝壳的形态特征进行种类的分类鉴定容易导致分类的混乱, 如Harry(1985)记录世界上现存的100多种牡蛎中大约有近2/3是同物异名。随着分子生物学的发展和应用, 许多分类学者结合牡蛎的形态特征和分子标记对牡蛎的种类和系统发育进行了分析, 解决了牡蛎的很多分类争议。王海艳等(2004)结合形态和分子生物学的方法对我国沿海常见牡蛎的种类和分布进行了研究, 开发了巨蛎属(Crassotrea)常见牡蛎的种特异性标记(Wang et al, 2008), 可以快速对种类鉴定, 并把福建牡蛎(C. gigas angulata)订为长牡蛎(C. gigas gigas)的亚种(Wang et al, 2010)。Lam等(2003, 2004)通过线粒体基因COI和16S rRNA发现了牡蛎新种香港牡蛎(C. hongkongensis), 并根据形态特征和COI基因序列鉴定, 描述了从香港采集到的12种牡蛎的形态特征、栖息环境及其分布。Wang等(2004)发现香港牡蛎不只分布在香港, 在我国南方沿海有大量分布, 是当地重要经济贝类, 并把当地渔民称为的“白肉”定为香港牡蛎, “红肉”定为近江牡蛎(C. ariakensis)。
雷州半岛沿海属热带季风气候, 具有优越的地理环境和自然条件, 生物资源丰富。《广东潮间带生物调查报告》中记录了牡蛎科(Ostreidae)16种, 其中粤西中西部有咬齿牡蛎(Saccostrea mordax)和近江牡蛎(C. ariakensis)两种。韩维栋等于2002年记录了雷州半岛红树林区牡蛎科(Ostreidae)的4种牡蛎, 近江牡蛎、S. glomerata、猫爪牡蛎(Talonostrea talonata)以及一种巨蛎属sp, 其中仅巨蛎属sp牡蛎分布在雷州半岛东部沿岸(韩维栋等, 2009)。梁超愉等(2005)对雷州半岛红树林潮间带底栖生物种类的组成调查中, 发现了棘刺牡蛎(S. echinata)、咬齿牡蛎、近江牡蛎和一种巨蛎属sp牡蛎4种, 其中仅棘刺牡蛎分布在东部沿岸。由于调查者没有进行系统的形态和分子生物学分析, 可能会有同物异名或异物同名的现象发生。
为了弥补雷州半岛东部沿岸牡蛎资源调查研究的匮乏, 我们采集了该地区大量的野生牡蛎样品, 并对样品进行了形态及分子生物学鉴定, 以期明确我国雷州半岛东部沿岸常见牡蛎的种类及分布。
1 材料与方法 1.1 样品采集与保存本次牡蛎调查区域从北至南包括广东省茂名市、湛江市、雷州市潮间带共11个站位。样品采集地点见图 1。采样时不分样品大小, 采集形态差别较大个体, 样品保存于95%的乙醇中。
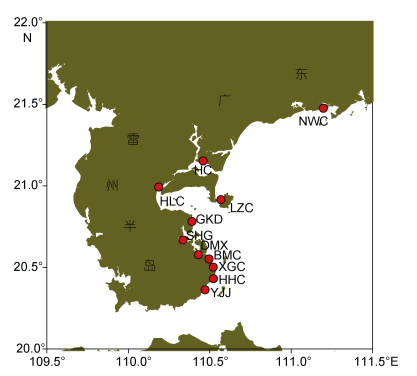 |
| 图 1 本次实验牡蛎样品采集地点 Fig. 1 Location of oysters sampling 注: NWC:茂名市博贺镇那尾村; HC:湛江市南三岛湖村; HLC:雷州市沈塘镇海岚村; LZC:湛江市硇洲岛六竹村; GKD:雷州市东里镇沟口东村; SHG:湛江市和安镇松树港; DMX:湛江市锦和镇东门下村; BMC:湛江市外罗镇白茅村; XGC:湛江市下洋镇下港村; HHC:湛江市前山镇后海村; YJJ:湛江市前山镇盐井角 |
观察记录牡蛎贝壳的各部分特征, 包括壳型、壳上鳞片情况、壳表面的颜色、放射肋分布、韧带槽形状及大小、壳内面颜色、闭壳肌痕颜色及形状等。通过形态特征对牡蛎进行初步鉴定。
1.3 基因组DNA的提取和目的基因PCR扩增利用Wang等(2008)开发的巨蛎属牡蛎的种特异性标记, 通过多重PCR的方法快速将巨蛎属牡蛎鉴定到种, 对无法通过该方法鉴定到种的巨蛎属及其他属样品进行细胞色素氧化酶Ⅰ (cytochrome oxidase Ⅰ, COI)基因扩增, 并结合系统发育分析的方法, 确定每个样品的种名。
从牡蛎闭壳肌中取出20—30g闭壳肌组织, 将组织中的酒精晾干之后使用DneasyTM Tissue Kit (Tiangen)试剂盒, 提取牡蛎基因组DNA。通过聚合酶链式反应(polymerase chain reaction, PCR)扩增巨蛎属牡蛎的特异性片段和COI基因片段, 扩增使用引物见表 1(Folmer et al, 1994; Wang et al, 2008)。多重PCR反应体系为25μL, 包括8μL ddH2O, 12.5μL 2×TSINGKE Master Mix, 0.5μL COI引物, 0.4μL特异性引物和1.5μL DNA模板。COI基因PCR反应体系为25μL, 包括9.5μL ddH2O, 12.5μL 2×TSINGKE Master Mix, 0.5μL引物和2μL DNA模板。使用BIO-RAD thermal cycler进行PCR, 循环参数如下:预变性95℃加热2min, 变性95℃加热1min, 引物退火48—51℃ 1min, 引物延伸72℃进行1min, 循环数35, 最后72℃延伸10min。多重PCR获得的产物用1.5%的琼脂糖凝胶电泳进行检测, 通过PCR产物的长度将样品鉴定到种。
| 引物名称 | 种的特异性 | 引物序列 | 长度(bp) |
| COL 1490 (forward) | 所有种 | 5′-GGTCACAAATCATAAAGATATTGG-3′ | |
| COCar183r | 近江牡蛎C. ariakensis | 5′-AAAAAAGATTATAACTAATGCATGTCG(T)G-3′ | 183 |
| COCan222r | 福建牡蛎C. gigas angulata | 5′-AGTTACCAAACCCCCCAATTATCAG(C)G-3′ | 222 |
| COCgi269r | 长牡蛎C. gigas gigas | 5′-TCGAGGAAATTGCATGTCTGCTACA(T)A-3′ | 269 |
| COChk387r | 香港牡蛎C. hongkongensis | 5′-GGAGTAAGTGGATAAGGGTGGATAG-3′ | 387 |
| COCsi546r | 熊本牡蛎C. sikamea | 5′-AAGTAACCTTAATAGATCAGGGAAC(A)C-3′ | 546 |
| COH 2198 (reverse) | 所有种 | 5'-TAAACTTCAGGGTGACCAAAAAATCA-3' | 697 |
多重PCR未鉴定出来的种, 进行COI基因扩增并测序。本次实验获得114个牡蛎样品的COI片段, 表 2为从NCBI下载的相关序列。使用MEGAv.7.0.26 (Kumar et al, 2016)进行序列比对, 单倍型的计算使用DNASPv.5.10.01 (Librado et al, 2009)。获得的单倍型和从NCBI上下载的相关序列用于系统发育分析。应用ModelTest 3.7 (Posada et al, 1998)基于Akaike Information Criterion (AIC)为COI基因片段选择核苷酸最佳替换模型, 并估计相关参数, 用于BI(Bayesian inference)和ML(Maximum likelihood tree)方法建树。
| 物种名称 | 序列号 | 样品地点 | 参考文献 | 备注 |
| C. sikamea | FJ841966 | Shatian, China | Wu et al, 2010 | complete mitogenome |
| C. angulata | FJ841965 | Dianbai, China | Wu et al, 2010 | complete mitogenome |
| Saccostrea sp. XL-13 | KU947216 | Daya Bay, China | non-mordax ineage F | |
| Saccostrea sp. LJ-13 | KU947207 | Daya Bay, China | non-mordax lineage B | |
| Ostrea sp. STH-2012 | JQ027291 | PT, Taiwan, China | ||
| Striostrea prismatica | KT317606 | Baja California Sur, Mexico | Raith, 2016 |
利用软件MrBayes3.2.6(Ronquist et al, 2012)构建贝叶斯树, 以随机树为起始树运行200万代, 每10000代抽样一次。使用MEGA v.7.0.26构建ML树, 参数设置使用Model Test 3.7的结果进行设置。使用MEGAv.7.0.26计算牡蛎物种间和种内的Kimura 2-parameter遗传距离(Kimura, 1980)。
2 结果与分析 2.1 形态特征及多重PCR鉴定、COI基因扩增本次采集的牡蛎经过形态特征和多重PCR鉴定, 可以确定到种的巨蛎属牡蛎有两种。从琼脂糖凝胶电泳图(图 2)中可以看出, 9个样品中1、2、3、6号样品的特异性条带与对照组福建牡蛎(Can)的特异性条带位置相同, 再结合形态特征, 将这4个样品定为福建牡蛎。对照熊本牡蛎(Csi)特异性条带与4、5、7、9号样品的特异性条带位置相同, 再结合形态特征, 将这4个样品定为熊本牡蛎。通过多重PCR的方法本次实验的363个牡蛎样品中鉴定出137个熊本牡蛎, 101个福建牡蛎。对形态鉴定不属于巨砺属的牡蛎, 如图 2中8号样品属于Ostrea属, 只扩增出COI基因条带, 随后将其进行COI基因测序, 共对114个样品的COI基因进行测序并进行系统发育分析。
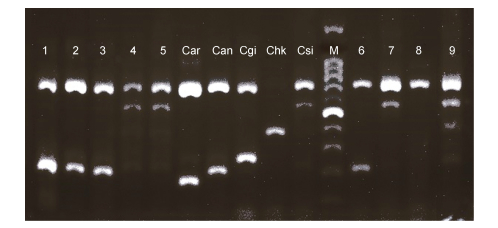 |
| 图 2 多重PCR(7种引物)琼脂糖凝胶电泳图 Fig. 2 Multiplex species-specific PCR with 7 primers from the COI gene 注: 1—9:本次实验的9个样品; M: 100bp DNA ladder, 最亮的条带为500bp; Car:近江牡蛎C. ariakensis; Can:福建牡蛎C. gigas angulata; Cgi:长牡蛎C. gigas gigas; Chk:香港牡蛎C. hongkongensis; Csi:熊本牡蛎C. sikamea |
 |
| 图 3 雷州半岛东部沿岸采集到的常见牡蛎种类的贝壳形态 Fig. 3 The shell morphology of common oysters collected from the eastern coast of Leizhou Peninsula, China 注: A—B.熊本牡蛎C. sikamea; C—D.福建牡蛎C. gigas angulata; E—F.团聚牡蛎Saccostrea malabonensis; G—H.棘刺牡蛎S. echinata |
个体偏小, 肉质细腻, 其软体部相对较大, 具有较高的经济价值。贝壳呈长形或近长形, 扁平。壳表面放射肋不明显, 常呈青色, 带有褐色花纹。壳质薄, 内面白色, 闭壳肌痕黄褐色或白色。壳边缘处有褶皱。
又名葡萄牙牡蛎, Wang等(2010)通过形态比较、地理分布和分子生物学分析将此种定为长牡蛎的亚种。壳近长形或椭圆形, 附着面大, 右壳较平, 有明显放射肋。壳面常呈褐色, 生长线呈鳞片状, 壳内面白色, 闭壳肌痕褐色。
2.2 系统发育分析结果使用DnaSPv.5.10.01计算本次实验所获得的114条COI序列的单倍型, 共获得52个单倍型。采用MEGAv.7.0.26构建的最大似然树和使用MrBayes3.2.6构建的贝叶斯树拓扑结构相同, 最大似然树如图 4, 以Striostrea prismatica作为外群。
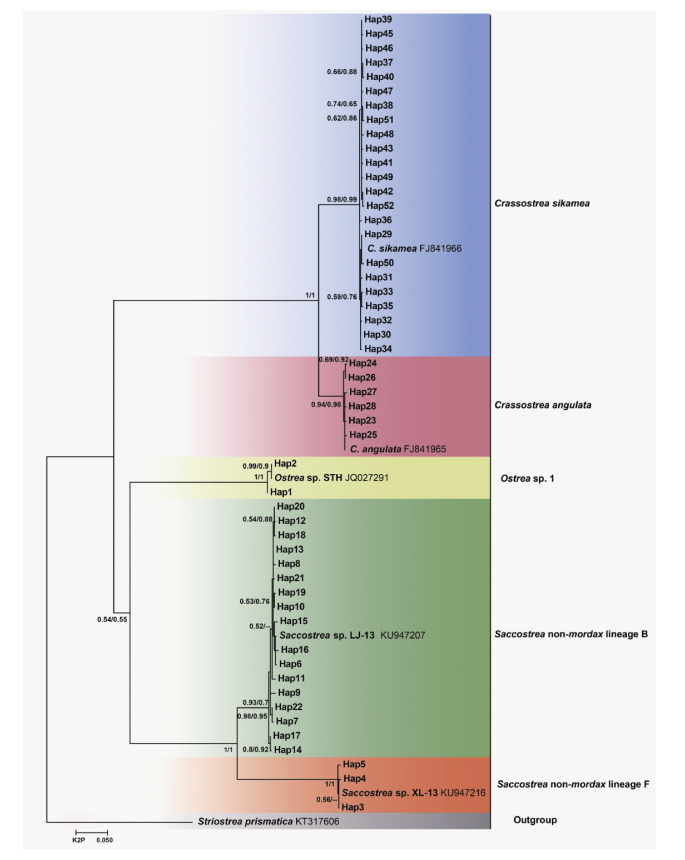 |
| 图 4 基于COI基因使用最大似然法(ML)构建的系统进化树 Fig. 4 The maximum likelihood tree (ML) based on COI gene 注:支上数字表示Bootstrap value/ Posterior probabilities, --表示Bootstrap value或Posterior probabilities的值小于0.5 |
由图 4可以看出, 有23个单倍型与熊本牡蛎C. sikamea(FJ841966)聚为一支, 且贝叶斯后验概率(Posterior probabilities)为0.99, 表现出很高的支持率, 结合熊本牡蛎的形态特征, 将它们定为熊本牡蛎。单倍型23—28与福建牡蛎C. gigas angulata(FJ841965)聚在一起, 且支持率较高(Posterior probabilities= 0.94, Bootstrap value= 0.98), 结合形态特征定为福建牡蛎。单倍型1、2与牡蛎属sp(JQ027291)聚在一起, 贝叶斯后验概率为1, 暂定为Ostrea sp.1, 本次采样仅在博贺镇采集到少量样品。
两个分支属于小蛎属牡蛎, Lam等(2006)利用线粒体基因16S rRNA对印度-西太平洋的小蛎属牡蛎进行了系统分析, 将其分为两个大的分支, 分别为僧帽牡蛎复合体(Saccostrea cucullata superspecies)和咬齿牡蛎复合体(Saccostrea mordax superspecies), 这两个分支分别包括9个和2个支系, S. cucullata-A、B、C、D、E、F、G、S. kegaki、S. glomerata和S. mordax-A、B。Sekino等(2016)通过僧帽牡蛎(S. cucullata)的模式标本产地及描述认为僧帽牡蛎不能代表印度-西太平洋的小蛎属牡蛎, 建议用non-mordax分支代替僧帽牡蛎复合体(Saccostrea cucullata superspecies), 同时又提出三个分支, non-mordax lineage H、I、J。本次实验中单倍型3—5和Saccostrea sp. XL-13(KU9 47216)聚为一支(Bootstrap value=1, Posterior probabilities=1), 该序列之后被证明属于Saccostrea non-mordax lineage F分支。该分支样品近长圆形, 壳质薄, 左壳深凹、右壳较平, 壳面褐色或黑褐色, 放射肋不明显, 壳边缘具鳞片, 常多个个体聚在一起生长, 有些个体附着于螺上生长, 右壳边缘有均匀褶皱, 壳顶腔靠近前侧。其形态特征与团聚牡蛎的形态特征相似, 因此定为团聚牡蛎(S. malabonensis), 图 3E—F。
单倍型6—22与Saccostrea sp. LJ-13(KU947207)聚在一起, 而且有较高的支持率, 该序列之后被证明属于Saccostrea non-mordax lineage B分支。通过形态观察, 单倍型6—22样品多圆形或椭圆形, 左壳常以整面固着在岩石上, 壳表黑色或紫褐色, 生长线细密呈鳞片状, 鳞片边缘卷曲成管状棘刺, 棘刺的多少强弱与个体所处环境有关。壳内颜色多变, 右壳内边缘有明显的单行小齿, 分布于整个或部分壳边缘, 闭壳肌痕形状不规则。其形态特征与我国南方沿海潮间带最常见种类棘刺牡蛎的形态特征相符, 因此单倍型6—22定为棘刺牡蛎, 图 3G—H。梁超愉等(2005)在雷州半岛东部沿岸红树林潮间带也发现了棘刺牡蛎, 与本文结果相同。
2.3 遗传距离分析使用MEGA v.7.0.26计算根据系统进化树得到的5个分支之间的Kimura 2-parameter平均遗传距离, 结果如表 3所示。
| 牡蛎种类 | 1 | 2 | 3 | 4 | 5 |
| 1. Ostrea sp.1 | |||||
| 2. Saccostrea malabonensis | 0.288 | ||||
| 3. Saccostrea echinata | 0.256 | 0.176 | |||
| 4. Crassostrea gigas angulata | 0.285 | 0.304 | 0.297 | ||
| 5. Crassostrea sikamea | 0.309 | 0.321 | 0.280 | 0.100 | |
| 6. Striostrea prismatica | 0.294 | 0.319 | 0.283 | 0.319 | 0.309 |
由表 3可知, 平均遗传距离的范围为10.0%—32.1%, 福建牡蛎和熊本牡蛎之间的遗传距离最小为10.0%, 小蛎属两个种之间的平均遗传距离为17.6%。以上结果说明, 雷州半岛东部沿岸采到的牡蛎样品可以分为3属5个种, 分别为熊本牡蛎、福建牡蛎, 牡蛎属未定种(Ostrea sp.1), 棘刺牡蛎和团聚牡蛎(S. malabonensis)。
3 讨论 3.1 雷州半岛东部沿岸潮间带常见巨蛎属Crassostrea牡蛎(1) 熊本牡蛎C. sikamea
宋忠魁等(2010)利用多重PCR技术鉴定了广西茅尾海3种常见牡蛎, 其中俗称的“蚝蛎”证明是熊本牡蛎。Wang等(2013)通过基于线粒体基因的系统发育分析, 确定了熊本牡蛎的分类地位以及在中国沿海的分布, 从江苏到海南都有分布。本次在潮间带采样发现大量熊本牡蛎, 为雷州半岛东部沿岸牡蛎优势种。其主要分布于硇洲岛和沈塘镇, 张智瀛(2014)对雷州半岛海域的溶解氧及盐度进行了调查和研究, 显示硇洲岛和沈塘镇附近海域溶解氧含量较高, 盐度较低, 因此熊本牡蛎应该更适合在低盐海域生长和繁殖。
(2) 福建牡蛎C. gigas angulata
该种主要分布于西太平洋, 后被引进到葡萄牙和西班牙养殖, 生长快、产量高、适应环境能力强, 是牡蛎养殖的首选种类(Lam et al, 2004)。杜玄等(2009)研究了福建沿海的巨牡蛎属牡蛎种类, 其中福建牡蛎是当地优势种。Wang等(2010)通过基于线粒体基因的系统进化及遗传距离分析将福建牡蛎修订为长牡蛎(C. gigas gigas)的亚种, 该种在福建大量养殖且产量最多, 因此命名为福建牡蛎(王海艳等, 2016)。本次采样发现福建牡蛎在雷州半岛东部沿岸潮间带有大量分布, 且分布区与熊本牡蛎重叠。Lam等(2004)仅在香港两个有持续淡水冲入的海湾发现了福建牡蛎的栖息地。
3.2 我国沿海常见牡蛎属(Ostrea)牡蛎我国南北沿海较常见的牡蛎属牡蛎主要为密鳞牡蛎(O. denselamellosa), 一般栖息于潮下带至水深30m以内的盐度较高且较稳定的浅海, 在朝鲜半岛、日本沿海均有分布(徐凤山等, 2008; 王海艳等, 2016)。密鳞牡蛎个体大, 属幼生型, 适宜繁殖的海水盐度为27—32, 我国一般北部比南部分布多(张玺等, 1956)。本次样品采集未涉及潮下带, 所以没有发现密鳞牡蛎, 潮间带采样发现牡蛎属一未定种(Ostrea sp.1), 形态与密鳞牡蛎差异极大。
3.3 雷州半岛东部沿岸潮间带常见小蛎属牡蛎(1) 棘刺牡蛎S. echinata
张玺等(1956)最早报道我国沿海有棘刺牡蛎的存在, 并描述了它的形态特征。Lam等(2006)对印度-西太平洋的小蛎属牡蛎进行了系统分析, Saccostrea non-mordax lineage B在香港的海下湾(Hoi Ha Wan)和鹤咀(Cape d’Aguilar)两个取样点都有分布, lineage A仅在日本冲绳有分布, 同时根据形态特征提出棘刺牡蛎可能是lineage A或B中的一种。Huber(2010)认为lineage B可能是棘刺牡蛎。本次实验采集到了lineage B分支的小蛎属牡蛎, 根据形态特征和分布我们将其定为棘刺牡蛎。
棘刺牡蛎分布于我国浙江以南沿海, 附着于高低潮线间岩石上, 在日本也有分布。
(2) 团聚牡蛎S. malabonensis
张玺等(1956)和徐凤山等(2008)描述的团聚牡蛎拉丁名为Saccostrea glomerata。Lam等(2006)通过对分布于印度-西太平洋小蛎属牡蛎的系统分析, 认为S. glomerata只在南半球有分部。Anderson等(1994)利用rDNA内转录间隔区的相似性, 认为广泛分布于澳大利亚东部的悉尼岩牡蛎(S. commercialis)是S. glomerata的同物异名, 从此悉尼岩牡蛎拉丁名改为S. glomerata, 并为国际学者所接受。
本次实验采集到了19个lineage F的样品, 有3个单倍型, 常多个个体聚集在一起, 通过形态以及分子生物学分析, 认为它们与S. malabonensis是同一种。Lam等(2006)对印度-西太平洋的小蛎属牡蛎的系统分析中, lineage F在广东的两个取样点没有分布, 但是在海南和台湾有分布。Hamaguchi等(2014)第一次报道了lineage F在日本有分布, 从形态上将Saccostrea malabonensis与小蛎属其他牡蛎区分开。Sekino等(2016)利用线粒体和核基因对日本的小蛎属牡蛎进行了系统分析, 同时通过Saccostrea malabonensis的原始描述:左壳凹陷并有较厚放射肋, 且附着面窄, 认为lineage F可能是Saccostrea malabonensis。Li等(2017)通过形态和线粒体基因对缅甸的牡蛎进行了分析, 认为日本及中国分布的属于lineage F分支的牡蛎和菲律宾的Saccostrea malabonensis是同一种。本研究部分样品的分子序列与lineage F(S. malabonensis)聚为一支, 并且形态与S. malabonensis的原始描述一致, 表明雷州半岛东部沿海也有S. malabonensis的分布, 鉴于该种牡蛎常多个个体聚集在一起, 故仍沿用团聚牡蛎的中文名。
鉴于上述研究及S. glomerata的模式标本产地在斐济岛, 我们认为分布于我国浙江以南沿海潮间带的团聚牡蛎不应再沿用S. glomerata这一种名, 应命名为团聚牡蛎S. malabonensis。团聚牡蛎分布于我国浙江以南沿海潮间带, 同时在日本、新加坡以及西表岛和吕宋岛都有分布(Inaba et al, 2004)。
4 结论雷州半岛东部沿岸潮间带物种丰富, 至少有5种牡蛎分布于潮间带(表 4), 其中熊本牡蛎和福建牡蛎为优势种。熊本牡蛎数量最多, 占样品总量的50.96%, 福建牡蛎的数量次之, 占样品总量的30.03%, 牡蛎属未定种(Ostrea sp.1)只在博贺镇有发现, 且数量较少。两种小蛎属牡蛎数量也较少, 且两种牡蛎分布地点有重合。
| 物种名称 | Crassostrea sikamea | Crassostrea gigas angulata | Ostrea sp.1 | Saccostrea echinata | Saccostrea malabonensis | ||||||
| 地点 | 样品个数 | 185 | 109 | 2 | 48 | 19 | |||||
| 茂名市博贺镇那尾村 | 2 | 2 | |||||||||
| 湛江市南三岛湖村 | 50 | 31 | 18 | 2 | 1 | ||||||
| 雷州市沈塘镇海岚村 | 62 | 58 | 2 | 9 | |||||||
| 湛江市硇洲岛六竹村 | 80 | 35 | 28 | 4 | 16 | ||||||
| 雷州市东里镇沟口东村 | 16 | 16 | 4 | ||||||||
| 湛江市和安镇松树港 | 14 | 8 | 6 | 1 | |||||||
| 湛江市锦和镇东门下村 | 2 | 2 | |||||||||
| 湛江市外罗镇白茅村 | 3 | 1 | 2 | 14 | |||||||
| 湛江市下洋镇下港村 | 57 | 23 | 34 | ||||||||
| 湛江市前山镇后海村 | 14 | 12 | 7 | ||||||||
| 湛江市前山镇盐井角 | 20 | 13 | 5 | 7 | 2 | ||||||
| 王海艳, 2004.中国近海常见牡蛎分子系统演化和分类的研究.青岛: 中国科学院海洋研究所博士学位论文, 39-110 http://www.wanfangdata.com.cn/details/detail.do?_type=degree&id=Y689642 |
| 王海艳, 张涛, 马培振, 等, 2016. 中国北部湾潮间带现生贝类图鉴. 北京: 科学出版社, 104-105 |
| 杜玄, 郭希明, 钱鲁闽, 2009. 福建沿海巨蛎属牡蛎的主要种类及其分布. 台湾海峡, 28(3): 399–404 DOI:10.3969/j.issn.1000-8160.2009.03.015 |
| 宋忠魁, 蔡小辉, 童潼, 等, 2010. 广西茅尾海常见牡蛎的分子鉴定. 海洋科学, 34(8): 11–16 |
| 张玺, 楼子康, 1956. 中国牡蛎的研究. 动物学报, 8(1): 65–93 |
| 张智瀛, 2014. 浅析雷州半岛海域夏季溶解氧的分布特点. 中国新技术新产品,(13): 157–158 |
| 徐凤山, 张素萍, 2008. 中国海产双壳类图志. 北京: 科学出版社, 112-113 |
| 梁超愉, 张汉华, 颉晓勇, 等, 2005. 雷州半岛红树林滩涂底栖生物多样性的初步研究. 海洋科学, 29(2): 18–25, 31 DOI:10.3969/j.issn.1000-3096.2005.02.005 |
| 韩维栋, 高秀梅, 2009. 雷州半岛红树林生态系统及其保护策略. 广州: 华南理工大学出版社, 65-66 |
| Anderson T J, Adlard R D, 1994. Nucleotide sequence of rDNA internal transcribed spacer supports synonymy of Saccostrea commercialis and S. glomerata. Journal of Molluscan Studies, 60: 196–197 DOI:10.1093/mollus/60.2.196 |
| Folmer O, Black M, Hoeh W, et al, 1994. DNA primers for amplification of mitochondrial cytochrome c oxidase subunit I from diverse metazoan invertebrate. Molecular Marine Biology and Biotechnology, 3(5): 294–299 |
| Hamaguchi M, Shimabukuro H, Usuki H, et al, 2014. Occurrences of the Indo-West Pacific rock oyster Saccostrea cucullata in mainland Japan. Marine Biodiversity Records, 7: e84 DOI:10.1017/S1755267214000864 |
| Harry H W, 1985. Synopsis of the supraspecific classification of living oysters (Bivalvia: Gryphaeidae and Ostreidae). Veliger, 28: 121–158 |
| Huber M, 2010. Compendium of Bivalves. Hackenheim: Conch Books, 1-901 |
| Inaba A, Torigoe K, 2004. Oysters in the world, Part 2: systematic description of the recent oyster. Nishinomiya, Hyogo, Japan: Bulletin of Nishinomiya Shell Museum, 28-29 |
| Kimura M, 1980. A simple method for estimating evolutionary rates of base substitutions through comparative studies of nucleotide sequences. Journal of Molecular Evolution, 16(2): 111–220 DOI:10.1007/BF01731581 |
| Kumar S, Stecher G, Tamura K, 2016. MEGA7: Molecular evolutionary genetics analysis version 7.0 for bigger datasets. Molecular Biology and Evolution, 33(7): 1870–1874 DOI:10.1093/molbev/msw054 |
| Lam K, Morton B, 2003. Mitochondrial DNA and morphological identification of a new species of Crassostrea (Bivalvia: Ostreidae) cultured for centuries in the Pearl River Delta, Hong Kong, China. Aquaculture, 228(1-4): 1–13 DOI:10.1016/S0044-8486(03)00215-1 |
| Lam K, Morton B, 2004. The oysters of Hong Kong (Bivalvia: Ostreidae and Gryphaeidae). The Raffles Bulletin of Zoology, 52(1): 11–28 |
| Lam K, Morton B, 2006. Morphological and mitochondrial-DNA analysis of the indo-west pacific rock oysters (Ostreidae: Saccostrea species). Journal of Molluscan Studies, 72(3): 235–245 DOI:10.1093/mollus/eyl002 |
| Li C, Haws M, Wang H Y, et al, 2017. Taxonomic classification of three oyster (Ostreidae) species from Myanmar. Journal of Shellfish Research, 36(2): 365–371 DOI:10.2983/035.036.0209 |
| Librado P, Rozas J, 2009. DnaSP v5: a software for comprehensive analysis of DNA polymorphism data. Bioinformatics, 25(11): 1451–1452 DOI:10.1093/bioinformatics/btp187 |
| Posada D, Crandall K, 1998. MODELTEST: testing the model of DNA substitution. Bioinformatics, 14(9): 817–818 DOI:10.1093/bioinformatics/14.9.817 |
| Raith M, Zacherl D C, Pilgrim E M, et al, 2016. Phylogeny and species diversity of Gulf of California oysters (Ostreidae) inferred from mitochondrial DNA. American Malacological Bulletin, 33(2): 263–283 DOI:10.4003/006.033.0206 |
| Ronquist F, Teslenko M, Van Der Mark P, et al, 2012. Mr Bayes 3.2: efficient Bayesian phylogenetic inference and model choice across a large model space. Systematic Biology, 61(3): 539–542 DOI:10.1093/sysbio/sys029 |
| Sekino M, Yamashita H, 2016. Mitochondrial and nuclear DNA analyses of Saccostrea oysters in Japan highlight the confused taxonomy of the genus. Journal of Molluscan Studies, 82(4): 492–506 DOI:10.1093/mollus/eyw022 |
| Wang H Y, Guo X M, 2008. Identification of Crassostrea ariakensis and related oysters by multiplex species-specific PCR. Journal of Shellfish Research, 27(3): 481–487 DOI:10.2983/0730-8000(2008)27[481:IOCAAR]2.0.CO;2 |
| Wang H Y, Guo X M, Zhang G F, et al, 2004. Classification of Jinjiang oysters Crassostrea rivularis (Gould.1861) from China, based on morphology and phylogenetic analysis. Aquaculture, 242: 137–155 DOI:10.1016/j.aquaculture.2004.09.014 |
| Wang H Y, Qian L M, Liu X, et al, 2010. Classification of a common cupped oyster from southern China. Journal of Shellfish Research, 29(4): 857–866 DOI:10.2983/035.029.0420 |
| Wang H Y, Qian L M, Wang A M, et al, 2013. Occurrence and distribution of Crassostrea sikamea (Amemiya 1928) in China. Journal of Shellfish Research, 32(2): 439–446 DOI:10.2983/035.032.0224 |
| Wu X Y, Xu X D, Yu Z N, et al, 2010. Comparison of seven Crassostrea mitogenomes and phylogenetic analyses. Molecular Phylogenetics and Evolution, 57(1): 448–454 DOI:10.1016/j.ympev.2010.05.029 |
 2018, Vol. 49
2018, Vol. 49


