中国海洋湖沼学会主办。
文章信息
- 王忠良, 丁燏, 许尤厚, 王蓓, 张健东. 2018.
- WANG Zhong-Liang, DING Yu, XU You-Hou, WANG Bei, ZHANG Jian-Dong. 2018.
- 马氏珠母贝(Pinctada fucata)血细胞转录组测序数据中SNP标记的开发及其功能注释分析
- SNP DISCOVERY AND FUNCTIONAL ANNOTATION IN TRANSCRIPTOME DATASETS FROM HEMOCYTES OF PINCTADA FUCATA
- 海洋与湖沼, 49(2): 403-412
- Oceanologia et Limnologia Sinica, 49(2): 403-412.
- http://dx.doi.org/10.11693/hyhz20171000264
-
文章历史
- 收稿日期:2017-10-16
- 收修改稿日期:2017-11-22
2. 南海水产经济动物增养殖广东省普通高校重点实验室 湛江 524088;;
3. 广西北部湾海洋生物多样性养护重点实验室(钦州学院) 钦州 535000
2. Key Laboratory of Aquaculture in South China Sea for Aquatic Economic Animal of Guangdong Higher Education Institutes, Zhanjiang 524088, China;;
3. Guangxi Key Laboratory of Beibu Gulf Marine Biodiversity Conservation, Qinzhou University, Qinzhou 535000, China
马氏珠母贝(Pinctada fucata)是人工培育海水珍珠的主要母贝, 超过90%的海水珍珠产量来自于马氏珠母贝, 在我国海水养殖业中占有重要地位(Zhan et al, 2016)。然而, 由于长期的人工养殖和近亲繁殖, 马氏珠母贝已呈现出种苗活力下降、生长速度减慢、死亡率增加、育珠贝变小、珍珠质量差等明显的种质退化现象, 严重制约了我国海水珍珠产业的健康持续发展(Wang et al, 2009)。因此, 马氏珠母贝的遗传改良和优良品种培育是当前提高珍珠贝养殖效益和健康养殖水平的首要任务。
规模化开发马氏珠母贝分子标记是其种质资源发掘、经济性状关联分析、分子遗传育种技术建立和优良品种培育的基础性工作之一。单核苷酸多态性标记(Single Nucleotide Polymorphism, SNP)是指单个核苷酸发生改变而引起的DNA序列多态性, 最早由美国学者Lander于1996年提出, 是继扩增片段长度多态性(AFLP)、微卫星(SSR)等之后的新一代分子标记(Lander, 1996)。由于具有数量多、密度大、遗传稳定性高、便于高通量自动化检测分析等优点, SNP标记被广泛应用于高密度遗传连锁图谱构建、基因定位、品种鉴定、群体遗传学研究和分子辅助育种等领域(Liu et al, 2004; Mir et al, 2013; 张晓萌等, 2013)。
近年来, 水产动物中SNP标记的开发和应用已有若干报道, 如鱼类的大菱鲆(Scophthalmus maximus) (王婷等, 2014)、斑点叉尾(Ictalurus punctatus) (Li et al, 2014)、大西洋鲑(Salmo salar) (Houston et al, 2014; Tsai et al, 2016), 甲壳类的凡纳滨对虾(Litopenaeus vannamei) (Yu et al, 2014)、斑节对虾(Penaeus monodon) (Sellarset al, 2014)、中华绒螯蟹(Eriocheir sinensis) (Cui et al, 2013)、三疣梭子蟹(Portunus trituberculatus) (张德宁等, 2015), 贝类的栉孔扇贝(Chlamys farreri) (李纪勤等, 2013)、长牡蛎(Crassostrea gigas) (Wang et al, 2017)。马氏珠母贝中也开发了少量经济性状相关SNP标记, 但数量还未达到高密度遗传连锁图谱构建和QTL精细定位的要求(Zhan et al, 2016)。
目前, SNP标记的大规模开发主要依靠基因组测序和序列的拼接比对, 但这种方法费用高、适用范围有限, 限制了SNP标记在缺乏全基因组信息的非模式生物中的开发和利用。随着高通量测序技术的快速发展, 转录组测序成本大幅下降, 比较转录组学方法筛选性状相关基因已成为研究特定机理的普遍方法, 同时通过序列比对可识别大量的SNP位点(Pante et al, 2012; Núñez-Acuña et al, 2013; Cui et al, 2014; Yu et al, 2014; Mastrochirico-Filho et al, 2016; Li et al, 2017)。本文基于前期马氏珠母贝血细胞转录组测序数据进行SNP标记的开发和关联基因的功能注释分析, 以期为马氏珠母贝遗传多样性分析、遗传图谱构建和分子辅助育种研究等提供候选标记资源, 促进马氏珠母贝种质资源保护、优良品种培育和珍珠养殖业的健康发展。
1 材料与方法 1.1 马氏珠母贝及高通量转录组测序数据马氏珠母贝(Pinctada fucata) (平均壳长70mm)购自广东省湛江市雷州后洪珍珠贝养殖场; 暂养于室内玻璃钢水槽中(80L水体), 每槽饲养20只; 水温25±1℃, 盐度28, 饲养期间连续充气; 暂养过程中以螺旋藻粉为饵料投喂, 每天换水(100%)一次。室内暂养一周后将珠母贝分为溶藻弧菌感染组(AI)和对照组(BI); AI组珠母贝闭壳肌注射0.1mL、浓度为5×107 CFU/mL的溶藻弧菌悬液, BI组注射同体积的PBS; 细菌感染4h后闭壳肌采集血淋巴, 800×g、4℃离心10min收集血细胞; TRIzol法提取总RNA, 经浓度和纯度检测后用于Illumina/Hiseq-2000高通量转录组测序(Wang et al, 2016)。
转录组测序共产生70407878条Raw Reads, 经去除含有接头、重复及测序质量较低的原始读数后获得56345139条Clean Reads; 使用de novo组装软件Trinity (Grabherr et al, 2011)对Clean Reads进行组装, 并进行去冗余处理和进一步拼接, 共得到74007条unigenes (Raw Reads数据的SRA登录号为SRP041567) (Wang et al, 2016)。
1.2 SNP位点的检测首先应用SOAP软件将各样本测序所得Clean Reads与Trinity拼接的转录本进行比对(错配≤2);合并单端和双端比对结果, 过滤掉Duplicated Reads和Multi-mapped Reads, 并将比对结果按转录本和坐标位置进行排序; 应用SOAPsnp软件(Li et al, 2009)对排序后的文件进行SNP位点检测, 将得到的SNP结果; 按质量值(≥20)、测序深度(≥10和≤100)和SNP间距(≥5)等条件进行过滤并去杂合, 最终获得纯合SNP位点数据(Duet al, 2014; Lv et al, 2014)。
1.3 SNP位点所在unigene的注释与功能分析应用BLAST程序将含有SNP的unigene (SNP-unigene)序列比对到nr、nt、Swiss-Prot和COG(直系同源簇, Cluster of Orthologous Groups)等数据库, 从而获得该unigene的注释信息(E-value < 1.0E−5);采用Blast2GO软件获得unigene的GO(基因本体论, Gene ontology)注释, 并使用WEGO软件对所有unigene做GO功能分类统计; 通过BLASTx比对SNP-unigene到COG数据库和KEGG(京都基因与基因组百科, Kyoto Encyclopedia of Genes and Genomes)数据库, 分别获得COG分类注释和KEGG信号通路注释。
2 结果与分析 2.1 转录组数据及SNP位点数据分析本研究的数据来源于对照组转录组(BI)和溶藻弧菌感染组转录组(AI)。经SOAPsnp软件检测, 67632条unigenes中共获得962995个SNP位点(BI: 493243个; AI: 469752个)(表 1); BI转录组和AI转录组的SNP发生频率分别为0.0098和0.0093(约1个SNP/100bp); SNP密度频数分析显示, 每1000碱基含0—5个SNP位点的unigene最多(图 1)。所有SNP位点中, 纯合SNP位点105232个(BI: 50702个; AI: 54530个)(表 1); Unigene中所含SNP位点数目统计表明, BI转录组中含有1个SNP位点的unigene 4597条(13.64%), 含有2—10个SNP位点的unigene 14354条(42.60%), 含有10个以上SNP位点的unigene 14746条(43.76%); AI转录组中含有1个SNP位点的unigene 4632条(13.65%), 含有2—10个SNP位点的unigene 15140条(44.61%), 含有10个以上SNP位点的unigene 14163条(41.74%)(图 2)。
| 类别 | BI转录组 | AI转录组 | 总计 |
| SNP-unigene | 33697 | 33935 | 67632 |
| SNP位点总数 | 493243 | 469752 | 962995 |
| 纯合SNP位点 | 50702 | 54530 | 105232 |
| 颠换SNP | 33713 | 36327 | 70040 |
| 转换SNP | 16989 | 18203 | 35192 |
 |
| 图 1 SNP密度频数分布图 Fig. 1 SNP frequency in BI and AI transcriptomes |
 |
| 图 2 SNP的分布统计 Fig. 2 SNP distribution in BI and AI transcriptomes |
SNP类型分析表明, 来源于BI转录组的纯合SNP位点中, 颠换位点33713个(66.49%), 转换位点16989个(33.51%); 来源于AI转录组的纯合SNP位点中, 颠换位点36327个(66.62%), 转换位点18203个(33.38%)。BI转录组和AI转录组转换SNP数目与颠换SNP数目比值分别为0.504和0.501; 6种单核苷酸变异类型中, 以A/T颠换SNP最多, 分别占纯合SNP总数的25.45%(BI: 12906个)和25.72%(AI: 14024个)(图 3)。
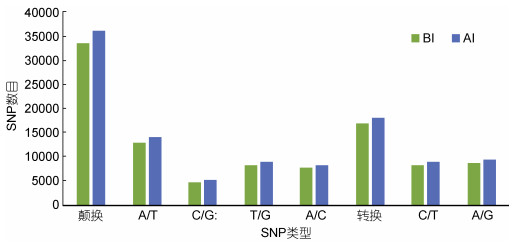 |
| 图 3 SNP类型分析结果 Fig. 3 SNP numbers of different mutation types in BI and AI transcriptomes |
基于所有67632条SNP-unigenes序列与Nr、Nt及Swiss-prot数据库比对后获得的蛋白功能注释信息, 分别进行GO、COG基因功能分类及KEGG信号通路富集分析。GO分析结果表明, 共有30376条SNP-unigenes(44.91%)匹配到GO条目(GO term), 并被分配到生物过程、细胞组分及分子功能三大功能分类中的42个亚类(图 4); COG结果显示, 共有18150条SNP-unigenes(26.84%)能够匹配相应的COG注释信息; 根据功能信息可分为26类, 其中以“一般功能预测”类最多, 包含3690条SNP-unigenes(图 5); KEGG富集分析显示, 共有10981条SNP-unigenes (16.24%)富集到32条KEGG子集中, 其中以“信号转导”子集中富集的SNP-Unigene最多, 共计1450条(图 6)。
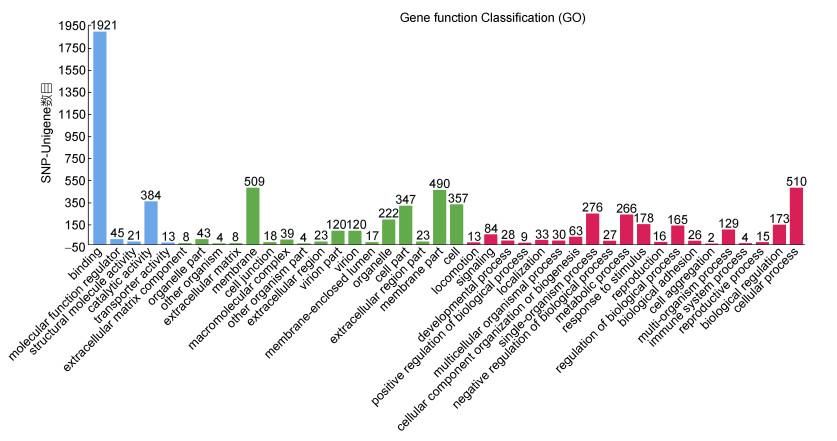 |
| 图 4 SNP-unigenes GO功能注释分析 Fig. 4 Gene ontology (GO) classification of SNP-unigenes |
 |
| 图 5 SNP-unigenes GOG功能注释分析 Fig. 5 The cluster of orthologous groups (COG) classification of SNP-unigenes |
 |
| 图 6 SNP-unigenes的KEGG信号通路富集分析 Fig. 6 The Kyoto encyclopedia of genes and genomes (KEGG) classification of SNP-unigenes |
根据KEGG信号通路的富集分析, 共有629个SNP-unigenes注释到“Platelet activation”等15条与“病原感染”和“免疫功能”相关的一条或多条信号通路中, 其中以“Platelet activation”信号通路中注释的unigene最多(86条), 其次分别为“Chemokine signaling pathway”、“Fc gamma R-mediated phagocytosis”、“Fc gamma R-mediated phagocytosis”和“Leukocyte transendothelial migration”等信号通路(表 2)。
| 免疫防御相关KEGG信号通路 | 信号通路ID | SNP-unigene数目 |
| Platelet activation | ko04611 | 86 |
| Chemokine signaling pathway | ko04062 | 78 |
| Fc gamma R-mediated phagocytosis | ko04666 | 69 |
| Leukocyte transendothelial migration | ko04670 | 63 |
| T cell receptor signaling pathway | ko04660 | 50 |
| Toll-like receptor signaling pathway | ko04620 | 46 |
| Fc epsilon RI signaling pathway | ko04664 | 39 |
| B cell receptor signaling pathway | ko04662 | 35 |
| NOD-like receptor signaling pathway | ko04621 | 35 |
| Natural killer cell mediated cytotoxicity | ko04650 | 34 |
| RIG-I-like receptor signaling pathway | ko04622 | 30 |
| Cytosolic DNA-sensing pathway | ko04623 | 27 |
| Antigen processing and presentation | ko04612 | 19 |
| Complement and coagulation cascades | ko04610 | 9 |
| Hematopoietic cell lineage | ko04640 | 9 |
| 合计 | 629 |
同时, 根据转录组中unigene的SNP位点分布情况及KEGG功能注释信息筛选BI和AI转录组中特异分布的免疫防御相关基因SNP位点, 共获得123个特异分布的SNP位点, 其中BI转录组中36个SNP位点, 存在于12个免疫防御相关基因中; AI转录组中87个SNP位点, 存在于22个免疫防御相关基因中(表 3)。
| 转录组 | SNP-unigene ID | 所属KEGG信号通路 | SNP数目 | 合计 |
| BI | comp39780_c0 | B cell receptor signaling pathway/ Toll-like receptor signaling pathway/ T cell receptor signaling pathway/ Fc epsilon RI signaling pathway/ Fc gamma R-mediated phagocytosis/ NOD-like receptor signaling pathway/ Platelet activation | 7 | 36 |
| comp671474_c0 | Complement and coagulation cascades/Platelet activation | 3 | ||
| comp290115_c0 | Cytosolic DNA-sensing pathway | 5 | ||
| comp732503_c0 | Cytosolic DNA-sensing pathway | 4 | ||
| comp51341_c0 | Fc gamma R-mediated phagocytosis | 1 | ||
| comp57855_c0 | Hematopoietic cell lineage | 1 | ||
| comp44879_c0 | Leukocyte transendothelial migration/ Platelet activation | 3 | ||
| comp59941_c0 | Leukocyte transendothelial migration/Platelet activation / T cell receptor signaling pathway | 3 | ||
| comp1007_c0 | NOD-like receptor signaling pathway | 3 | ||
| comp653385_c0 | NOD-like receptor signaling pathway | 2 | ||
| comp229504_c0 | Platelet activation | 2 | ||
| comp738091_c0 | RIG-I-like receptor signaling pathway | 2 | ||
| AI | comp1306306_c0 | Leukocyte transendothelial migration/Platelet activation | 1 | 87 |
| comp14833_c0 | Hematopoietic cell lineage | 1 | ||
| comp1620486_c0 | Chemokine signaling pathway | 4 | ||
| comp19568_c0 | Leukocyte transendothelial migration/Platelet activation | 1 | ||
| comp22997_c0 | B cell receptor signaling pathway/Chemokine signaling pathway/Fc epsilon RI signaling pathway/Fc gamma R-mediated phagocytosis/Leukocyte transendothelial migration/Natural killer cell mediated cytotoxicity/Platelet activation/T cell receptor signaling pathway/Toll-like receptor signaling pathway | 4 | ||
| comp2349_c0 | Complement and coagulation cascades/Platelet activation | 2 | ||
| comp340170_c0 | Chemokine signaling pathway/Platelet activation | 5 | ||
| comp35583_c0 | Cytosolic DNA-sensing pathway | 3 | ||
| comp364321_c0 | Fc epsilon RI signaling pathway/Fc gamma R-mediated phagocytosis/Platelet activation | 5 | ||
| comp44949_c0 | Fc gamma R-mediated phagocytosis | 9 | ||
| comp47012_c0 | Fc gamma R-mediated phagocytosis | 5 | ||
| comp509876_c0 | Hematopoietic cell lineage | 3 | ||
| comp51181_c0 | Complement and coagulation cascades | 8 | ||
| comp524944_c0 | Chemokine signaling pathway/Platelet activation | 1 | ||
| comp54383_c0 | Leukocyte transendothelial migration/Platelet activation | 1 | ||
| comp55367_c0 | Chemokine signaling pathway/Fc epsilon RI signaling pathway/Fc gamma R-mediated phagocytosis/Leukocyte transendothelial migration/Natural killer cell mediated cytotoxicity/Platelet activation/T cell receptor signaling pathway/Toll-like receptor signaling pathway/Platelet activation | 2 | ||
| comp57390_c0 | Hematopoietic cell lineage/Platelet activation | 7 | ||
| comp579306_c0 | Fc gamma R-mediated phagocytosis | 6 | ||
| comp60708_c0 | Fc epsilon RI signaling pathway/Fc gamma R-mediated phagocytosis/Leukocyte transendothelial migration/T cell receptor signaling pathway | 6 | ||
| comp631969_c0 | Hematopoietic cell lineage/Platelet activation | 1 | ||
| comp63931_c0 | Cytosolic DNA-sensing pathway | 5 | ||
| comp748376_c0 | Platelet activation | 7 | ||
| 合计 | 123 |
基于标准化的基因表达水平分析, 在溶藻弧菌感染前、后的血细胞转录组中共检测到636个差异表达的unigenes (Differentially Expressed Genes, DEGs) (Wang et al, 2016)。根据KEGG信号通路分析结果及unigene的SNP位点检测信息, 获得了部分重要差异表达免疫防御相关基因SNP位点的分布情况。结果显示, 在BI和AI转录组中分别检测到700和894个SNP位点(共1594个SNP位点), 并分布于17个差异表达免疫防御相关基因中(表 4)。
| 基因名称 | 免疫后表达水平 | BI-SNP数目 | AI-SNP数目 |
| Alpha-L-iduronidase | 上调 | 14 | 49 |
| Apoptosis regulator R1 | 上调 | 51 | 95 |
| Arrestin | 上调 | 79 | 45 |
| cAMP-responsive element modulator | 上调 | 64 | 69 |
| Cytosolic phospholipase A2 | 上调 | 41 | 37 |
| Dual specificity protein phosphatase 10 | 上调 | 29 | 43 |
| Dual specificity protein phosphatase 7 | 上调 | 49 | 46 |
| Heat shock protein 90 | 上调 | 56 | 47 |
| Interleukin-1 receptor | 上调 | 53 | 60 |
| MAP kinase-activated protein kinase 2 | 上调 | 75 | 77 |
| myeloid differentiation primary response protein 88 | 上调 | 0 | 41 |
| Nuclear transcription factor Y subunit alpha | 上调 | 34 | 62 |
| Putative inhibitor of apoptosis | 上调 | 6 | 15 |
| Serine/threonine protein kinase NLK | 上调 | 20 | 68 |
| Toll-like receptor 2 | 下调 | 39 | 37 |
| Toll-like receptor 3 | 上调 | 38 | 45 |
| Tumor necrosis factor | 上调 | 52 | 58 |
| 合计 | 700 | 894 |
在基因组中, SNP标记相比于SSR等分子标记具有更高的稳定性, 且符合孟德尔遗传规律, 方便用于遗传研究分析中; 同时, SNP标记的准确性和重现性也优于其它分子标记。因此, 作为第三代分子标记的SNP具有广阔的应用前景。然而, SNP开发、检测的技术难点限制了SNP标记在非模式生物中的开发和应用。目前, SNP标记的大规模开发主要是利用基因组测序及序列的拼接比对产生, 但这种途径成本较高(庄伟, 2010); 而对于没有基因组参考序列的非模式生物, 多利用公共数据库的EST序列进行筛选, 但EST序列与实际序列间可能存在差异, 所以这种方法的工作量大、开发效率低, 且覆盖不广(刘庆明, 2012)。随着高通量测序技术的发展, 转录组测序很好地弥补了以上方法的不足和缺陷, 其不仅成为研究生物免疫防御、生长发育、环境互作等复杂分子机制的重要手段, 也是多态性SNP分子标记鉴定的重要数据来源(周华等, 2012)。
本研究基于Hiseq-2000测序技术从67632条unigenes中共获得962995个候选SNP位点, SNP发生频率为0.0098和0.0093(平均约100bp含有1个SNP), 研究结果与女王扇贝(Aequipecten opercularis)和黑扇贝(Mimachlamys varia)SNP分布频率相近(1个SNP/100bp) (Arias et al, 2009), 稍低于长牡蛎的SNP分布频率(1个SNP/40—60bp) (Sauvage et al, 2007), 但明显高于栉孔扇贝的SNP分布频率(1个SNP/ 426.5bp) (李纪勤等, 2013)。各物种SNP分布频率的差异可能与其生长环境的复杂程度及遗传背景的差异有关(张德宁等, 2015), 另一个可能的原因是在EST序列中SNP的筛选条件不同(李纪勤等, 2013)。SNP碱基替换类型分为转换(由嘌呤置换嘌呤或嘧啶置换嘧啶)和颠换(由嘌呤置换嘧啶或由嘧啶置换嘌呤)两类, 理论上发生转换的概率与发生颠换的概率比值应该为0.5。本研究中, BI转录组和AI转录组转换SNP数目与颠换SNP数目比值分别为0.504和0.501, 与理论比率相符。然而, 长牡蛎、三疣梭子蟹、中国对虾(Fenneropenaeus chinensis)等无脊椎动物SNP标记中转换的比例远大于颠换, 转换/颠换比值大于0.5(王家丰, 2013; 张德宁等, 2015; 逄锦菲, 2013)。这种差异被称为“转换偏差”(Gojobori et al, 1982; Liet al, 1984; Collins et al, 1994)。这种不同物种间转换/颠换比值的较大差异可能与不同生物在进化中承受的压力不同有一定关系(Zhao et al, 2002)。
利用转录组测序数据开发SNP标记具有一个极为显著的优点, 即标记位点来自与功能基因直接相关的编码序列, 便于开展进一步的基因功能及遗传性状解析研究(王婷等, 2014)。本研究获得的67632条SNP-unigenes中, 分别有30376条、18150条和10981条SNP-unigenes在GO、COG及KEGG数据库中获得功能注释, 大部分unigene参与细胞代谢、生物合成、生物(非生物)胁迫(刺激)反应、信号转导、机体组成及激素分泌等过程, 表明这些SNP-unigene广泛参与马氏珠母贝的各种生命活动, 并影响其各种遗传性状的建立; 同时, 这些SNP标记的开发为将来的基因功能、基因表达调控模式等研究提供了跟踪标志。此外, 来自于编码区的SNP因其对氨基酸编码的影响可能导致疾病发生或对环境毒物敏感度的改变, 因此SNP也被用于疾病遗传学和抗病性状关联性研究中。如刘敬文(2014)开展了凡纳滨对虾免疫相关基因SNP标记与WSSV抗性的关联分析, 最终确定了681个SNP组成的免疫基因候选标记库以指导抗性品种的选育; 逄锦菲(2013)利用开发的SNP候选位点, 以中国对虾的抗病群体和敏感群体为研究材料进行SNP基因型与抗病性状的相关性分析, 并鉴别出抗病相关SNP位点。为了进一步研究马氏珠母贝免疫防御机制, 筛选抗病相关SNP位点, 本文根据转录组unigene的注释信息, 共获得了629个含有SNP位点的免疫防御相关unigenes, 并被注释到“Platelet activation”等多条重要的免疫防御信号通路中; 同时, 根据KEGG功能注释信息及SNP位点分布情况筛选到123个细菌感染前、后转录组中特异分布的免疫防御相关基因SNP位点。以上免疫防御相关SNP-unigene及特异分布SNP位点的获得为以分子标记应用为基础的马氏珠母贝抗病家系选育提供了有力辅助。
4 结论本研究首次应用高通量转录组测序数据实现了马氏珠母贝SNP标记的高效率大规模开发, 共获得962995个SNP位点, 位点分布频率约为1个SNP/100bp; 共有629个SNP-unigenes被注释到多种免疫防御相关信号通路中; 根据SNP位点分布情况, 筛选到123个特异分布的免疫防御相关候选SNP位点。以上研究结果对于丰富马氏珠母贝分子标记类型、阐明免疫防御分子机制、探究种群遗传多样性和保护种质资源均具有重要意义。
| 王婷, 黄智慧, 马爱军, 等, 2014. 基于转录组数据的大菱鲆(Scophthalmus maximus)SNP标记开发及多态性分析. 海洋与湖沼, 45(6): 1300–1307 |
| 王家丰, 2013. 长牡蛎基因区SNP标记规模开发及其在遗传育种研究中的应用. 青岛:中国科学院研究生院(海洋研究所)博士学位论文 |
| 刘庆明, 2012. 应用高分辨率熔解曲线开发大菱鲆SNP标记研究. 上海:上海海洋大学硕士学位论文 |
| 刘敬文, 2014. 凡纳滨对虾免疫基因SNPs开发及其与WSSV抗性的关联分析. 青岛:中国科学院研究生院(海洋研究所)硕士学位论文 |
| 庄伟, 2010. 半滑舌鳎(Cynoglossus semilaevis)基因组SNP标记的开发与检测. 青岛:中国海洋大学硕士学位论文 |
| 张晓萌, 马普, 王洪迪, 等, 2013. SNPs在水产动物中的研究进展. 生物技术通报, 8: 7–11 |
| 张德宁, 吕建建, 刘萍, 等, 2015. 三疣梭子蟹生长相关SNP位点的鉴定. 中国水产科学, 22(3): 393–401 |
| 李纪勤, 包振民, 李玲, 等, 2013. 栉孔扇贝EST-SNP标记开发及多态性分析. 中国海洋大学学报:自然科学版,(1): 56–63 |
| 周华, 张新, 刘腾云, 等, 2012. 高通量转录组测序的数据分析与基因发掘. 江西科学, 30(5): 607–611 |
| 逄锦菲, 2013. "黄海2号"中国对虾高通量SNP筛选及其与抗WSSV性状的关联分析. 上海:上海海洋大学硕士学位论文 |
| Arias A, Freire R, Boudry P, et al, 2009. Single nucleotide polymorphism for population studies in the scallops Aequipecten opercularis and Mimachlamys varia. Conservation Genetics, 10(5): 1491–1495 DOI:10.1007/s10592-008-9766-z |
| Collins D W, Jukes T H., 1994. Rates of transition and transversion in coding sequences since the human-rodent divergence. Genomics, 20(3): 386–396 DOI:10.1006/geno.1994.1192 |
| Cui J, Wang H D, Liu S K, et al, 2014. Transcriptome analysis of the gill of Takifugu rubripes using Illumina sequencing for discovery of SNPs. Comparative Biochemistry and Physiology Part D:Genomics and Proteomics, 10: 44–51 DOI:10.1016/j.cbd.2014.03.001 |
| Cui Z, Li X, Liu Y, et al, 2013. Transcriptome profiling analysis on whole bodies of microbial challenged Eriocheir sinensis larvae for immune gene identification and SNP development. PLoS One, 8(12): e82156 DOI:10.1371/journal.pone.0082156 |
| Du X, Li L, Zhang S, et al, 2014. SNP identification by transcriptome sequencing and candidate gene-based association analysis for heat tolerance in the bay scallop Argopecten irradians. PLoS One, 9(8): e104960 DOI:10.1371/journal.pone.0104960 |
| Gojobori T, Li W H, Graur D., 1982. Patterns of nucleotide substitution in pseudogenes and functional genes. Journal of Molecular Evolution, 18(5): 360–369 DOI:10.1007/BF01733904 |
| Grabherr M G, Haas B J, Yassour M, et al, 2011. Trinity:reconstructing a full-length transcriptome without a genome from RNA-Seq data. Nature Biotechnology, 29(7): 644–652 DOI:10.1038/nbt.1883 |
| Houston R D, Taggart J B, Cézard T, et al, 2014. Development and validation of a high density SNP genotyping array for Atlantic salmon (Salmo salar). BMC Genomics, 15(1): 90 DOI:10.1186/1471-2164-15-90 |
| Lander E S., 1996. The new genomics:global views of biology. Science, 274(5287): 536–539 DOI:10.1126/science.274.5287.536 |
| Li R Q, Li Y R, Fang X D, et al, 2009. SNP detection for massively parallel whole-genome resequencing. Genome Research, 19(6): 1124–1132 DOI:10.1101/gr.088013.108 |
| Li S J, Liu H, Bai J J, et al, 2017. Transcriptome assembly and identification of genes and SNPs associated with growth traits in largemouth bass (Micropterus salmoides). Genetica, 145(2): 175–187 DOI:10.1007/s10709-017-9956-z |
| Li W H, Wu C I, Luo C C., 1984. Nonrandomness of point mutation as reflected in nucleotide substitutions in pseudogenes and its evolutionary implications. Journal of Molecular Evolution, 21(1): 58–71 DOI:10.1007/BF02100628 |
| Li Y, Liu S K, Qin Z K, et al, 2014. Construction of a high-density, high-resolution genetic map and its integration with BAC-based physical map in channel catfish. DNA Research, 22(1): 39–52 |
| Liu Z J, Cordes J F., 2004. DNA marker technologies and their applications in aquaculture genetics. Aquaculture, 238(1-4): 1–37 |
| Lv J J, Liu P, Gao B Q, et al, 2014. Transcriptome analysis of the Portunus trituberculatus:de novo assembly, growth-related gene identification and marker discovery. PLoS One, 9(4): e94055 DOI:10.1371/journal.pone.0094055 |
| Mastrochirico-Filho V A, Hata M E, Sato L S, et al, 2016. SNP discovery from liver transcriptome in the fish Piaractus mesopotamicus. Conservation Genetics Resources, 8(2): 109–114 DOI:10.1007/s12686-016-0521-3 |
| Mir R R, Hiremath P J, Riera-Lizarazu O, et al, 2013. Evolving molecular marker technologies in plants:from RFLPs to GBS. In:Lübberstedt T, Varshney R eds. Diagnostics in Plant Breeding. Dordrecht:Springer,: 229–247 |
| núñez-Acuña G, Gallardo-Escárate C., 2013. Identification of immune-related SNPs in the transcriptome of Mytilus chilensis through high-throughput sequencing. Fish & Shellfish Immunology, 35(6): 1899–1905 |
| Pante E, Rohfritsch A, Becquet V, et al, 2012. SNP detection from de novo transcriptome sequencing in the bivalve Macoma balthica:marker development for evolutionary studies. PLoS One, 7(12): e52302 DOI:10.1371/journal.pone.0052302 |
| Sauvage C, Bierne N, Lapègue S, et al, 2007. Single nucleotide polymorphisms and their relationship to codon usage bias in the Pacific oyster Crassostrea gigas. Gene, 406(1-2): 13–22 DOI:10.1016/j.gene.2007.05.011 |
| Sellars M J, Dierens L, McWilliam S, et al, 2014. Comparison of microsatellite and SNP DNA markers for pedigree assignment in Black Tiger shrimp, Penaeus monodon. Aquaculture Research, 45(3): 417–426 DOI:10.1111/are.2014.45.issue-3 |
| Tsai H Y, Robledo D, Lowe N R, et al, 2016. Construction and annotation of a high density SNP linkage map of the Atlantic salmon (Salmo salar) genome. G3:Genes, Genomes, Genetics, 6(7): 2173–2179 |
| Wang J L, Li Q., 2017. Characterization of novel EST-SNP markers and their association analysis with growth-related traits in the Pacific oyster Crassostrea gigas. Aquaculture International, 25(5): 1707–1719 DOI:10.1007/s10499-017-0142-1 |
| Wang Z L, Wu Z H, Jian J C, et al, 2009. Cloning and expression of heat shock protein 70 gene in the haemocytes of pearl oyster (Pinctada fucata, Gould 1850) responding to bacterial challenge. Fish & Shellfish Immunology, 26(4): 639–645 |
| Wang Z L, Wang B, Chen G, et al, 2016. Transcriptome analysis of the pearl oyster (Pinctada fucata) hemocytes in response to Vibrio alginolyticus infection. Gene, 575(2): 421–428 DOI:10.1016/j.gene.2015.09.014 |
| Yu Y, Wei J K, Zhang X J, et al, 2014. SNP discovery in the transcriptome of white Pacific shrimp Litopenaeus vannamei by next generation sequencing. PLoS One, 9(1): e87218 DOI:10.1371/journal.pone.0087218 |
| Zhan X, Wen H Y, Shi Y H, et al, 2016. Association between novel EST-SNPs and commercial traits in Pinctada fucata martensii. Aquaculture Reports, 3: 209–213 DOI:10.1016/j.aqrep.2016.03.006 |
| Zhao Z, Boerwinkle E., 2002. Neighboring-nucleotide effects on single nucleotide polymorphisms:a study of 2. 6 million polymorphisms across the human genome. Genome Research, 12(11): 1679–1686 |
 2018, Vol. 49
2018, Vol. 49


