中国海洋湖沼学会主办。
文章信息
- 胡利腾, 夏立萍, 武敏敏, 陈晋, 张建设. 2018.
- HU Li-Teng, XIA Li-Ping, WU Min-Min, CHEN Jin, ZHANG Jian-She. 2018.
- 太平洋真宽水蚤(Eurytemora pacifica) Cu/Zn SOD基因克隆及在重金属胁迫下的表达分析
- CLONING AND EXPRESSION ANALYSIS OF Cu/Zn SOD GENE OF EURYTEMORA PACIFICA UNDER METAL STRESS
- 海洋与湖沼, 49(2): 384-394
- Oceanologia et Limnologia Sinica, 49(2): 384-394.
- http://dx.doi.org/10.11693/hyhz20171000261
-
文章历史
- 收稿日期:2017-10-15
- 收修改稿日期:2017-11-15
超氧化物歧化酶(SOD)是一种广泛存在于微生物、动植物细胞的金属酶, 根据其本身所相关的金属离子的不同, SOD可以分为三种类型: Cu/Zn SOD, Mn SOD以及Fe SOD (Fridovich, 1986)。不同类型的SOD在不同组织或同一个组织不同阶段中的表达均有所不同。其中, Cu/Zn SOD作为最早被发现的、存在最为广泛的超氧化物歧化酶, 具有重要的生理功能和药用前景(Fattman et al, 2003)。一般而言, 生物体内存在两种Cu/Zn SOD一种是胞外Cu/Zn SOD (extracellular Cu/Zn SOD), 在N末端具有信号肽结构, 另一种是胞质Cu/Zn SOD (cytoplasmic Cu/Zn SOD)不带有信号肽结构。Cu/Zn SOD主要存在于真核细胞的细胞浆内, 呈蓝绿色(Shen et al, 1989), 分子量在32kDa上下, 是有两个亚基构成, 所有亚基分子量约为16kDa, 所有亚基具有1个Cu和1个Zn (Putnam et al, 2000)。来自不同的Cu/Zn SOD氨基酸序列, 一种来自细菌(Chary et al, 1990)真菌高等植物叶绿体或细胞质(LIN et al, 2002), 另一种来自人和高等动物的细胞质(Amano et al, 1990), 同源性都比较接近(Arhontaki et al, 2002; Bodył et al, 2007)。在Cu/Zn SOD的氨基酸生成中, 色氨酸与酩氨酸的含量较低, 或者没有。但甘氨酸含量占得比重较高, 其中6到8个氨基酸残基中就含有一个甘氨酸残基。在高级结构中, Cu/Zn SOD含有8股逆向平行β-折叠, 还含有大约5%的α-螺旋与环状的无规则结构。Cu/Zn SOD中仅有的一对键内二硫键, 是有55位和144位的半胱氨酸构成的二硫键, 对Cu/Zn SOD结构的稳定有着重要的影响。对比活性中心的作用Cu和Zn离子具有明显差异, Cu是必要的, 酶一旦失去Cu就会导致失活, 然而Zn在被Co、Hg、Cd取代后对活性不造成影响。
最近几年, 工业、社会经济的快速崛起, 产生大量生活、工业污水及废物等流入海洋、湖泊、池塘等, 造成了水域生态系统严重破坏。随着海洋环境污染监测要求的提高, 有研究表明, 当污染影响到水生动物时, 其体内会产生氧化应激, 且诱导ROS水平明显加大, 加快水生生物体内基因表达或抗氧化酶的酶活力的变化(Nahrgang et al, 2010)。Varessa Nairsumie等人对膝沟藻超氧化物歧化酶在重金属汞、镉、锡、铜胁迫下的表达研究发现, 慢性毒性效应下细胞中SOD活性在第一天即开始增加, 增量分别是对照组活性的1.34、1.48、1.27及1.39倍; 急性毒性下, SOD值增加幅度大致与慢性毒性胁迫相同, 但上升速度更快、时间更短。推断SOD基因在藻类金属毒性胁迫免疫中发挥重要的调节作用(Gressler et al, 2010)。Hansen等(2006)研究了河鳟SOD在不同金属浓度下的转录水平表达量, 结果表明各慢性金属胁迫下其表达均有所增强, 其中铜暴露下鳟鱼的氧化应激易感性有所提高, 尤其是肝脏出的表达量较高; 此外, 河鳟对金属胁迫的适应性来自于驯化适应而非遗传(Hansen et al, 2006)。赵元凤等(2003)研究了Cd2+、0#柴油、二甲苯对毛蚶肌肉超氧化物歧化酶(SOD)活性的影响, 结果表明3种污染物对毛蚶SOD表现出不同程度诱导作用, 其中对Cd2+污染反应十分敏感, 且毒性强弱次序为Cd2+、0#柴油、二甲苯(赵元凤等, 2003)。因此有研究表明, SODs可成为鱼类对水环境污染的敏感指标(郑清梅等, 2014), 适用于环境污染的生化分析(Whitfield et al, 2002)。
太平洋真宽水蚤(Eurytemora pacifica)作为海洋浮游生物的重要组成部分, 作为太平洋东部海域东海海域(特别是舟山海域)优势浮游动物之一, 由于其分布的广泛性与海洋食物链中的特殊地位使得其在海洋海域生态环境状况的指示中具有重要作用, 在海洋生态环境监测与防控中具有举足轻重的地位。然而, 目前对太平洋真宽水蚤Cu/Zn SOD基因的克隆与重金属胁迫下的表达研究尚未见报道。本研究以太平洋真宽水蚤为实验材料, 通过RACE方法获得Cu/Zn SOD基因序列, 利用分子生物学手段对Ep.Cu/Zn SOD基因的生物学特性及重金属胁迫下的表达特征进行研究。
1 材料与方法 1.1 实验材料实验所用太平洋真宽水蚤(Eurytemora pacifica)采集自浙江省舟山市长峙岛附近海域, 体长为1.20—1.40mm, 经解剖镜下筛选纯化, 实验室暂养7d后取样实验, 暂养条件:水温(20±0.5)℃, 海水盐度28, 光暗周期12h︰12h, 24h充氧, 定期投喂饵料海水小球藻。实验时, 选取子代中外观形态与附肢完整, 生命活性强且健康的太平洋真宽水蚤的成体为实验对象; 实验样品处理后, 由液氮速冻后, 置于–80℃保存。克隆材料选取是从每组对照组中各随机选取10只太平洋真宽水蚤, qRT-PCR的实验材料为不同金属胁迫处理后的太平洋真宽水蚤的总RNA, 每个实验组下个体重复3次。
1.2 RNA提取和cDNA合成在对照组中挑取10mg/μL组织个体, 放入1.5mL的灭菌离心管管内作为总RNA提取, 实验方法是通过TaKaRa公司的Trizol Total RNA提取试剂盒方法进行操作, 得到的总RNA以1.5%非变性琼脂糖电池检验, 并放置在紫外分光光度计(Bio-Rad, USA)下检测, 以TaKaRa M-MLV RTase cDNA Synthesis Kit试剂盒对所得到的RNA反转录, 获取cDNA。
1.3 RACE得到目的基因全长序列通过已知的桡足类Cu/Zn SOD全长序列设计并引物获得太平洋真宽水蚤Cu/Zn-SOD基因的中间序列片段、采用RACE技术, 设计Cu/Zn SOD基因特异性引物, 3’RACE与5’cDNA按照TaKaRa的RACE试剂盒说明书步骤进行实验, 并将得到的目的片段通过分离纯化后链接到PMD18-T载体上, 再转接到Dh5α感受态细泡中, 选择阳性克隆送上海美吉生物技术有限公司测序。实验所用引物见表 1。
| 基因 | 引物 | 引物序列(5′—3′) | 扩增片段(bp) | 作用 |
| Cu/Zn SOD | Cu/Zn SOD F | CGAACGGGTGTATGTCTGCT | 93 | 核心序列 |
| Cu/Zn SOD R | CGTCACTGATGGCGACCTTA | |||
| Cu/Zn SOD 5′outer | TCACTGATGGCGACCTTAGCAAC | 345 | 5′RACE | |
| Cu/Zn SOD 5′inner | CGGCAGTAACATTCCCTAAATCG | 287 | ||
| Cu/Zn SOD 3′outer | TAACACGAACGGGTGTATGTCTG | 254 | 3′RACE | |
| Cu/Zn SOD 3′inner | GGCGATTTAGGGAATGTTACTGC | 192 |
用Dnaman8.0软件将得到的测序结果拼接, 获得太平洋真宽水蚤Cu/Zn SOD基因的cDNA全长序列。非编码区与编码区的检测采用NCBI在线网址ORF finder (http://www.ncbi.nlm.nih.gov/gorf/orfig.cgi)来预测。分子量和等电点在Expasy网站(http://web.expasy.org/computepi)进行分析比较, 使用SignalP 4.1 Server网址为与TMHMM Serverv.2.0分别对其进行信号肽以及跨膜区的预测; 使用Swissport在线软件对Cu/Zn SOD基因的蛋白质结构进行预测分析http://swissmodel.expasy.org/; 用SAS在线序列处理工具包对Cu/Zn SOD基因氨基酸序列与其他物种进行同源性比对, 使用MEGA6.0以Maximum Likelihood method构建系统进化树。
1.5 实时荧光定量PCR(qRT-PCR)表达分析利用Prime primer 5.0软件在Cu/Zn SOD基因编码区范围内设计荧光定量PCR所需的特异性引物, 内参引物为18S rRNA。实验所用引物由上海华大生物公司合成, 采用PAGE纯化方式。样品经过重金属铬和镍及其联合半致死浓度LC50, 以及1/2LC50、1/4LC50以及1/8LC50暴露处理, 进行总RNA提取, 然后反转录得到cDNA模板作为实验荧光定量PCRD的样品。设置3个实验重复和3个技术重复的内参和目的基因。实验参照数据以未重金属胁迫组太平洋真宽水蚤为对照, 删除每3个重复中差异较大的数据, 剩下的数据采用2–△△CT法进行Cu/Zn SOD基因表达分析, 分析数据使用Spss 22.0进行显著性差异分析和单因素方差分析, 当P < 0.05时为显著差异。
| 引物 | 引物序列(5′—3′) | 扩增片段(bp) | 作用 |
| Cu/Zn SOD F | CGAACGGGTGTATGTCTGCT | 91 | qRT-PCR primer |
| Cu/Zn SOD R | CGTCACTGATGGCGACCTTA | 209 | qRT-PCR control primer |
| 18S F | TGCATGGCCGTTCTTAGTTG | ||
| 18S R | TCAATTCAGTGTAGCGCGTG |
以太平洋真宽水蚤RNA反转录后cDNA为模板, 以Cu/Zn SOD.F/R为引物扩增获得目的片段, 根据该终极那序列RACE引物, 采用RACE方法获得Cu/Zn SOD基因351bp的5′端序列以及510bp的3′端序列(图 1)。
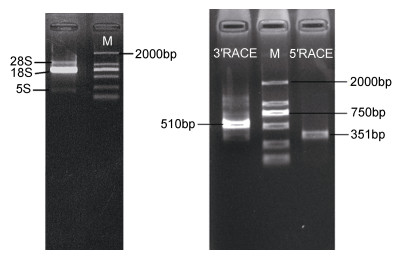 |
| 图 1 RNA, Cu/Zn-SOD 5′/3′RACE电泳图片 Fig. 1 Electrophoresis pictures of RNA, and 5′/3′RACE of Cu/Zn-SOD |
利用DNAMAN 8.0软件将扩增获得到的核心片段以及351bp的5′RACE和510bp的3′RACE序列片段进行拼接, 得到太平洋真宽水蚤Cu/Zn SOD基因的cDNA序列全长(MF289343)为837bp, ORF长456bp, 编码152个氨基酸(aa), 5′UTR与3′UTR分别由297bp, 84bp的核苷酸组成, 具有典型的polyA尾巴以及AATAAA加尾信号(图 2)。
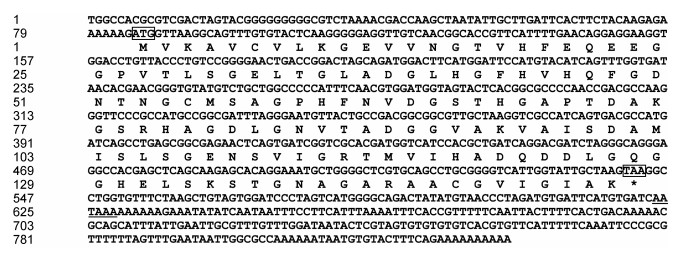 |
| 图 2 EP. Cu/Zn SOD cDNA序列及编码氨基酸序列 Fig. 2 Nucleotide and deduced amino acid sequences of Ep. Cu/Zn-SOD 注:预测的开放阅读框中起始密码子(ATG)和终止密码子(TAA)用黑色线框标出, “*”表示终止密码子 |
使用在线分析软件SMS多重序列同源比对, 将Ep. Cu/Zn SOD基因编码的氨基酸序列与目前已经公布的部分甲壳类的Cu/Zn SOD基因进行比对(图 3), 并通过DNAMAN8.0软件对所有序列与太平洋真宽水蚤Cu/Zn SOD蛋白序列之间的同源相似性进行计算(表 3)。结果显示, 太平洋真宽水蚤(Eurytemora pacifica)与太平洋纺锤水蚤(Acartia pacifica)相似度最高为84%, 与其他甲壳类如海湾扇贝(Argopecten irradians)、近江牡蛎(Crassostrea hongkongensis)和印地安死水牡蛎(Crassostrea madrasensis)等相似度均为71%。表明Ep. Cu/Zn SOD基因为属于Cu/Zn SOD家族基因成员。
 |
| 图 3 Ep. Cu/Zn SOD基因氨基酸序列多重比对 Fig. 3 Multiple sequence alignment of deduced amino acid sequence of Ep. Cu/Zn SOD gene |
| 序列名称 | 登录号 | 来源物种 | 同源性/% |
| 太平洋真宽水蚤Cu-Zn SOD | MF289343 | Eurytemora pacifica | |
| 太平洋纺锤水蚤Cu-Zn SOD | ALS04106.1 | Acartia pacifica | 84 |
| 海湾扇贝Cu-Zn SOD | ACE76954.1 | Argopecten irradians | 71 |
| 近江牡蛎Cu-Zn SOD | ADR70998.1 | Crassostrea hongkongensis | 71 |
| 印地安死水牡蛎Cu-Zn SOD | AFV52312.1 | Crassostrea madrasensis | 71 |
| 曼氏无针乌贼Cu-Zn SOD | AHJ09887.1 | Sepiella maindroni | 64 |
| 美国鲎Cu-Zn SOD | XP013783884.1 | Limulus polyphemus | 69 |
| 背角无齿蚌Cu-Zn SOD | AMO00630.1 | Sinanodonta woodiana | 70 |
| 合浦珠母贝Cu-Zn SOD | AFM75822.1 | Pinctada fucata | 70 |
| 食用牡蛎Cu-Zn SOD | ADF80414.1 | Ostrea edulis | 70 |
| 太平洋牡蛎Cu-Zn SOD | XP011426932.1 | Crassostrea gigas | 69 |
| 褶纹冠蚌Cu-Zn SOD | ACI28282.1 | Cristaria plicata | 68 |
使用MEGA 6.0软件构建系统进化树, 计算方法采用Maximum likelihood method, 重复计算10000次。系统进化树中所用物种名及GenBank的登录号等相关信息在表 4列出。结果如图 4所示, 太平洋真宽水蚤Cu/Zn SOD基因与太平洋纺锤水蚤(Acartia pacifica)位于同一进化枝, 氨基酸序列相似度最高, 表明亲缘关系最近; 同曼氏无针乌贼(Sepiella maindroni)和褶纹冠蚌(Cristaria plicata)的氨基酸序列相似度较低, 表明亲缘关系相对较远; 总体上, 桡足类(Copepods)与贝类(Shellfish)系统进化发育树聚为一簇, 与鱼类的进化关系较远。
| 位点 | 名称 | 序列 |
| PS00001 | N-glycosylation site | N-{P}-[ST]-{P} |
| PS00005 | Protein kinase C phosphorylation site | [ST]-x-[RK] |
| PS00006 | Casein kinase Ⅱ phosphorylation site | [ST]-x(2)-[DE] |
| PS00008 | N-myristoylation site | G-{EDRKHPFYW}-x(2)-[STAGCN]-{P} |
| PS00342 | Microbodies C-terminal targeting signal | [STAGCN]-[RKH]-[LIVMAFY] > |
| PS00087 | Copper/Zinc superoxide dismutase signature | [GA]-[IMFAT]-H-[LIVF]-H-x(2)-[GP]-[SDG]-x-[STAGDE] |
| PS00332 | Copper/Zinc superoxide dismutase signature | G-[GN]-[SGA]-G-x-R-x-[SGA]-C-x(2)-[Ⅳ] |
| PS00443 | Glutamine aminotransferases class-Ⅱ act | < x(0, 11)-C-[GS]-[Ⅳ]-[LIVMFYW]-[AG] |
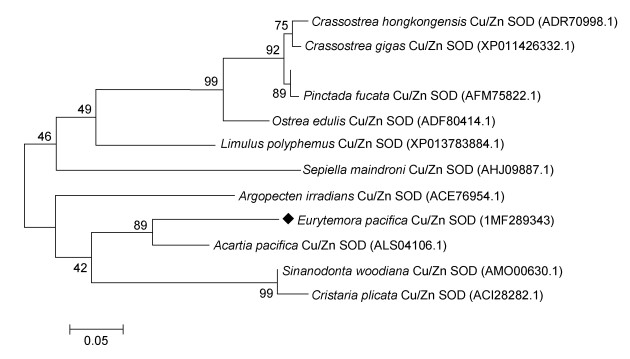 |
| 图 4 Cu/Zn SOD的系统进化树 Fig. 4 Molecular phylogenetic analysis of Cu/Zn SOD by maximum likelihood method |
分析使用DNAMAN软件对Cu/Zn SOD蛋白进行氨基酸疏水性分析, 结果如图 5, 氨基酸的疏水性最大为2.48, 最小值为–2.56, 均值为–0.087368421, 所有多肽链中大多数氨基酸的分值较低, 疏水性氨基酸70个, 亲水氨基酸为82个, 亲水性氨基酸显著多于疏水性氨基酸, 推测该蛋白为亲水性蛋白。
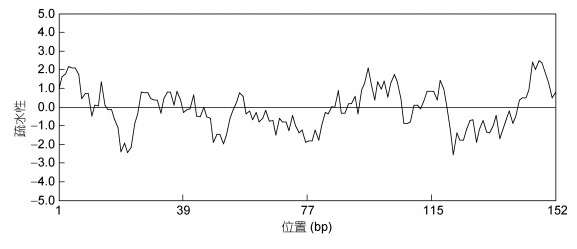 |
| 图 5 Cu/Zn SOD氨基酸疏水性分析 Fig. 5 Analysis of Cu/Zn SOD hydrophobic amino acid |
使用TMHMM 2.0在线软件对蛋白进行跨膜区分析, 结果如图 6所示; 该蛋白的氨基酸没有明显的跨膜区。使用Swiss Pdb Viewer4.0.1软件进行Cu/Zn SOD蛋白进行功能位点模式(Prosite pattern)分析, 发现具有8个蛋白翻译后修饰位点: PS00001 N-糖基化位点; PS00005磷酸化位点; PS00006酪蛋白激酶II磷酸化位点; PS00008豆蔻酰化位点; PS00342微体C-末端的定位信号; PS00087/PS00332铜锌超氧化物歧化酶签名; PS00443谷氨酸转氨酶的II类段位(表 4)。
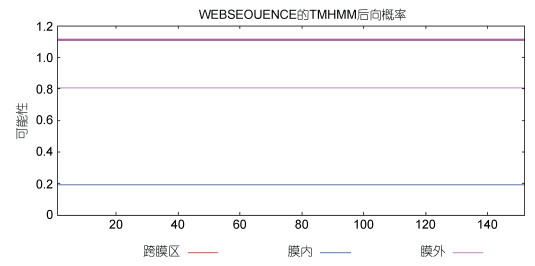 |
| 图 6 Cu/Zn SOD氨基酸序列跨膜区分析 Fig. 6 Transmembrane region analysis of Cu/Zn SOD amino acid sequence |
使用Expasy网站中Peptide Cutter软件(http://web.expasy.org/peptide_cutter/)对蛋白的酶切位点进行分析, 分析结果如下表 5所示。
| 酶 | 酶切位点 |
| Arg-C proteinase | 3 |
| Asp-N endopeptidase | 10 |
| Asp-N endopeptidase + N-terminal Glu | 17 |
| CNBr | 4 |
| Chymotrypsin-high specificity (C-term to [FYW], not before P) | 4 |
| Chymotrypsin-low specificity (C-term to [FYWML], not before P) | 26 |
| Clostripain | 3 |
| Formic acid | 10 |
| Glutamyl endopeptidase | 7 |
| Hydroxylamine | 2 |
| LysC | 6 |
| LysN | 6 |
| NTCB (2-nitro-5-thiocyanobenzoic acid) | 3 |
| Pepsin (pH1.3) | 22 |
| Pepsin (pH > 2) | 22 |
| Proteinase K | 66 |
| Staphylococcal peptidase I | 6 |
| Thermolysin | 45 |
| Trypsin | 9 |
Swiss model中在线预测的Cu/Zn SOD蛋白质三级结构模型如图 7所示, 该模型显示太平洋真宽水蚤Cu/Zn SOD成熟肽有2个明显的α螺旋, 以及若干β折叠区等组成, 模型中标出两个α螺旋, 4个主要β折叠区, 以及C末端与N末端区。
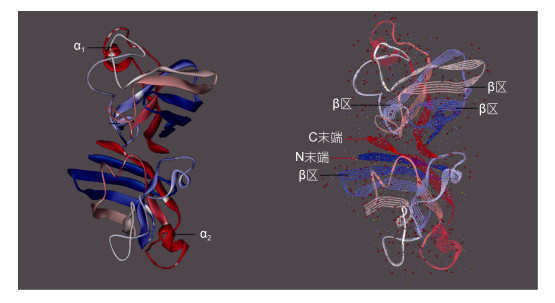 |
| 图 7 Cu/Zn SOD蛋白质三级结构预测图 Fig. 7 The 3 dimensional structures of Cu/Zn SOD domain predicted by Cn3D-4.1 |
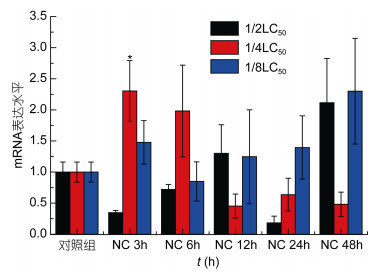
通过real time PCR方法对Ep.Cu/Zn SOD基因在重金属铬和镍, 及其联合胁迫下的表达情况进行检测, 以太平洋真宽水蚤18S rRNA基因作为内参基因, 选取正常组太平洋真宽水蚤的表达量作为参照, 每个处理下做了3个重复, 通过2–△△Ct方法计算相对表达量, 同时用SPSS 22.0软件进行显著性分析。实验结果表明, Ep. Cu/Zn SOD基因在两种重金属及其联合胁迫下均有表达, 但不同时间段以及不同浓度下的表达量高低有所不同(图 8, 图 9, 图 10)。
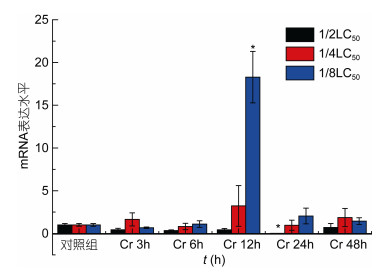 |
| 图 8 铬暴露下Ep. Cu/Zn SOD mRNA表达量水平 Fig. 8 The mRNA expression levels of Ep. Cu/Zn SOD under Cr exposure 注:柱上线表示平均值±标准差, ‘*’表示差异显著(P < 0.05, n=3) |
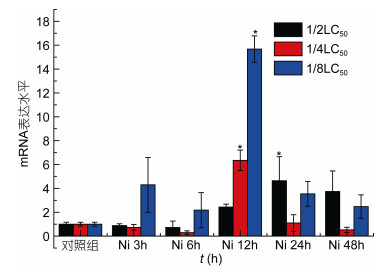 |
| 图 9 镍暴露下Ep. Cu/Zn SOD mRNA表达量水平 Fig. 9 The mRNA expression levels of Ep. Cu/Zn SOD under Ni exposure 注:柱上线表示平均值±标准差, ‘*’表示差异显著(P < 0.05, n=3) |
 |
| 图 10 铬与镍联合暴露下Ep. Cu/Zn SOD mRNA表达量水平 Fig. 10 The mRNA expression levels of Ep. Cu/Zn SOD under Cr-Ni exposure 注:柱上线表示平均值±标准差, ‘*’表示差异显著(P < 0.05, n=3) |
分析结果如下: Ep.Cu/Zn SOD基因mRNA表达水平铬胁迫下1/2LC50各个时间段与实验对照组相比, 峰值降低, 尤其在24小时有显著变化, 1/4LC50与1/8LC50均在12小时表达量达到峰值, 尤其1/8LC50表达特别显著(图 8); 镍胁迫下, 12小时1/4LC50与1/8LC50低浓度表达量峰值, 24小时1/2LC50高浓度达到峰值(图 9)。铬和镍联合胁迫下, 1/4LC50浓度总体呈现先上升后下降的趋势, 在3小时表达量达到峰值。1/2LC50与1/8C50随着时间变化总体呈现逐步上升的趋势, 其中在48小时达到峰值(图 10)。Ep.Cu/Zn SOD基因在两种重金属及其联合胁迫下表达量结果表明:铬对Cu/Zn SOD表达的抑制作用要明显的高于镍, 及其两者联合; 联合胁迫明显的低于铬和镍单一胁迫, 表明两种金属有一定的相互抑制作用, 总体表现:金属胁迫下Ep.Cu/Zn SOD基因在低浓度下的表达量高于高浓度下的表达量, 高浓度组金属胁迫对基因的表达抑制性较强, 表明重金属离子对Ep.Cu/Zn SOD表达水平的抑制率高浓度高于低浓度; 金属胁迫下, 浓度效应与时间效应趋于明显。
3 讨论正常生物体内氧自由基的产生和清除处于动态平衡中, 在过程中会出现大量的活性氧自由基, 超出自身的清除能力因此受到伤害, 其中就有Cu/Zn SOD在内的抗氧化体系。本研究中得到的序列共837个核苷酸, 编码152个氨基酸, 5′非编码区长297bp, 3′非编码区长84bp, 分子量约为15.050kDa, 理论等电点为5.73。常见得铜锌超氧化物歧化酶分为两种:一种是胞外铜锌超氧化物歧化酶, 另一种是胞内铜锌超氧化物歧化酶和(Zelko et al, 2002)。胞外铜锌超氧化物歧化酶一般在组织细胞外的基质中存在, 存在176—251个氨基酸, 其中N末端有一小段信号肽; 胞内铜锌超氧化物歧化酶主要存在于细胞质及细胞核内(Okado-Matsumoto et al, 2001), 存在147—167个氨基酸, 且N末端没有发现信号肽; 通过本实验研究获取太平洋真宽水蚤铜锌超氧化物歧化酶具有152个氨基酸, 并且在N末端没有发现信号肽, 推测太平洋真宽水蚤铜锌超氧化物歧化酶为胞内铜锌超氧化物歧化酶。胞内铜锌超氧化物歧化酶最重要的特征就是相似性和保守性(胡宝庆等, 2011), 就如4个Zn2+结合位点(His-61, -69, -78, Asp-81)和4个Cu2+结合位点(His-44, -46, -61, -118), 其中“咪唑桥”结构的形成就是在His-61上Cu2+与Zn2+相连接构成的; Cys-55与Cys-144组合为一对, 保持酶结构稳定性二硫键具有重要的影响(Ciriolo et al, 2001); 它是有2个α螺旋和8个反向平行的β折叠构成β桶状结构(Tainer et al, 1983); 两段保守的铜锌超氧化物歧化酶蛋白家族标签序列136GNAGGRAACGVI147和42GFHIHQFGDNT52; 接近相同的C末端尾序列(GVIGT)和N末端头序列(QAVCVL) (Li et al, 2011), 这些都在太平洋真宽水蚤铜锌超氧化物歧化酶中全部存在。通过比较其他甲壳类动物序列表明, 太平洋真宽水蚤Cu/Zn SOD cDNA的氨基酸序列与其他甲壳动物的一致性较高; 存在8个蛋白翻译后修饰位点及蛋白家族标签序列, 无跨膜结构域与信号肽。说明, 氨基酸序列与其他甲壳动物Cu/Zn SOD序列的同源性较高, 存在8个蛋白翻译后修饰位点及蛋白家族标签序列。为了比较Cu/Zn SOD基因的种系发育情况, 使用Maximum Likelihood method法构建系统发育树, 与太平洋纺锤水蚤(Acartia pacifica)相似度最高, 表明亲缘关系最近; 与贝类(Shellfish)系统进化发育树聚为一簇, 与鱼类的进化关系较远。结果表现出Cu/Zn SOD的系统进化属内相近性相似, 推测与Cu/Zn SOD基因高度保守有关。
本研究通过荧光定量PCR法(Heid et al, 1996)检测太平洋真宽体内Cu/Zn SOD基因在不同金属胁迫下的表达情况, 发现该基因在两种金属单一与联合胁迫下均有表达, 这是由于水中的有害物质能够造成生物体内产生活性氧分子(ROS)和氧自由基, 破坏生物体内的细胞和组织, 使氧化压力不断变化。如果细胞和组织受到严重破坏, 氧化压力超过自身机体的调节能力时, SOD等抗氧化酶的活性不会再有变化, ROS没有及时清除就会对机体造成不同程度损伤, 氧化压力如果连续不断或者在短时间内到达相对强度, 生物体正常代谢就会受到影响, 抗氧化酶活性就有可能会降低, 甚至失去活性。当细胞和组织受到轻度破坏时, 为清理因受到污染物胁迫而新产生的ROS, SOD等抗氧化酶的活性就会加强, 这是机体自身正常的应激反应(王凡等, 2007; 袁晓倩等, 2007; 刘海芳等, 2009)。因此, 生物体中SOD活性变化规律和重金属污染物胁迫之间, 通常会共同表现出相应的时间-效应关系和浓度-效应关系, 就像重金属毒性实验中常见的先不断升高然后逐渐下降的趋势, 或者更加普遍的低浓度诱导高浓度相互抑制现象(袁晓倩等, 2007), 表明Cu/Zn SOD基因是一种丰度较高、表达广泛的蛋白, 且单一胁迫下均在12小时表达量最高, 在研究污染物特别是重金属对生物体中SOD活性变化时, SOD活性受污染物浓度以及暴露时间的长短会出现先上升然后下降的变化规律是很常见的。这就是生物体中的SOD在污染物暴露前期产生“毒物兴奋效应”(Stebbing 1982; 杨丽华等, 2003; 陈志鑫, 2010), 当激活SOD后, 诱导表现更加明显, 活性会随之变化上升, 等到暴露时间的不断变长, “毒物兴奋效应”就会消失, 受污染物影响ROS会不断产生, 生理机能受到损害, SOD活性就会受到抑制。这与已知的脊椎动物以及无脊椎动物体内Cu/Zn SOD分布情况相类似; 联合胁迫下两种金属胁迫之间存在一定拮抗作用。在单一重金属离子组SOD表达量呈现出低中浓度组先升高后降低, 高浓度组先降低后升高在降低的低浓度诱导高浓度抑制的变化趋势。在某些情况下, SOD活性变化趋势更加复杂, 这是因为实验动物的选择和个体差异、污染物的特点以及作用机理、污染物暴露浓度和取样时间的不同、实验条件等都有关联。本研究不仅是单一的重金属离子的毒性胁迫实验, 还有联合毒性胁迫, 这样使实验动物处于不同污染物的联合毒性胁迫下, SOD活性的变化趋势相对比较复杂。研究结果表明, Cu/Zn SOD在阻止金属毒性胁迫、保护细胞免受氧化损伤方面发挥重要作用。推测在重金属氧化损伤中, Cu/Zn SOD基因可以有效消除过多的氧自由基, 高表达阻止和降低了细胞损伤。
现在大量研究证实了Cu/Zn SOD在氧化损伤等免疫防御中发挥显著作用, 但对桡足类免疫调控机制的了解资料不足; 针对太平洋真宽水蚤Cu/Zn SOD基因进行序列分析与表达检测, 为研究其他桡足类乃至甲壳类生物体内免疫、防御等提供了重要的参考资料。
4 结论本研究通过RACE技术首次克隆获得太平洋真宽水蚤Cu/Zn SOD基因全长cDNA序列, 并对其序列生物学特征进行了分析与探讨, 采用荧光定量RT-PCR技术检测了重金属胁迫下Cu/Zn SOD基因在太平洋真宽水蚤体内的差异表达, 研究结果为进一步探讨太平洋真宽水蚤Cu/Zn SOD基因相关功能性研究和SOD家族其他基因的克隆奠定基础, 为其他桡足类Cu/Zn SOD基因的研究及系统演化与分类系统提供借鉴; 同时为Cu/Zn SOD作为海洋水体污染监测与防控的有效生物标志物以及桡足类在海洋污染指示生物中的研究价值与研究意义等提供借鉴。
| 王凡, 赵元凤, 刘长发., 2007. Cu2+对牙鲆肌肉抗氧化防御系统的影响. 淡水渔业, 37(2): 27–29 |
| 刘海芳, 王凡., 2009. 重金属对水产动物污染的生物标志物的研究进展. 水产科学, 28(5): 299–302 |
| 杨丽华, 方展强, 郑文彪, 等, 2003. 镉对鲫鱼鳃和肝脏超氧化物歧化酶活性的影响. 安全与环境学报, 3(3): 13–16 |
| 陈志鑫., 2010. Hg2+、Pb2+、Cd2+对中华哲水蚤(Calanus sinicus)总超氧化物歧化酶(T-SOD)及线粒体COI基因影响的初步研究. 青岛:中国海洋大学硕士学位论文,: 20–22 |
| 郑清梅, 温茹淑, 韩春艳, 等, 2014. 草鱼铜锌超氧化物歧化酶全长cDNA的克隆与组织表达. 基因组学与应用生物学, 33(6): 1236–1246 |
| 胡宝庆, 谢彦海, 王新生, 等, 2011. 褶纹冠蚌胞内Cu/Zn-SOD的cDNA克隆及蛋白质三维结构预测. 华中农业大学学报, 30(2): 235–242 |
| 赵元凤, 吕景才, 李丹彤, 等, 2003. 海洋污染对毛蚶超氧化物歧化酶影响的研究. 海洋学报, 25(3): 77–82 |
| 袁晓倩, 许晓路, 申秀英., 2007. 三丁基锡对水生生物毒理学影响的研究进展. 浙江师范大学学报(自然科学版), 30(3): 326–330 |
| Amano A, Shizukuishi S, Tsunemitsu A, et al, 1990. The primary structure of superoxide dismutase purified from anaerobically maintained Bacteroides gingivalis. FEBS Letters, 272(1-2): 217–220 DOI:10.1016/0014-5793(90)80488-5 |
| Arhontaki K, Eliopoulos E, Goulielmos G, et al, 2002. Functional constraints of the Cu, Zn superoxide dismutase in species of the Drosophila melanogaster subgroup and phylogenetic analysis. Journal of Molecular Evolution, 55(6): 745–756 DOI:10.1007/s00239-002-2370-9 |
| Bodył A, Mackiewicz P., 2007. Analysis of the targeting sequences of an iron-containing superoxide dismutase (SOD) of the dinoflagellate Lingulodinium polyedrum suggests function in multiple cellular compartments. Archives of Microbiology, 187(4): 281–296 DOI:10.1007/s00203-006-0194-5 |
| Chary P, Hallewell R A, Natvig D O., 1990. Structure, exon pattern, and chromosome mapping of the gene for cytosolic copper-zinc superoxide dismutase (sod-1) from Neurospora crassa. The Journal of Biological Chemistry, 265(31): 18961–18967 |
| Ciriolo M R, Battistoni A, Falconi M, et al, 2001. Role of the electrostatic loop of Cu, Zn superoxide dismutase in the copper uptake process. The FEBS Journal, 268(3): 737–742 |
| Fattman C L, Schaefer L M, Oury T D., 2003. Extracellular superoxide dismutase in biology and medicine. Free Radical Biology and Medicine, 35(3): 236–256 DOI:10.1016/S0891-5849(03)00275-2 |
| Fridovich I., 1986. Superoxide dismutases. Advances in Enzymology and Related Areas of Molecular Biology, 58(6): 61–79 |
| Gressler V, Yokoya N S, Fujii M T, et al, 2010. Lipid, fatty acid, protein, amino acid and ash contents in four Brazilian red algae species. Food Chemistry, 120(2): 585–590 DOI:10.1016/j.foodchem.2009.10.028 |
| Hansen B H, R mma S, Garmo A, et al, 2006. Antioxidative stress proteins and their gene expression in brown trout (Salmo trutta) from three rivers with different heavy metal levels. Comparative Biochemistry and Physiology Part C:Toxicology & Pharmacology, 143(3): 263–274 |
| Heid C A, Stevens J, Livak K J, et al, 1996. Real time quantitative PCR. Genome Research, 6(10): 986–994 DOI:10.1101/gr.6.10.986 |
| Li J M, Su Y L, Gao X L, et al, 2011. Molecular characterization and oxidative stress response of an intracellular Cu/Zn superoxide dismutase (CuZn SOD) of the whitefly, Bemisia tabaci. Archives of Insect Biochemistry and Physiology, 77(3): 118–133 DOI:10.1002/arch.v77.3 |
| Lin M, Lin M T, Lin C T., 2002. Copper/zinc-superoxide dismutase from lemon cDNA and enzyme stability. Journal of Agricultural and Food Chemistry, 50(25): 7264–7270 DOI:10.1021/jf0207070 |
| Nahrgang J, Camus L, Carls M G, et al, 2010. Biomarker responses in polar cod (Boreogadus saida) exposed to the water soluble fraction of crude oil. Aquatic Toxicology, 97(3): 234–242 DOI:10.1016/j.aquatox.2009.11.003 |
| Okado-Matsumoto A, Fridovich I., 2001. Subcellular distribution of superoxide dismutases (SOD) in rat liver Cu, Zn-SOD in mitochondria. Journal of Biological Chemistry, 276(42): 38388–38393 DOI:10.1074/jbc.M105395200 |
| Putnam C D, Arvai A S, Bourne Y, et al, 2000. Active and inhibited human catalase structures:ligand and NADPH binding and catalytic mechanism. Journal of Molecular Biology, 296(1): 295–309 DOI:10.1006/jmbi.1999.3458 |
| Shen J, Subramaniam S, Wong C F, et al, 1989. Superoxide dismutase:fluctuations in the structure and solvation of the active site channel studied by molecular dynamics simulation. Biopolymers, 28(12): 2085–2096 DOI:10.1002/(ISSN)1097-0282 |
| Stebbing A R D., 1982. Hormesis-the stimulation of growth by low levels of inhibitors. Science of the Total Environment, 22(3): 213–234 DOI:10.1016/0048-9697(82)90066-3 |
| Tainer J A, Getzoff E D, Richardson J S, et al, 1983. Structure and mechanism of copper, zinc superoxide dismutase. Nature, 306(5940): 284–287 DOI:10.1038/306284a0 |
| Whitfield A K, Elliott M., 2002. Fishes as indicators of environmental and ecological changes within estuaries:a review of progress and some suggestions for the future. Journal of Fish Biology, 61(SA): 229–250 |
| Zelko I N, Mariani T J, Folz R J., 2002. Superoxide dismutase multigene family:a comparison of the CuZn-SOD (SOD1), Mn-SOD (SOD2), and EC-SOD (SOD3) gene structures, evolution, and expression. Free Radical Biology and Medicine, 33(3): 337–349 DOI:10.1016/S0891-5849(02)00905-X |
 2018, Vol. 49
2018, Vol. 49


