中国海洋湖沼学会主办。
文章信息
- 胡丽萍, 姜黎明, 黄晓婷, 包振民. 2016.
- HU Li-Ping, JIANG Li-Ming, HUANG Xiao-Ting, BAO Zhen-Min. 2016.
- 基于16S rDNA基因序列探讨引进物种紫扇贝(Argopecten purpuratus)在海湾扇贝属(Argopecten)中的分类地位
- PHYLOGENETIC POSITION OF ARGOPECTEN PURPURATUS IN GENUS ARGOPECTEN INFERRED BY MITOCHONDRIAL 16S rDNA GENE SEQUENCES
- 海洋与湖沼, 47(6): 1149-1157
- Oceanologia et Limnologia Sinica, 47(6): 1149-1157.
- http://dx.doi.org/10.11693/hyhz20160600120
-
文章历史
- 收稿日期:2016-06-08
- 收修改稿日期:2016-07-13
2. 烟台市水产研究所 烟台 264003;
3. 山东东方海洋科技股份有限公司 烟台 264000
2. Yantai Fisheries Research Institute, Yantai 264003, China;
3. Shandong Oriental Ocean SCI-TECH CO., LTD, Yantai 264000, China
紫扇贝(Argopecten purpuratus, Lamarck, 1819)是原产于南太平洋的一种优质扇贝, 具有出肉率高、味道鲜美、生长快速、抗逆性强、足丝发达等特点, 当地生长14-16个月可达商品规格(9cm), 经济价值高, 其加工产品在美国和欧洲深受欢迎, 智利和秘鲁等地已开展大规模的人工养殖。目前对该扇贝的研究主要集中在生理生态条件(Navarro et al, 2000, 2006; Díaz et al, 2006; Avendaño et al, 2008)、壳色遗传(Winkler et al, 2001)等方面, 分子生物学的研究鲜有报道。青岛农业大学王春德教授于2008年从秘鲁引进紫扇贝并繁育成功(王春德等, 2009)。但紫扇贝适宜养殖温度范围较窄(12-26°C), 在我国北方海区无法越冬, 山东以南海区不能度夏, 这给该扇贝的养殖推广带来了困难。
海湾扇贝(Argopecten irradians irradians, Lamarck, 1819), 原产于美国大西洋沿岸, 于1982年从美国引进中国(张福绥等, 1986), 以生长快著称, 且耐温范围较广(-1-31°C), 已成为我国主要扇贝养殖品种(张福绥等, 2000)。考虑到紫扇贝和海湾扇贝同属于海湾扇贝属(Argopecten), 且两者存在互补性状, 王春德等(2009)尝试将引进的紫扇贝和海湾扇贝杂交, 成功获得具有显著杂种优势的杂交种, 不仅生长迅速、抗逆性也明显增强, 特别是在山东地区能够顺利度夏和越冬, 显示了巨大的产业潜力。然而, 对于这一全新引进的扇贝物种, 对其遗传学及种质资源的评估资料仍处短缺状态。
线粒体DNA (mtDNA)作为核外遗传物质, 具有分子量小、结构简单、进化速度快等特点, 适合于种群遗传分析及分子系统进化的研究(Meyer, 1993; Canapa et al, 2000; Sunetha et al, 2000; 辛俭等, 2014)。mtDNA中的16SrDNA序列在种内遗传多样性和种间系统学关系的研究中占有重要的地位(Foighil et al, 1995; Schnelder et al, 1999; Lakra et al, 2009)。本研究通过测定紫扇贝和海湾扇贝mtDNA 16S rDNA基因片段序列, 分析了紫扇贝在中国繁殖后代中的遗传变异趋势, 并计算了两个扇贝物种的基因差异和遗传距离。结合GenBank中海湾扇贝属及我国本土扇贝物种--栉孔扇贝相应片段进行比较并进行系统进化分析, 从分子水平上了解紫扇贝的遗传背景及其系统进化地位, 以期为扇贝种质资源保护及杂交育种策略提供基础性资料。
1 材料与方法 1.1 试验材料紫扇贝和海湾扇贝均于2010年10月下旬取自山东省青岛市胶南扇贝养殖场, 养殖水环境条件为:水温16°C, 盐度30, pH8.2。每个物种各取10个个体, 活体解剖取扇贝闭壳肌, 于液氮冷冻后转-80°C超低温冰箱保存备用。其中紫扇贝为引种后繁殖的子一代个体, 平均壳高4.81cm, 平均体重27.36g, 海湾扇贝是从当地养殖场中随机选取个体, 平均壳高4.63cm, 平均体重26.62g。
1.2 方法 1.2.1 基因组DNA的提取和检测取约100mg扇贝闭壳肌组织, 采用标准的酚-氯仿方法(Sambrook et al, 1989)提取基因组DNA, 将乙醇沉淀后的基因组DNA溶解于100μL的TE溶液中, 用紫外分光光度计测定其浓度和纯度, 然后将基因组DNA稀释至50ng/μL, 4°C下保存备用。
1.2.2 16S rDNA的序列扩增及序列测定实验用16S rDNA引物序列为: 16SF (5′-CGCCTGTTTATCA AAAACAT-3′)和16SR (5′-CCGGTCTGAACTCAGAT CACGT-3′)(Canapa et al, 2000), 引物由上海生工公司合成。PCR反应总体积为50μL, 体系中包括: 1×PCR缓冲液, 1.5mmol/L的MgCl2, 0.2mmol/L的dNTP, 1μmol/L的正反向引物, 1U的Taq DNA聚合酶(TAKARA), 100ng的DNA模板。PCR扩增反应程序: 94°C, 5min预变性; 然后30个循环, 94°C, 45s变性, 55°C, 45s退火, 72°C, 1min延伸; 最后72°C进行5min延伸, 置于4°C保存。
取5μL扩增产物经1%的琼脂糖凝胶电泳, 溴化乙锭染色后紫外观察拍照。剩余PCR产物采用Sangon PCR产物纯化试剂盒进行回收及纯化, 具体方法按试剂盒说明书步骤进行。纯化后的DNA片段测序采用ABI PRISM 3730XL测序仪完成, 测序引物为PCR扩增引物。
1.2.3 序列分析用BioEdit软件去掉序列两端不可信部分, 采用Clustalx1.81进行序列对位排序, 使用Genedoc软件进行人工校正。然后应用MEGA5.1软件计算分子变异参数如多态性位点, 转换、颠换及插入缺失位点等, 并根据Kimura双参数方法计算遗传距离。
1.2.4 序列下载从GenBank下载7种海湾扇贝属扇贝以及外群栉孔扇贝的16S rDNA同源序列, 序列信息见表 1。
| 种类 | GenBank序列号 |
| Argopecten irradians | AF526235 |
| (Lamarck, 1819) | |
| Argopecten irradians irradians | AF362383 |
| (Lamarck, 1819) | |
| Argopecten irradians concentricus | AF362384 |
| (Say, 1822) | |
| Argopecten purpuratus | EU379472 |
| (Lamarck, 1819) | |
| Argopecten nucleus | EU379461 |
| (Born, 1780) | |
| Argopecten gibbus | EU379443 |
| (Linnaeus, 1758) | |
| Argopecten ventricosus | AJ972429 |
| (Sowerby, 1842) | |
| Chlamys farreri | GU119980 |
| (Jones et Preston, 1940) |
利用MEGA5.1软件并根据Kimura双参数模型计算遗传距离, 以栉孔扇贝作外群, 分别采用邻接(neibour-joining, NJ)法和最大似然(maximum-likelihood, ML)法对海湾扇贝属物种构建分子系统树, 采用重抽样法(Bootstrap)1000检验系统树分枝的置信度。
2 结果 2.1 紫扇贝与海湾扇贝DNA序列组成及变异经PCR扩增, 分别得到了两种扇贝清晰的16S rDNA基因片段扩增产物(图 1)。经序列测定, 大小均为542bp (含引物序列)(图 2)。通过MEGA5.1对两种扇贝16S rDNA基因片段序列的平均A、T、G、C含量进行计算, 结果见表 2。紫扇贝与海湾扇贝的碱基组成差异不大, 相应碱基比例相当, 且A+T所占的比例略大于G+C, 分别为紫扇贝54.2%和海湾扇贝55.1%。
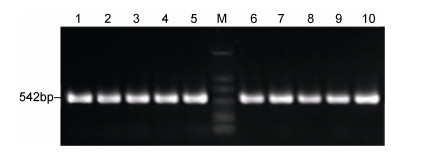 |
| 图 1 紫扇贝和海湾扇贝16S rDNA基因片段的PCR扩增产物 Fig. 1 PCR amplified products of 16S rDNA gene fragment in Argopecten purpuratus and Argopecten irradians irradians 注: M: DL2000 Marker; 泳道1-5:紫扇贝; 泳道6-10:海湾扇贝 |
 |
| 图 2 紫扇贝(a)和海湾扇贝(b)的16S rDNA基因片段核苷酸序列(主导类型) Fig. 2 The nucleotide sequences of 16S rDNA gene fragments inA. purpuratus (a) and A. i. irradians(b) |
| 物种 | 实验个体数 | 单倍型数 | 片段长度(bp) | 平均碱基含量(%) | ||||
| T | C | A | G | A+T | ||||
| 紫扇贝(A. purpuratus) | 10 | 6 | 542 | 29.7 | 18.1 | 24.5 | 27.7 | 54.2 |
| 海湾扇贝(A. i. irradians) | 10 | 6 | 542 | 29.5 | 17.6 | 25.6 | 27.4 | 55.1 |
对紫扇贝10个个体, 海湾扇贝10个个体, 应用ClustalX1.81和MEGA5.1进行种内及种间序列的比对分析。种内序列分析表明, 紫扇贝和海湾扇贝种内不同个体序列间的变异均为转换和颠换导致, 且转换位点数多于颠换位点数, 无插入/缺失变异位点。紫扇贝种内序列排序后共检测到6个变异核苷酸位点, 其中转换核苷酸位点数为4个, 颠换位点数为2个; 共检测到6种单倍型, 其中主导类型A为5个个体共有, 其余类型仅为某一个体独有(表 3)。海湾扇贝种内共检测到10个变异核苷酸位点, 其中转换位点7个, 颠换位点3个; 检测到单倍型共6种, 其中主导类型A为5个个体共有, 其余类型仅为某一个体独有(表 4)。
| 单倍型 | A | B | C | D | E | F | |
| 核苷酸变异位点 | 41 | A | . | . | . | G | G |
| 57 | T | A | . | . | . | . | |
| 262 | G | . | . | A | A | A | |
| 287 | T | . | C | . | . | . | |
| 474 | A | . | . | . | . | T | |
| 514 | A | . | . | . | . | G | |
| 单倍型个体数 | 5 | 1 | 1 | 1 | 1 | 1 |
| 单倍型 | A | B | C | D | E | F | |
| 核苷酸变异位点 | 184 | G | A | A | . | A | A |
| 201 | T | . | . | G | . | . | |
| 209 | T | C | . | . | . | . | |
| 281 | T | . | A | . | . | . | |
| 285 | A | G | G | . | G | G | |
| 324 | T | . | . | . | . | C | |
| 397 | A | . | . | . | . | G | |
| 408 | A | . | . | . | . | G | |
| 410 | C | . | . | T | . | . | |
| 513 | A | C | . | . | . | . | |
| 单倍型个体数 | 5 | 1 | 1 | 1 | 1 | 1 |
对紫扇贝和海湾扇贝两物种间的变异碱基进行统计, 共得到41个变异位点, 且为转换/颠换位点, 无插入/缺失变异。其中转换位点为23个, 颠换位点为16个, 既有转换又有颠换的位点为2个。两物种序列的同源性为92.4%。
2.2 海湾扇贝属物种遗传分化紫扇贝和海湾扇贝种内遗传距离范围分别为0.000-0.009和0.000-0.013, 平均为0.003和0.005。海湾扇贝属内种间遗传距离见表 5。所有物种间的遗传距离为0.007-0.120。最小遗传距离出现在A. irradians和A. nucleus之间(0.007), 而A. i. concentricus与A. ventricosus之间的遗传距离最大, 达到0.120。与紫扇贝A. purpuratus遗传距离最小的是A. ventricosus, 遗传距离为0.031, 而与海湾扇贝A. i. irradians遗传距离最小的是A. nucleus, 遗传距离为0.008, 甚至在海湾扇贝种内遗传距离范围内(0.000-0.013)。
| A. irradians | A. i. concentricus | A. i. irradians | A. nucleus | A. gibbus | A. purpuratus | A. ventricosus | |
| A. irradians | |||||||
| A. i. concentricus | 0.011 | ||||||
| A. i. irradians | 0.009 | 0.020 | |||||
| A. nucleus | 0.007 | 0.018 | 0.008 | ||||
| A. gibbus | 0.050 | 0.061 | 0.050 | 0.047 | |||
| A. purpuratus | 0.088 | 0.100 | 0.088 | 0.085 | 0.088 | ||
| A. ventricosus | 0.107 | 0.120 | 0.107 | 0.105 | 0.100 | 0.031 |
基于16S rDNA基因序列, 应用NJ和ML分别重建了系统发生树(图 3), 得到的海湾扇贝属物种系统发生树拓扑结构几近一致, 大分支节点的支持率很高。
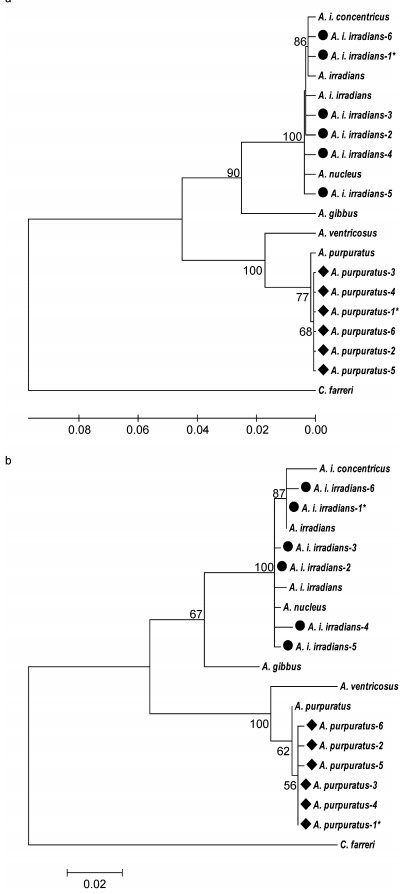 |
| 图 3 应用MEGA5.1软件中邻接法(NJ, a)和最大似然法(ML, b)构建基于16S rDNA序列的海湾扇贝属系统发生树 Fig. 3 The neighbor-joining phylogenetic tree (a) and maximum-likelihood phylogenetic tree (b) constructed based on 16S rDNA sequences ofArgopecten species using MEGA5.1 注:分支处数字为大于50%的Bootstrap值(1000次重复); ●标记本研究供试海湾扇贝; ◆标记本研究供试紫扇贝; *表示包含5个完全相同序列的个体 |
除外群栉孔扇贝(C. farrerii)外, 7个海湾扇贝属物种明显分为2个单系群, 其中一个单系群包括所有的紫扇贝A. purpuratus和扇贝A. ventricosus, 且置信度为100%;另一个单系群包括了其余5个海湾扇贝属物种A. irradians、A. i. concentricus、A. i. irradians(包括供试6个海湾扇贝)、A. nucleus和A. gibbus。该单系群又分为两个分支, 一个分支仅包含A. gibbus一个种, 其余海湾扇贝属物种A. irradians、A. i. concentricus、A. i. irradians和A.nucleus聚为另一分支。在NJ树和ML树中, 均为A. irradians、A. i. concentricus和部分A. i. irradians个体序列首先聚为一支, 再与其余测试A. i. irradians个体以及A. nucleus聚类。
3 讨论 3.1 16S rDNA序列在贝类遗传学研究中的应用DNA分子测序技术的快速发展, 使得线粒体(mtDNA)基因序列的多态性分析更为简单和方便。mtDNA上不同基因因其进化速率不同, 遗传变异解析能力也不同, 如16S rDNA基因, 已被广泛应用于海洋贝类分子遗传多样性研究(Mahidol et al, 2007)、种质鉴定(Kong et al, 2009)及种属间的分子系统学研究(Saavedra et al, 2006; Lakra et al, 2009)等。Boulding等(1993)通过对虾夷扇贝一个养殖群体和两个野生群体样品进行16S rDNA基因分析, 检测到了丰富的遗传变异; 刘亚军等(2002)和苏天凤等(2005)分别在不同栉孔扇贝地理种群和近江牡蛎中也检测到了较高的遗传变异。李霞等(2009)通过对海扇贝、虾夷扇贝、栉孔扇贝和海湾扇贝16S rDNA同源序列的比对发现, 它们分别存在3、7、8、13个差异位点, 核苷酸变异位点较为丰富, 且碱基颠换和插入变异比例大于碱基转换。本研究中, 对紫扇贝和海湾扇贝的16S rDNA基因同源序列检测分析表明, 两者种内分别存在6和10个差异位点, 核苷酸变异也较丰富。与李霞等(2009)结论不同的是, 本研究中的紫扇贝和海湾扇贝同源序列间的变异均来自转换或者颠换, 无插入/缺失变异, 且转换位点多于颠换位点。这些变异位点可用于分子系统学和遗传学的分析。
3.2 扇贝16S rDNA碱基组成及变异分析在紫扇贝和海湾扇贝检测样品中, 4种碱基(A、T、G、C)在两个扇贝物种中的组成比例相当, 均为C碱基比例较低, 其余三种碱基含量较为接近。对本研究中海湾扇贝属所有扇贝物种的碱基组成分析后的结果与之一致, 且均表现为A+T含量高于G+C含量(A+T占比52.6%-55.5%), 这一结果与其他研究者在双壳贝类的16S rDNA、12S rDNA和COⅠ基因碱基组成分析中观察到的结果一致(Boulding et al, 1993; 孔晓瑜等, 2001; 李霞等, 2009)。另外, 在头足类(Anderson, 2000; Zheng et al, 2001)和甲壳类(高天祥等, 2000)等的线粒体基因碱基组成分析中也发现有类似的现象。推测mtDNA具有较高的A+T含量可能在无脊椎动物中是一种普遍现象。
从遗传学角度来说, 一个物种种内核苷酸变异值的大小在一定程度上反映了物种的遗传多样性, 而一个物种遗传多样性水平的高低与其适应环境、生存及进化能力是紧密关联的(刘亚军等, 2002)。遗传多样性水平降低可能会导致物种的适应性和生存能力下降, 并最终导致物种种质的衰退。本研究对紫扇贝和海湾扇贝16S rDNA基因片段序列的种内变异分析结果表明, 紫扇贝的遗传变异水平较海湾扇贝低, 这可能是由于紫扇贝试验样本作为子一代, 仅由少数几个引进亲本近交繁育而来, 遗传背景相对单一导致。
mtDNA上不同基因因其进化速率不同, 遗传变异解析能力也不同。在本研究中, 16S rDNA基因在紫扇贝和海湾扇贝种内个体及种间检测到的遗传变异较小, 要进行更为准确和全面的研究, 需要结合mtDNA其他的基因(如COI、Cytb等)序列得到多组序列信息, 甚至可以把mtDNA序列数据与核基因组DNA数据结合起来, 共同用于对物种遗传变异的分析, 从而获得较为客观、全面的物种遗传多样性数据。
3.3 海湾扇贝属各物种间的亲缘关系基于线粒体基因序列探讨物种亲缘关系的研究在双壳贝类研究中已非首例。Canapa等(2000)通过对扇贝科6种扇贝16S rDNA基因序列的比较分析, 探讨了这些扇贝物种的系统发生关系。Barucca等(2004)采用线粒体16S和12S rDNA基因序列的比较分析, 对多种扇贝科物种进行了亲缘关系分析, 其中涉及的海湾扇贝属物种仅有A. irradians。在Saavedra等(2006)基于16S和12S rDNA基因序列对美国多种双壳贝类的系统发生关系研究中, 包含的海湾扇贝属物种也仅有A. purpuratus和A. ventricosus。包含海湾扇贝属扇贝种类最多的是Puslednik等(2008)的研究, 该研究通过对两种线粒体基因(16S和12S rDNA)和一个核基因(H3组蛋白)的序列分析, 对扇贝46个物种构建了ML系统发生树。其中涉及的海湾扇贝属5种扇贝被分为两个大支, 其中一支包含A. purpuratus和A. ventricosus; 另外一支包含其余3个扇贝物种A. irradians、A. nucleus和A. gibbus, 且A. irradians和A. nucleus首先聚为一组, 这与我们的研究结果一致。然而, 对本研究中A. i. irradians(供试海湾扇贝)和A. i. concentricus的系统进化及分子遗传学分析在以往的研究中均无报道。
A. i. irradians(美国海湾扇贝)最早于1982年引进我国, 是原产于美国大西洋沿岸的一种海湾扇贝, 而A. i. concentricus(墨西哥湾扇贝)同样属于美国大西洋沿岸的海湾扇贝亚种, 于1991年被引入中国并被成功养殖(张福绥等, 1994, 2000)。本研究中, 物种遗传距离分析结果表明, A. i. irradians和A. i. concentricus与海湾扇贝A. irradians的遗传距离分别为0.009和0.011, 均处于海湾扇贝的种内遗传距离范围(0.000-0.013)之内, 而A. i. irradians与A. i. concentricus之间的遗传距离(0.020)却大于海湾扇贝种内的最大遗传距离, 由此可以做出推测: A. i. irradians与A. i. concentricus为海湾扇贝种(A. irradians)的两个不同地理种群, 因受不同环境条件影响而导致遗传物质发生改变, 从而分化为海湾扇贝的两个亚种。同样的, 从遗传距离角度考虑, A. nucleus(加勒比海扇贝)与A. irradians、A. i. irradians和A. i. concentricus的遗传距离分别为0.007、0.008和0.018, 在系统树构建中, A. nucleus与以上三者也最早聚在一起, 表现出与三种海湾扇贝具有非常近的亲缘关系, 仅从16S rDNA基因分析推测, A.nucleus还未达到种的分化程度, 可将其归属海湾扇贝的一个亚种, 然而该结论还需要更多的证据来证明。
3.4 杂交育种亲本的选择杂交育种是贝类新品种选育的基本方法之一, 也是培育优良品种的重要方法。迄今为止, 国内外已有一些贝类杂交的研究报道, 在获得优势杂种的工作中取得了一定成绩(Bower et al, 1997; 常亚青等, 2002; 刘小林等, 2003)。在国内, 很多学者对栉孔扇贝、虾夷扇贝、海湾扇贝以及华贵栉孔扇贝等多种经济扇贝物种进行了杂交尝试, 并对杂交子代基因组的组成和变异进行了研究(刘宪杰等, 2006; Huang et al, 2011)。杂交实验证明了扇贝间的杂交具有可行性, 但是, 获得的杂交子代却仅发育到早期幼虫阶段便夭折, 一直未能获得可成活的杂种成体扇贝。然而, 本研究中涉及的紫扇贝和海湾扇贝已被证实其杂交可获得具有明显杂种优势的子一代成体(Wang et al, 2011; Hu et al, 2013)。在杂交育种工作中, 亲本的选配在很大程度上决定了育种计划的成败。当两亲本遗传分化太大时, 可能会因为两者遗传物质不相容而无法成功获得杂交后代; 而当两亲本遗传分化不明显时, 又会无法产生杂种优势。已有的扇贝杂交试验也证实了这一点, 对栉孔扇贝、虾夷扇贝、海湾扇贝以及华贵栉孔扇贝间的杂交均为属间杂交, 两亲本的遗传分化较大; 而紫扇贝和海湾扇贝的杂交尝试却为属内种间杂交, 亲本遗传分化相对较小, 两者遗传物质相容, 并可稳定遗传给子代。
通过遗传分化及聚类分析可以看出, 紫扇贝(A.purpuratus)和海湾扇贝(A. i. irradians)被分在两个不同的单系群中, 且两者的遗传距离为0.088, 与之类似, 遗传差异稍大的紫扇贝(A. purpuratus)和墨西哥湾扇贝(A. i. concentricus)(遗传距离为0.100)已成功进行杂交试验, 且杂种后代优势明显(南乐红等, 2012)。聚在同一个单系群中的扇贝遗传距离均小于紫扇贝和海湾扇贝的遗传距离(0.088), 也即遗传分化较小。鉴于遗传差异小的亲本较易实现杂交, 可考虑将聚在同一个单系群中的扇贝物种用作亲本进行杂交育种尝试。
4 结论本研究利用线粒体16S rDNA序列分析了我国两种引进海湾扇贝属物种的遗传变异水平, 并结合已发表相关序列信息对海湾扇贝属物种进行分子系统发生分析。研究结果为海湾扇贝属物种进化提供参考, 为海湾扇贝属物种的引种以及通过杂交手段开展育种工作提供依据。此外, 为了更加准确地阐述紫扇贝和海湾扇贝这两个引进物种的进化关系以及遗传变异水平, 综合利用多种序列和多种分析手段进行讨论将是我们下一步需要展开的工作。
| 王春德, 刘宝忠, 李继强, 等, 2009. 紫扇贝与海湾扇贝种间杂交的研究. 海洋科学, 33 (10) : 84–87, 91 |
| 孔晓瑜, 刘亚军, 喻子牛等, 2001.栉孔扇贝和海湾扇贝线粒体DNA 16S rRNA基因片段序列研究.见:中国贝类学会主编.贝类学论文集.北京:海洋出版社, 59-63 |
| 刘小林, 常亚青, 相建海, 等, 2003. 栉孔扇贝不同种群杂交效果的初步研究I. 中国种群与俄罗斯种群的杂交.海洋学报, 25 (1) : 93–99 |
| 刘亚军, 喻子牛, 姜艳艳, 等, 2002. 栉孔扇贝16S rRNA基因片段序列的多态性研究. 海洋与湖沼, 33 (5) : 477–483 |
| 刘宪杰, 常亚青, 2006. 扇贝种间杂交的可行性及幼体早期发育的初步研究. 大连水产学院学报, 21 (4) : 346–349 |
| 苏天凤, 江世贵, 周发林, 等, 2005. 近江牡蛎16S rRNA基因片段序列变异分析. 高技术通讯, 15 (2) : 100–103 |
| 李霞, 秦艳杰, 初冠囡, 等, 2009. 采用16S rRNA基因序列分析海扇贝与3种扇贝的亲缘关系. 大连水产学院学报, 24 (3) : 269–273 |
| 辛俭, 张玉荣, 徐冬冬, 等, 2014. 基于线粒体16S rDNA和COΙ基因探讨中国近海黄姑鱼类的分子系统进化关系. 海洋与湖沼, 45 (2) : 307–313 |
| 张福绥, 何义朝, 亓铃欣, 等, 1994. 墨西哥湾扇贝的引种和子一代苗种培育. 海洋与湖沼, 25 (4) : 372–377 |
| 张福绥, 何义朝, 刘祥生, 等, 1986. 海湾扇贝引种、育苗及试养. 海洋与湖沼, 17 (5) : 367–374 |
| 张福绥, 何义朝, 杨红生, 2000. 海湾扇贝引种工程及其综合效应. 中国工程科学, 2 (2) : 30–35 |
| 南乐红, 张金盛, 丰玮, 等, 2012. 紫扇贝和墨西哥湾扇贝种间杂交的初步研究. 中国农学通报, 28 (20) : 131–135 |
| 高天祥, 张秀梅, 吉崎悟朗, 等, 2000. 中华绒螯蟹和日本绒螯蟹线粒体12S rRNA基因序列研究. 中国海洋大学学报, 30 (1) : 43–47 |
| 常亚青, 刘小林, 相建海, 等, 2002. 栉孔扇贝中国种群与日本种群杂交一代的早期生长发育. 水产学报, 26 (5) : 385–390 |
| Anderson F E, 2000. Phylogeny and historical biogeography of the Loliginid squids (Mollusca:Cephalopoda) based on mitochondrial DNA sequence data. Molecular Phylogenetics and Evolution, 15 (2) : 191–214 DOI:10.1006/mpev.1999.0753 |
| Avendaño M, Cantillánez M, Thouzeau G, 2008. Effects of water depth on survival and growth of Argopecten purpuratus (Lamarck, 1819) spat in northern Chile.. Aquaculture International, 16 (5) : 377–391 DOI:10.1007/s10499-007-9151-9 |
| Barucca M, Olmo E, Schiaparelli S, et al, 2004. Molecular phylogeny of the family Pectinidae (Mollusca:Bivalvia) based on mitochondrial 16S and 12S rRNA genes. Molecular Phylogenetics and Evolution, 31 (1) : 89–95 DOI:10.1016/j.ympev.2003.07.003 |
| Boulding E C, Boom J D C, Beckenbach A T, 1993. Genetic variation in one bottlenecked and two wild populations of the Japanese scallop (Patinopecten yessoensis):empirical parameter estimates from coding regions of mitochondrial DNA. Canadian Journal of Fisheries and Aquatic Sciences, 50 (6) : 1147–1157 DOI:10.1139/f93-131 |
| Bower S M, Blackbourn J, Meyer G R, 1997. A new and unusual species of Perkinsus pathogenic to cultured Japanese scallops, Patinopecten yessoensis, in British Columbia, Canada. Journal of Shellfish Research, 16 : 333 |
| Canapa A, Barucca M, Marinelli A, et al, 2000. Molecular data from the 16S rRNA gene for the phylogeny of Pectinidae (Mollusca:Bivalvia). Journal of Molecular Evolution, 50 (1) : 93–97 DOI:10.1007/s002399910010 |
| Díaz M A, Silva M C, Forner J P, 2006. Effect of immersion time of cultch on spatfall of the scallop Argopecten purpuratus (Lamarck 1819) in the Marine Reserve at La Riconada, Antofagasta, Chile. Aquaculture International, 14 (3) : 267–283 DOI:10.1007/s10499-005-9033-y |
| Foighil DÓ, Gaffney P M, Hilbish T J, 1995. Differences in mitochondrial 16S ribosomal gene sequences allow discrimination among American. gigas (Thunberg) C.ariakensis Wakiya]oyster species.Journal of Experimental Marine Biology and Ecology, 192 (2) : 211–220 |
| Hu L P, Huang X T, Mao J X, et al, 2013. Genomic characterization of interspecific hybrids between the scallops Argopecten purpuratus and Airradians irradians. PLoS One, 8 (4) : e62432 DOI:10.1371/journal.pone.0062432 |
| Huang X T, Bi K, Hu L P, et al, 2011. Fertilization and cytogenetic examination of interspecific reciprocal hybridization between the scallops, Chlamys farreri and Mimachlamys nobilis. PLoS One, 6 (11) : e27235 DOI:10.1371/journal.pone.0027235 |
| Kong L F, Li Q, 2009. Genetic evidence for the existence of cryptic species in an endangered clam Coelomactra antiquata. Marine Biology, 156 (7) : 1507–1515 DOI:10.1007/s00227-009-1190-5 |
| Lakra W S, Goswami M, Gopalakrishnan A, 2009. Molecular identification and phylogenetic relationships of seven Indian Sciaenids (Pisces:Perciformes, Sciaenidae) based on 16S rRNA and cytochrome c oxidase subunit Ⅰ mitochondrial genes. Molecular Biology Reports, 36 (5) : 831–839 DOI:10.1007/s11033-008-9252-1 |
| Mahidol C, Na-Nakorn U, Sukmanomon S, et al, 2007. Mitochondrial DNA diversity of the Asian moon scallop, Amusium pleuronectes (Pectinidae), in Thailand. Marine Biotechnology, 9 (3) : 352–359 DOI:10.1007/s10126-006-6137-y |
| Meyer A, 1993.Evolution of mitochondrial DNA in fishes.In:Mochachka P W, Mommsen T P eds.Biochemistry and Molecular Biology of Fishes.Amsterdam:Elsevier, 2:1-38 |
| Navarro J M, Fernández-Reiriz M J, Labarta U, 2006. Differential absorption of biochemical food components by the scallop Argopecten purpuratus exposed to different salinities and food concentrations. New Zealand Journal of Marine and Freshwater Research, 40 (3) : 455–466 DOI:10.1080/00288330.2006.9517435 |
| Navarro J M, Leiva G E, Martinez G, et al, 2000. Interactive effects of diet and temperature on the scope for growth of the scallop Argopecten purpuratus during reproductive conditioning. Journal of Experimental Marine Biology and Ecology, 247 (1) : 67–83 DOI:10.1016/S0022-0981(00)00140-4 |
| Puslednik L, Serb J M, 2008. Molecular phylogenetics of the Pectinidae (Mollusca:Bivalvia) and effect of increased taxon sampling and outgroup selection on tree topology. Molecular Phylogenetics and Evolution, 48 (3) : 1178–1188 DOI:10.1016/j.ympev.2008.05.006 |
| Saavedra C, Peña J B, 2006. Phylogenetics of American scallops (Bivalvia:Pectinidae) based on partial 16S and 12S ribosomal RNA gene sequences. Marine Biology, 150 (1) : 111–119 DOI:10.1007/s00227-006-0335-z |
| Sambrook J, Fritsch E F, Maniatis T A, 1989. Molecular Cloning:A Laboratory Manual. 2nd ed. New York: Cold Spring Harbor Laboratory Press, 1061 |
| Schnelder J A, Foighil DÓ, 1999. Phylogeny of giant clams (Cardiidae:Tridacninae) based on partial mitochondrial 16S rDNA gene sequences. Molecular Phylogenetics and Evolution, 13 (1) : 59–66 DOI:10.1006/mpev.1999.0636 |
| Sunetha K B, Dagle G, Nevdal G, 2000. Analysis of mitochondrial DNA sequences from two Maurolicus taxa:evidence for separate species. Journal of Fish Biology, 57 (6) : 1605–1609 DOI:10.1111/jfb.2000.57.issue-6 |
| Wang C D, Liu B Z, Li J Q, et al, 2011. Introduction of the Peruvian scallop and its hybridization with the bay scallop in China. Aquaculture, 310 (3-4) : 380–387 DOI:10.1016/j.aquaculture.2010.11.014 |
| Winkler F M, Estévez B F, Jollán L B, et al, 2001. Inheritance of the general shell color in the scallop Argopecten purpuratus (Bivalvia:Pectinidae). Journal of Heredity, 92 (6) : 521–525 DOI:10.1093/jhered/92.6.521 |
| Zheng X, Wang R, Xiao S, et al, 2001. Genetic variation in populations of the common Chinese cuttlefish Sepiella maindroni (Mollusca:Cephalopoda) using allozymes and mitochondrial DNA sequence analysis. Journal of Shellfish Research, 20 (3) : 1159–1165 |
 2016, Vol. 47
2016, Vol. 47


