中国海洋湖沼学会主办。
文章信息
- 盖珊珊, 张伟东. 2016.
- GAI Shan-Shan, ZHANG Wei-Dong. 2016.
- 基于第二代测序技术的条斑紫菜(Pyropia yezoensis)贝壳丝状体附生菌群研究
- MICROBIOTA OF SHELL-BORING CONCHOCELIS OF PYROPIA YEZOENSIS DETERMINED BY THE NEXT-GENERATION SEQUENCING
- 海洋与湖沼, 47(5): 990-996
- Oceanologia et Limnologia Sinica, 47(5): 990-996.
- http://dx.doi.org/10.11693/hyhz20160300071
-
文章历史
- 收稿日期:2016-03-29
- 收修改稿日期:2016-06-13
条斑紫菜(Pyropia yezoensis)是自然生长在寒温带海区的大型海藻, 因其具有较高的食用价值和经济效益, 长期以来在包括我国北方沿海在内的东北亚潮间带海域广泛栽培。据相关资料统计, 中、日、韩三国的条斑紫菜年产量已达5万吨(干重)以上, 产值达数亿美元(Blouin et al, 2011)。与许多紫菜种类的典型生活史一样(黒木宗尚, 1953; 曾呈奎等, 1954), 条斑紫菜包含叶状体和丝状体两个世代:叶状体是生产和食用的对象, 由丝状体成熟释放的壳孢子萌发发育而来; 丝状体能够以两种不同的形式存在, 不依赖于基质、自由生长的名为自由丝状体, 溶解钙质、钻入贝壳生长的被称作贝壳丝状体。
相对于自由丝状体而言, 贝壳丝状体更容易实现匀质化的生态因子调控, 并且可以通过洗刷贝壳抑制杂藻生长, 因此在紫菜栽培产业中, 一直采用室内培育贝壳丝状体的方式, 作为壳孢子的来源。然而, 随着栽培产业化规模的扩大和养殖密度的提高, 特别是自然气候条件变化和极端天气频繁出现, 紫菜丝状体病害问题也日益突显, 其中较常见的包括黄斑病、泥红病、白雾病、鲨皮病和绿变病等(贲秀林, 2007)。尽管在紫菜叶状体和丝状体病害的多数研究中仅发现单一的致病菌(马家海, 1996; 闫咏等, 2002; 马家海等, 2007; 严兴洪等, 2008; 王洪斌等, 2011; Guan et al, 2013), 但无法确定所识别的致病菌是否为唯一的病原菌, 并且无法明确同属不同种间是否表现出致病性与益生性的差异。例如, 闫咏等(2002)发现柠檬假交替单胞菌(Pseudoalteromonas citrea)能够引起条斑紫菜绿斑病。王洪斌等(2011)认为河豚毒素假交替单胞菌(P. tetraodonis)是条斑紫菜丝状体黄斑病的病原体。但在其它一些研究中, 杨锐等(2008)观察到假交替单胞菌属在健康紫菜样本中占优势, 而从病烂紫菜中未分离到这类细菌, 提出假交替单胞菌可能与紫菜的健康生长密切相关。在对不同养殖海区红藻表面的假交替单胞菌多样性进行分析时, 武洪庆等(2013)发现假交替单胞菌属普遍存在于大型海藻表面, 是健康条斑紫菜表面优势菌之一, 为藻类的正常生长和发育提供帮助。这些研究结果提示, 物种多样性可能在紫菜病害发生过程中发挥作用。王洪斌等(2011)也认为, 除了河豚毒素假交替单胞菌外, 不能排除黄斑病由其它细菌引起, 或由其它菌群与河豚毒素交替假单胞菌共同作用而致病的可能。目前, 人们对紫菜病害的研究仍局限于分离培养和基于Sanger测序技术的低通量检测阶段, 而忽略了多致病菌因素或病原菌增殖导致的菌群失衡对藻体微环境的影响。
近几年来, 以边合成边测序策略为特点的第二代高通量测序技术逐渐成熟(Ronaghi et al, 2001)。它在数据信息量的获得方面较第一代测序有了巨大突破, 既可用于基因组尺度的序列分析, 又能进行单核苷酸多态性检测、基因频率和转录水平测定等。最为重要的是, 第二代测序技术的建立与推广使得通过大规模、高通量测定DNA序列有效进行微生物的分型鉴定和丰度估计成为可能, 被广泛应用于包括土壤(Roesch et al, 2007)、海洋(Huber et al, 2007)、人体(Turnbaugh et al, 2009)、动植物(McKenna et al, 2008; Cuvelier et al, 2010)等环境样本的微生物组研究中, 并在对微生物致病性疾病的探索中展现出其技术优势和发展潜力。通过这些分析, 可以定义健康机体共生菌群的范围和数量, 由微生物种类和数量上的变化直观地反映健康或疾病状况, 也可以从组学尺度预测微生物毒力因子(如有机酸、裂解酶等)在致病过程中发挥的作用, 以及宿主对微生物群落变化的应答等。
本文拟对发病范围广泛、后果较为严重的贝壳丝状体黄斑病开展研究, 采用第二代测序技术揭示健康贝壳丝状体微生物菌群结构、发病后菌群的变化及其对丝状体生长的潜在作用。
1 材料和方法 1.1 样本采集和处理黄斑病和健康贝壳丝状体样本于2014年8月20日采自江苏省南通市某紫菜育苗场。育苗池海水来自于天然海区, 经过黑暗沉淀和过滤处理后注入育苗池。从不同育苗池中挑选黄斑病症状明显的贝壳4枚, 并从对应池中随机采集4枚健康丝状体贝壳。取样育苗池海水比重(1.019±0.1), 水温(24.7±0.1)°C, pH=(8.2±0.2)。贝壳丝状体于自然光周期(12L︰12D)下培养, 光强约1500lux。
采集贝壳后, 用无菌毛刷轻刷附生有患病或健康丝状体的贝壳表面, 用超纯水配制的人工海水(比重1.020)重悬, 每枚贝壳约收集1mL悬浮物, 分别置于1.5mL的离心管中, 后于-20°C冰箱中冷冻备用。
1.2 DNA制备和16S rDNA扩增用移液器将1mL悬浮物样本吹打重悬, 吸取200μL, 转移至新的1.5mL离心管中。分别向管中加入裂解液, 按照试剂盒(QIAamp DNA Mini Kit, Qiagen)标准流程制备总DNA, 琼脂糖凝胶电泳检测质量。
对各样本16S rRNA的V3高变区进行序列扩增。采用Bartram等(2011)的方法, 在V3区正向引物“CCTACGGGAGGCAGCAG”的5′端加接头、测序引物和4个随机的碱基N(用于测序时初始信号的识别), 相应的在反向引物“ATTACCGCGGCTGCTGG”的5′端加接头、索引序列(index)和测序引物, 得到~80bp的一对引物。引物由生工生物工程(上海)股份有限公司合成。分别以8个样品的DNA为模板, 使用Q5 High-Fidelity 2X Master Mix (NEB)高保真酶扩增V3区, 反应程序与上述文献保持一致。使用该引物进行PCR扩增后, 产物可直接用于测序。
1.3 文库质量控制和测序采用QIAquick PCR Purification Kit (Qiagen)试剂盒回收纯化PCR产物。各样本回收产物经过NanoDrop ND2000 (Thermo)和Fregment Analyzer (AATI)检测浓度和质量, 按照预估浓度等摩尔(0.5nmol/L)混合后用于测序。使用HisSeq2500(Illumina)测序平台, 配对末端PE-150测序。
1.4 数据分析通过识别index对各样本测序数据进行分选, 并滤除低质量的reads(读长, 指高通量测序中一个反应获得的测序序列)。使用FLASH(Magoč et al, 2011)默认参数, 根据重叠区将配对末端reads拼接成较长的序列。去除序列前后端引物和合成引物时添加的4个N碱基, 所得序列进入QIIME分析流程(Caporaso et al, 2010)。根据97%的序列相似性, 采用uclust聚类方法生成可操作分类单元(OTUs), 并基于Greengenes参考序列进行分类注释(DeSantis et al, 2006), 使用MEGAN展示和操作分类数据(Huson et al, 2007)。利用均一化后的OTUs和分类结果, 计算物种多样性、群落结构和样本间聚类关系。
2 结果 2.1 测序数据量和覆盖度受试8个样品经总DNA提取后(图 1), 用于16S rRNA基因扩增和高通量测序。测序共获得1.6Gb数据, 平均每个样本产生1332818±137411条reads。根据重叠区将这些配对末端reads拼接成了超过500万条序列, 各样本所得拼接序列数相近。
 |
| 图 1 受试样品总DNA电泳结果 Fig. 1 DNA electrophoresis of the samples studied 注: M1为λ-HindIII Marker; M2为D2000 Marker; 其余各道从左至右依次为样品he1—he4和ye1—ye4;上样量均为3μL |
拼接序列的长度分布十分集中, 绝大多数序列的长度不超过200bp, 其中长度为171—190bp和191—210bp的序列最多, 分别达到2938907和2202118条, 其余长度区间的序列不超过20000条。
采用uclust聚类(97%相似性), 贝壳丝状体样本16S rRNA序列共识别出7833种OTUs。对测序覆盖度进行统计发现, 各样本OTUs观察数已达到饱和(Good’s coverage > 0.997), 表明该测序深度能够较深入地反映取样环境下的微生物群落组成。
2.2 贝壳丝状体附生微生物组成贝壳丝状体样本16S rRNA V3区与Greengenes参考序列比对注释, 共识别出18个细菌门类。对数据进行均一化处理后发现, 变形细菌门(Proteobacteria)在整个群落中占绝对优势, 比例约为69.4%—83.6%, 其次为拟杆菌门(Bacteroidetes) (8.8%—22.0%)、放线菌门(Actinobacteria) (1.2%— 6.2%)和蓝细菌(Cyanobacteria)(0.6%—4.0%), 其余细菌合计只占总群落的1.6%—4.2%(表 1)。
| he1 | he2 | he3 | he4 | ye1 | ye2 | ye3 | ye4 | |
| 变形菌门Proteobacteria | 83.28 | 81.55 | 80.86 | 82.20 | 83.59 | 82.28 | 69.40 | 76.32 |
| 拟杆菌门Bacteroidetes | 10.45 | 8.76 | 11.43 | 9.96 | 11.10 | 11.41 | 21.96 | 18.49 |
| 放线菌门Actinobacteria | 2.52 | 6.22 | 2.74 | 2.20 | 2.59 | 3.10 | 1.95 | 1.24 |
| 蓝细菌Cyanobacteria | 1.88 | 0.63 | 0.77 | 2.56 | 1.10 | 1.46 | 3.97 | 1.58 |
| 绿弯菌门Chloroflexi | 1.01 | 1.91 | 0.75 | 1.77 | 0.10 | 0.08 | 0.16 | 0.33 |
| 绿菌门Chlorobi | 0.07 | 0.09 | 0.18 | 0.18 | 0.04 | 0.05 | 1.88 | 1.44 |
| 厚壁菌门Firmicutes | 0.14 | 0.19 | 2.77 | 0.23 | 0.03 | 0.04 | 0.04 | 0.03 |
| 酸杆菌门Acidobacteria | 0.06 | 0.17 | 0.08 | 0.13 | 0.98 | 1.26 | 0.14 | 0.09 |
| 浮霉菌门Planctomycetes | 0.42 | 0.32 | 0.32 | 0.64 | 0.23 | 0.19 | 0.19 | 0.27 |
| 疣微菌门Verrucomicrobia | 0.04 | 0.13 | 0.04 | 0.05 | 0.13 | 0.08 | 0.14 | 0.09 |
| 硝化螺旋菌门Nitrospirae | 0.04 | 0.03 | 0.03 | 0.03 | 0.08 | 0.04 | 0.10 | 0.05 |
| 芽单胞菌门Gemmatimonadetes | 0.01 | 0.01 | 0.01 | 0.01 | 0.02 | 0.02 | 0.05 | 0.02 |
| 螺旋体门Spirochaetes | 0.07 | 0.00 | 0.02 | 0.05 | 0.00 | 0.00 | 0.00 | 0.00 |
| 注:显示数值为各受试样本中细菌门所占比例(%); 仅示在所有受试样本中总丰度≥1‰的门类 | ||||||||
变形细菌中绝大多数属于α-变形菌纲(Alphaproteobacteria), 此外β-变形菌纲(Betaproteobacteria)、γ-变形菌纲(Gammaproteobacteria)相对丰度较高, 仅有少量的δ-变形菌纲(Deltaproteobacteria)、ε-变形菌纲(Epsilonproteobacteria)和ζ-变形菌纲(Zetaproteobacteria)细菌。黄杆菌纲(Flavobacteria)、酸微菌纲(Acidimicrobiia)、厌氧绳菌纲(Anaerolineae)和放线菌纲(Actinobacteria)等也是贝壳丝状体微生物群落中丰度较高的类群。
从贝壳丝状体样本共识别出396个细菌属, 其中140个属的16S rRNA序列超过100条, 94个属超过500条。放线菌(Actinobacteria)、Thalassobacter、Marivita、Loktanella、德沃斯氏菌(Devosia)、Maribacter、弧菌(Vibrio)、氢噬胞菌(Hydrogenophaga)、Phaeobacter和Arenimonas等是其中丰度最高的细菌属。
2.3 贝壳丝状体菌群的alpha多样性为研究丝状体患病前后微生物多样性变化, 我们统计了Chao1、ACE、Shannon和Simpson四种指数(表 2)。所有数据均显示, 相对于健康对照组, 黄斑病贝壳丝状体的微生物多样性显著增加(t-test, P < 0.05), 提示发生病害的丝状体菌群结构出现了较大程度的改变。
| Chao1 | ACE | Shannon | Simpson | |
| he1 | 3643.222 | 3585.123 | 7.165 | 0.977 |
| he2 | 3816.084 | 3848.863 | 7.113 | 0.972 |
| he3 | 4024.929 | 4035.836 | 6.488 | 0.935 |
| he4 | 4006.383 | 4010.577 | 6.958 | 0.956 |
| ye1 | 4150.868 | 4157.153 | 7.336 | 0.986 |
| ye2 | 4693.210 | 4723.940 | 7.236 | 0.984 |
| ye3 | 5676.537 | 5621.039 | 7.640 | 0.986 |
| ye4 | 5556.730 | 5560.481 | 7.441 | 0.982 |
为进一步揭示健康和患病丝状体菌群之间的差异, 我们基于OTUs组成对样本间菌群差异和距离进行了加权的UniFrac主坐标分析(PCoA)。健康和黄斑病丝状体可以被区分为不同的组(图 2a), 且健康组样本间的距离明显小于黄斑病组。UPGMA树在各样本间显示出同样的聚类关系(图 2b)。该结果说明健康贝壳丝状体保持着相似的微生物群落结构, 而患病后的丝状体则发生了不同的变化。造成这种情况的原因可能是原本处于高度相似状态、较为稳定的健康菌群平衡受到破坏, 菌群紊乱使优势细菌的种类和丰度在样本间变化幅度较大, 从而导致疾病组样品间β多样性较高。
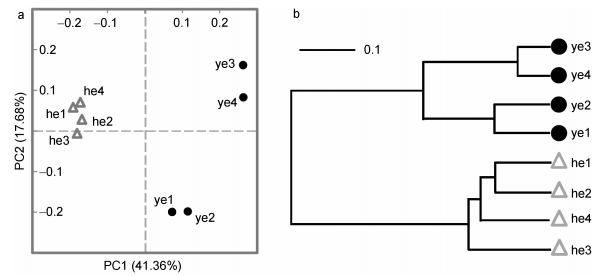 |
| 图 2 贝壳丝状体样本间的菌群聚类关系 Fig. 2 Sample clustering based on bacterial communities 注: a.根据OTUs组成对样本间菌群差异和距离进行加权的UniFrac主坐标分析[x轴和y轴分别表示主成分1(PC1)和主成分2(PC2)]; b. UPGMA树分析样品间聚类关系(比例尺表示分支距离, he1—he4代表健康样品, ye1—ye4代表黄斑病样品) |
为识别导致健康和黄斑病贝壳丝状体菌群差异的微生物种类, 我们比较了相对丰度较高(16S rRNA序列数 > 500)的93个细菌属在不同样本组间的分布。健康和黄斑病样本组间差异显著(t-test, P < 0.05)的细菌属有39个(图 3), 其中29个在健康样本中的相对丰度明显高于患病组样本, 包括放线菌、噬甲基菌(Methylophaga)、假单胞菌(Pseudomonas)、假苍白杆菌(Pseudochrobactrum)、Roseicyclus、Jannaschia和Methylotenera等。而Maribacter、Thalassococcus、Glaciecola、不动杆菌(Acinetobacter)和硝化螺旋菌(Nitrospira)等10个属在患病组样本中相对丰度显著高于健康组。
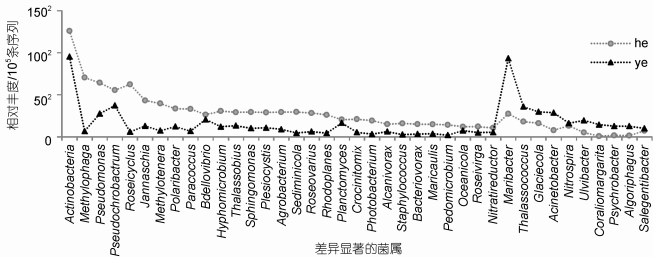 |
| 图 3 健康和黄斑病贝壳丝状体样本间的微生物差异 Fig. 3 Bacterial difference between healthy and yellow-spot diseased conchocelis 注: he代表健康样本, ye代表黄斑病贝壳丝状体样品; x轴仅显示组间差异显著(t-test, P < 0.05)的39个细菌属; y轴数值经过归一化处理, 表示每100000条序列识别出该物种的相对丰度 |
条斑紫菜养殖过程中的许多病害是由微生物导致的, 其特点是发病快、传染力强、危害严重, 所以有研究者一直在尝试分离培养紫菜附生菌(Tsukidate et al, 1983; 陈国耀等, 1999; 杨锐等, 2008; 武洪庆, 2012), 以期了解共生菌与紫菜健康的关系并识别出可能的致病菌。但由于受到技术发展水平的限制, 只有少数高丰度的海洋微生物种类能够被检测发现, 人们对紫菜(尤其是贝壳丝状体)附生菌群的认识还十分有限。
在对条斑紫菜附生细菌的报道中, 假交替单胞菌属细菌仅在健康藻体中分离得到, 且占有优势, 而在患病紫菜中未有发现, 因此认为该属与紫菜健康状态密切相关(杨锐等, 2008)。但武洪庆等(2013)对不同养殖海区红藻表面假交替单胞菌多样性进行分析, 从江苏如东和射阳的条斑紫菜叶状体上没有检测到该细菌存在, 因而对其能否体现健康与病烂紫菜附生细菌组成的主要差别产生了疑问。在本研究中, 有包括放线菌在内的29个属的细菌在健康和黄斑病丝状体样本组间差异显著, 且与健康状态相关联, 但不包含假交替单胞菌属, 所以该属不能用于指示丝状体健康状态。不同研究对于假交替单胞菌认识的差异可能由于藻体世代(叶状体或丝状体)交替、海区环境等的不同, 也可能与采用不同的微生物检测方法有关。
在仅有的条斑紫菜贝壳丝状体黄斑病研究中, 王洪斌等(2011)从江苏连云港采集患病样本, 利用特定培养基筛选其中的优势细菌, 并通过形态学、生理生化和分子生物学方法鉴定为河豚毒素交替假单胞菌, 但存在黄斑病由其它细菌引起, 或由其它菌群与河豚毒素交替假单胞菌共同作用而致病的可能。尽管许多真菌种类也可能是致病的重要因素(马家海, 1996; 马家海等, 2007; Guan et al, 2013), 但从黄斑病和健康贝壳丝状体菌群比较来看, 细菌群落结构差异显著, 说明可能有多种细菌参与了致病过程, 并在发病前后影响丝状体的生长状态。例如, 健康丝状体中相对丰度最高的放线菌属细菌, 因具有广泛的抗生素生产能力(Mahajan et al, 2012), 可能在丝状体生长中起保护和屏障作用。患病丝状体中相对变化显著且丰度最高的是黄杆菌科(Flavobacteriaceae)细菌Maribacter。这类细菌以产黄色素为普遍特征(Nedashkovskaya et al, 2004), 较广泛地分布于自然海区和多种海藻表面(武洪庆, 2012), 该菌为条件致病菌, 在一般情况下不引起感染, 仅在特定环境因子下引起感染和扩散, 可能具有较强的致病力。例如, 有研究者发现Maribacter的有些种类可能与其它细菌一起造成甲壳纲动物壳体损伤疾病(Chistoserdov et al, 2012; Quinn et al, 2012)。尽管Maribacter的一些菌株可能对青霉素、新生霉素和林可霉素等敏感(Hu et al, 2015), 但从食品安全角度出发, 应尽量避免在病害预防和控制过程中使用此类抗生素药物。
虽然没有对这些细菌开展分离和回复感染实验(用于验证柯赫氏法则), 但本研究筛选出了一些与病害关联的微生物标记, 缩小了验证范围, 为实验对象的选择提供了参考。另外, 光照、温度和水质条件等因素对紫菜的生理和生长状态十分重要(王淑刚等, 2013; 吴海一等, 2015), 因此在后续工作中, 应对贝壳丝状体育苗池水的环境生态因子和水体微生物群落变化进行深入研究, 特别是时间梯度的连续性分析, 以期在相关病害的检测和防治过程中发挥预警的作用。
4 结论本研究利用第二代测序技术, 获得了第一个高通量的条斑紫菜贝壳丝状体细菌群落数据集, 不仅对高、低丰度细菌门类加以量化, 还从属水平识别出近400个细菌分类, 扩大了贝壳丝状体核心细菌群落的范围, 并对健康和黄斑病状态下的贝壳丝状体菌群的进行了比较分析。结果发现, 健康和黄斑病样本组间差异显著的细菌属有39个, 其中如放线菌、噬甲基菌和假单胞菌等29个属在健康样本中的相对丰度显著高于病患组, 与丝状体健康状态密切相关; 而Maribacter、Glaciecola和Acinetobacter等10个细菌属在黄斑病样本中相对丰度显著较高, 可能与丝状体患病有关。本研究从菌群生态角度为丝状体病害的检测和预防提供了有价值的信息。
| 马家海, 1996. 条斑紫菜赤腐病的初步研究. 上海水产大学学报, 5 (1) : 1–7 |
| 马家海, 林秋生, 闵建, 等, 2007. 条斑紫菜拟油壶菌病的初步研究. 水产学报, 31 (6) : 860–864 |
| 王洪斌, 李信书, 夏亚明, 等, 2011. 条斑紫菜丝状体黄斑病病原体分离鉴定及生物学特性研究. 海洋环境科学, 30 (3) : 361–364 |
| 王淑刚, 杨锐, 周新倩, 等, 2013. 高温胁迫下坛紫菜(Pyropia haitanensis)对无机碳的利用. 海洋与湖沼, 44 (5) : 1378–1385 |
| 闫咏, 马家海, 许璞, 等, 2002. 1株引起条斑紫菜绿斑病的柠檬假交替单胞菌. 中国水产科学, 9 (4) : 353–358 |
| 严兴洪, 黄林彬, 周晓, 等, 2008. 坛紫菜叶状体的细菌性红烂病研究. 中国水产科学, 15 (2) : 313–322 |
| 杨锐, 方文雅, 单媛媛, 等, 2008. 条斑紫菜外生细菌的遗传多样性. 海洋学报, 30 (4) : 161–168 |
| 吴海一, 丁刚, 徐智广, 2015. 不同氮浓度下盐胁迫对坛紫菜(Pyropia haitanensis)生长和光合作用的影响. 海洋与湖沼, 46 (5) : 1210–1217 |
| 陈国耀, 沈怀舜, 朱庙先, 等, 1999. 条斑紫草叶状体附生菌及病原菌的初步调查. 水产养殖, (1) : 17–19 |
| 武洪庆, 2012. 青岛:中国科学院研究生院(海洋研究所)博士学位论文. |
| 武洪庆, 刘敏, 肖天, 2013. 不同养殖区红藻表面假交替单胞菌多样性分析. 海洋科学, 37 (10) : 17–23 |
| 贲秀林, 2007. 条斑紫菜丝状体培育的病害及防治. 水产养殖, 28 (1) : 43–44 |
| 黒木宗尚, 1953. アマノリ類の生活史の研究(1). 東北海区水産研究所研究報告, (2) : 67–103 |
| 曾呈奎, 张德瑞, 1954. 紫菜的研究I. 甘紫菜的生活史.植物学报, 3 (3) : 287–302 |
| Bartram A K, Lynch M D J, Stearns J C, et al, 2011. Generation of multimillion-sequence 16S rRNA gene libraries from complex microbial communities by assembling paired-end Illumina reads. Applied and Environmental Microbiology, 77 (11) : 3846–3852 DOI:10.1128/AEM.02772-10 |
| Blouin N A, Brodie J A, Grossman A C, et al, 2011. Porphyra: a marine crop shaped by stress. Trends in Plant Science, 16 (1) : 29–37 DOI:10.1016/j.tplants.2010.10.004 |
| Caporaso J G, Kuczynski J, Stombaugh J, et al, 2010. QⅡME allows analysis of high-throughput community sequencing data. Nature Methods, 7 (5) : 335–336 DOI:10.1038/nmeth.f.303 |
| Chistoserdov A Y, Quinn R A, Gubbala S L, et al, 2012. Bacterial communities associated with lesions of shell disease in the American lobster, Homarus americanus Milne-Edwards. Journal of Shellfish Research, 31 (2) : 449–462 DOI:10.2983/035.031.0205 |
| Cuvelier M L, Allen A E, Monier A, et al, 2010. Targeted metagenomics and ecology of globally important uncultured eukaryotic phytoplankton. Proceedings of the National Academy of Sciences of the United States of America, 107 (33) : 14679–14684 DOI:10.1073/pnas.1001665107 |
| DeSantis T Z, Hugenholtz P, Larsen N, et al, 2006. Greengenes, a chimera-checked 16S rRNA gene database and workbench compatible with ARB. Applied and Environmental Microbiology, 72 (7) : 5069–5072 DOI:10.1128/AEM.03006-05 |
| Guan X Y, Li J B, Zhang Z, et al, 2013. Characterizing the microbial culprit of white spot disease of the conchocelis stage of Porphyra yezoensis (Bangiales, Rhodophyta). Journal of Applied Phycology, 25 (5) : 1341–1348 DOI:10.1007/s10811-013-9976-8 |
| Hu J, Yang Q Q, Ren Y, et al, 2015. Maribacter thermophilus sp. nov., isolated from an algal bloom in an intertidal zone, and emended description of the genus Maribacter. International Journal of Systematic and Evolutionary Microbiology, 65 (1) : 36–41 |
| Huber J A, Welch D B M, Morrison H G, et al, 2007. Microbial population structures in the deep marine biosphere. Science, 318 (5847) : 97–100 DOI:10.1126/science.1146689 |
| Huson D H, Auch A F, Qi J, et al, 2007. MEGAN analysis of metagenomic data. Genome Research, 17 (3) : 377–386 DOI:10.1101/gr.5969107 |
| Magoč T, Salzberg S L, 2011. FLASH: fast length adjustment of short reads to improve genome assemblies. Bioinformatics, 27 (21) : 2957–2963 DOI:10.1093/bioinformatics/btr507 |
| Mahajan G B, Balachandran L, 2012. Antibacterial agents from actinomycetes-a review. Frontiers in Bioscience, 4 : 240–253 |
| McKenna P, Hoffmann C, Minkah N, et al, 2008. The macaque gut microbiome in health, lentiviral infection, and chronic enterocolitis. PLoS Pathogens, 4 (2) : e20 DOI:10.1371/journal.ppat.0040020 |
| Nedashkovskaya O I, Kim S B, Han S K, et al, 2004. Maribacter gen. nov., a new member of the family Flavobacteriaceae, isolated from marine habitats, containing the species Maribacter sedimenticola sp. nov., Maribacter aquivivus sp. nov., Maribacter orientalis sp. nov. and Maribacter ulvicola sp. nov. International Journal of Systematic and Evolutionary Microbiology, 54 (4) : 1017–1023 DOI:10.1099/ijs.0.02849-0 |
| Quinn R A, Metzler A, Smolowitz R M, et al, 2012. Exposures of Homarus americanus shell to three bacteria isolated from naturally occurring epizootic shell disease lesions. Journal of Shellfish Research, 31 (2) : 485–493 DOI:10.2983/035.031.0208 |
| Roesch L F W, Fulthorpe R R, Riva A, et al, 2007. Pyrosequencing enumerates and contrasts soil microbial diversity. The ISME Journal, 1 (4) : 283–290 |
| Ronaghi M, 2001. Pyrosequencing sheds light on DNA sequencing. Genome Research, 11 (1) : 3–11 DOI:10.1101/gr.11.1.3 |
| Tsukidate J, 1983. On the symbiotic relationship between Porphyra species and attached bacteria, and a bacterial pathogen in white rot. Bulletin of the Nansei Regional Fisheries Research Laboratory, 15 : 29–96 |
| Turnbaugh P J, Hamady M, Yatsunenko T, et al, 2009. A core gut microbiome in obese and lean twins. Nature, 457 (7228) : 480–484 DOI:10.1038/nature07540 |
 2016, Vol. 47
2016, Vol. 47


