中国海洋湖沼学会主办。
文章信息
- 梁日深, 吴丽钰, 苏国茂, 程硕, 周萌, 吴灶和. 2016.
- LIANG Ri-Shen, WU Li-Yu, SU Guo-Mao, CHENG Shuo, ZHOU Meng, WU Zao-He. 2016.
- 印度-太平洋石鲈亚科鱼类DNA条形码及分子系统进化研究
- DNA BARCODING FOR WORLDWIDE HAEMULINAE IN CLASSIFICATION AND PHYLOGENY
- 海洋与湖沼, 47(1): 99-107
- Oceanologia et Limnologia Sinica, 47(1): 99-107.
- http://dx.doi.org/10.11693/hyhz20150200059
-
文章历史
- 收稿日期: 2015-02-18
- 收修改稿日期: 2015-07-21
石鲈亚科(Haemulinae)隶属鲈形目Perciformes、仿石鲈科Haemulidae,主要分布于印度-太平洋亚热带及热带海区,少数分布于东大西洋,是重要的海洋经济食用及观赏性鱼类。世界上石鲈亚科约15属90余种,中国仅记录石鲈属1属3—6种(Mckay,1984; 成庆泰等,1987; 沈世杰,1993; 黄宗国,1994; 孟庆闻等,1995; Carpenter et al,2001),主要分布在南海。
形态上,石鲈亚科鱼类近缘物种异常相似,鉴定存在难度,故许多物种分类命名一直悬而未决,大多数种类但仅凭外部的可辨析成分难以有效区分,需要辅以内部解剖分析才能最终确定,这给石鲈亚科物种快速鉴定造成了很大的不便,同时还会破坏标本的完整性。目前形态分类资料上,石鲈亚科不少物种出现误鉴,分类命名相互混淆,许多种类的物种有效性与同种异名争议长期存在分歧。如国内绝大部分资料利用P. hasta表示断斑石鲈,P. kaakan表示点石鲈(成庆泰等,1987; 孟庆闻等,1995),而国外资料却认为P. hasta是P. kaakan的同种异名,P. kaakan是断斑石鲈的有效种名,点石鲈学名为P. argenteus (Mckay,1984; Carpenter et al,2001; Froese et al,2013; FAO-FIES,2014),但P. argenteus在其它资料中却又表示另外一个种类——银石鲈(沈世杰,1993); 又如: 部分资料认为细纹石鲈(Pomadasysperotaei)与佩氏石鲈(Pomadasys peroteti)是独立的物种(Mckay,1984; Randall,2005),另有资料却认为它们是同种异名(Froese et al,2013; FAO-FIES,2014)而有关石鲈亚科分子分类学方面的研究报道较少,大部分研究是在石鲈科鱼类系统发育分析中涉及相关石鲈亚科的鱼类(朱世华等,2006; 任岗等,2007; Sanciangco et al,2011; Tavera et al,2012),而利用DNA条形码技术(DNA barcode)对石鲈亚科鱼类进行物种聚类及分子鉴定的研究至今仍未见报道。
DNA条形码是通过一个标准目的基因的DNA序列分析进行物种鉴定的技术(Hebert,2003a,b)。在鱼类上,该标准基因是线粒体细胞色素C氧化酶亚基I(COⅠ)基因5端一段长度为648bp的片段,它能够在分子水平上成功区分物种,为生物的分类提供一种快速简便、可信可靠的分类方法(Hubert et al,2008)。目前,DNA条形码技术在鱼类分子分类与鉴定上已得到了广泛的应用,弥补传统形态分类鉴定的局限性,有效解决鱼类形态分类上许多争议问题(Schlei et al,2008; Pereira et al,2013; 毛云涛等,2014; 宋超等,2014)。如Pereira等(2013)利用COⅠ条形码序列对新热带区的254种淡水鱼类进行分子鉴定,其中252种鱼类能被清晰区分,并确定23个隐存种; 宋超等(2014)基因COⅠ序列对长江口舌鳎科鱼类的遗传差异进行分析,有效澄清了短吻三线舌鳎与紫斑舌鳎,长吻红舌鳎与短吻红舌鳎等同种异名的争议。
本研究在形态学分类鉴定的基础上,对29种石鲈亚科线粒体COⅠ基因进行序分析,构建石鲈亚科鱼类分子系统进化关系,探讨DNA条形码在石鲈亚科鱼类物种分子鉴定中的适用性及完善其检索系统的可行性,为石鲈亚科物种有效鉴定,解决物种分类问题,保护种质资源提供分子水平的数据支持。
1 材料与方法 1.1 材料来源实验样品采集于世界各地,其中国内石鲈属样品主要在南海采集,世界其它地区的样品由国外多个科研单位提供: 南非水生物多样性研究院(South African Institute for Aquatic Biodiversity,SAIAB),美国堪萨斯大学生物多样性研究中心(Biodiversity Institute,University of Kansas); 南太平洋岛国新喀里多尼亚努美阿泻湖水族馆(Noumea Aquarium des Lagons)。最后共采集石鲈亚科样品材料29种91个个体,归类于7个属。标本种类与来源见图 1及表 1。同时GenBank下载蓝颊石鲈 Pomadasys argyreus、切齿石鲈Pomadasys incisus、巴拿石鲈Pomadasys panamensis、红锯鳃石鲈Orthopristis rubber的COⅠ序列统一进行分析。
 |
| 图 1 实验样品采集地理分布图 Fig. 1 Geographic distribution ofexperimental samples |
| 属 | 种 | 标本来源 | 数量 |
| 石鲈属 | |||
| Pomadasys | 大斑石鲈Pomadasys maculatus | 广东,阳江,深圳 | 5 |
| 断斑石鲈Pomadasys hasta | 广东,深圳 | 4 | |
| 银石鲈Pomadasys argenteus | 新喀里多尼亚,努美阿 | 4 | |
| 侧扁石鲈Pomadasys corvinaeformis | 伯利兹,加勒比海 | 3 | |
| 大棘石鲈Pomadasys macracanthus | 萨尔瓦多,拉乌尼翁 | 2 | |
| 纵带石鲈Pomadasys striatum | 南非,夸祖鲁纳塔尔 | 4 | |
| 赤笔石鲈Pomadasys furcatus | 莫桑比克,马普托 | 4 | |
| 佩氏石鲈Pomadasys peroteti | 安哥拉,Barra do Kwanza Lodge | 4 | |
| 康氏石鲈Pomadasys commersonnii | 南非,东开普省 | 3 | |
| 黄鳍石鲈Pomadasys olivaceus | 南非,东开普省 | 2 | |
| 蓝颊石鲈 Pomadasys argyreus | HQ676793 | 1 | |
| 切齿石鲈Pomadasys incisus | KJ768282~KJ768284 | 3 | |
| 巴拿石鲈Pomadasys panamensis | HQ676799 | 1 | |
| 仿石鲈属 | |||
| Haemulon | 黄仿石鲈Haemulon flavolineatum | 百慕大,圣乔治斯岛 | 4 |
| 大口仿石鲈Haemulon macrostomum | 伯利兹,Carrie Bow Cay | 4 | |
| 蓝仿石鲈Haemulon sciurus | 伯利兹,Carrie Bow Cay | 3 | |
| 普氏仿石鲈Haemulon plumierii | 伯利兹,Carrie Bow Cay | 2 | |
| 银仿石鲈Haemulon chrysargyreum | 伯利兹,Carrie Bow Cay | 3 | |
| 异孔石鲈 | |||
| Anisotremus | 黑异孔石鲈Anisotremus surinamensis | 波多黎各,Playa Sardinera | 4 |
| 枝异孔石鲈Anisotremus virginicus | 伯利兹,Carrie Bow Cay | 5 | |
| 条纹异孔石鲈Anisotremus taeniatus | 美国,南加利福利亚州 | 3 | |
| 达氏异孔石鲈Anisotremus davidsonii | 美国,南加利福利亚州 | 3 | |
| 小仿石鲈属 | |||
| Haemulopsis | 白色小仿石鲈Haemulopsis leuciscus | 萨尔瓦多,拉乌尼翁 | 3 |
| 腋斑小仿石鲈Haemulopsis axillaris | 墨西哥,马萨特兰 | 2 | |
| 八带石鲈属 | |||
| Conodon | 八带石鲈Conodon nobilis | 美国,得克萨斯州 | 2 |
| 锯齿八带石鲈Conodon serrifer | 美国,南加利福利亚州 | 3 | |
| 锯鳃石鲈属 | |||
| Orthopristis | 金鳍锯鳃石鲈Orthopristis chrysoptera | 美国,南卡罗来纳州 | 3 |
| 红锯鳃石鲈Orthopristis rubber | GU702505~ GU702509 | 4 | |
| 异石鲈属 | |||
| Xenistius | 北美异石鲈Xenistius californiensis | 美国,南加利福利亚州 | 3 |
| 外类群 | |||
| 胡椒鲷亚科 | |||
| 胡椒鲷属 | 斜纹胡椒鲷 Plectorhinchus lineatus | 海南,三亚 | 3 |
| Plectorhinchus | 四带胡椒鲷Plectorhinchus diagrammus | 海南,三亚 | 3 |
| 少棘胡椒鲷属Diagramma | 少棘胡椒鲷Diagramma pictum | 广东,深圳 | 2 |
| 矶鲈属Parapristipoma | 三线矶鲈Parapristipoma trilineatum | 广东,阳江 | 3 |
基因组DNA提取采用常规提取方法,蛋白酶K 55°C消化3—4h,酚-氯仿抽提,提取的DNA于50μL灭菌水中溶解,琼脂糖凝胶电泳检测提取质量,-20°C保存并用于下游实验。
1.3 PCR扩增及序列测定PCR扩增的目的片段为COⅠ 5’端长度为651bp的序列。扩增引物为DNA 条形码通用引物,由上海英骏公司合成,序列为: COⅠ-F1:5- TCA ACY AAT CAY AAA GAT ATYGGC AC -3,COⅠ-R1:5- ACT TCY GGG TGR CCR AAR AATCA -3(Ward et al,2005)。PCR反应体系为50μL,其中10×PCR缓冲液(含Mg2+)5μL,dNTPs(各2.5mmol/L)2μL,上游与下游引物各1μL,2μLDNA聚合酶(1U/μL),ddH2O 37μL。PCR反应扩增程序为: 94°C预变性5min,94°C变性30s,54°C退火30s,72°C延伸1min,33个循环,最后72°C延伸10min。PCR产物经1.5%琼脂糖凝胶电泳分离检测,特异片段纯化后送到上海英骏生物技术有限公司进行双向测序。
1.4 数据分析所获序列利用BioEdit生物软件分析测序结果,并于NCBI上利用Blast进行序列相似性检索,验证序列的准确性。与GenBank下载的序列一起利用Clustal W(Thompson et al,1994)进行序列比对,同时辅以手工校正,仅保留一致性序列区域。利用 MEGA5.0(Tamura etal,2011)计算序列的碱基组成、变异位点、简约信息位点数、转换颠换频率,基于Kimura-2-parameter模型计算各物种种内、种间的遗传距离。分子系统进化树的构建利用最大似然法(Maximum likelihood,ML)和最大简约法(Maximum parsimony,MP)。最大似然树在PHYML v2.4.4软件中完成,最大简约法在PAUP Version 4.0b 10(Swofford,2003)软件包中完成,使用启发式搜寻,树二等分再连接tree-bisection-reconnection(TBR)参数构树,所有数据未加权。系统进化树各分支的支持率采用1000次重复抽样分析(Bootstrapanalysis)进行检验。
2 结果与分析 2.1 序列分析试验测得29种石鲈亚科COⅠ序列为651bp片段序列,编码272个氨基酸,序列中无碱基的插入、缺失。与GenBank下载的序列一起分析计算可知: 除去外类群,序列中A、T、G、C碱基平均含量分别为21.9%、27.5%、19.7%、30.9%,其中A+T含量(49.4%)略低于G+C含量(50.6%)。在651bp COⅠ基因编码序列中,密码子第1位四种碱基含量差异不大,相对最高的是G-1(31.7%); 密码子第2位中,T-2含量(41.0%)最高,并明显高于其它三种碱基; 密码子第3位中C-3含量最高(36.9%),G-3含量最低(12.2%),表现出明显的反G偏倚(图 2)。在G+C含量中,密码子第1位点G+C含量(59.0%)高于第2和第3位(43.8%,49.1%)。此外,在长度为651bp序列中,除去外类群,保守位点417个(64.1%),变异位点234(35.9%),简约性信息位点224(34.4%)。石鲈属各物种两两之间变异位点数为14—130,其中黄鳍石鲈与切齿石鲈之间变异位点数最少,而黄鳍石鲈与其它石鲈属种类变异位点数均较高(106—130,大多超过113),甚至超过了其与石鲈亚科其它属鱼类的变异位点数,如与仿石鲈属(104—118),异孔石鲈属(107—111),八带石鲈属(107—111)等。此外,佩氏石鲈(109—127),切齿石鲈(100—127)也存在属内种间变异位点数超过属间变异位点数的现象。
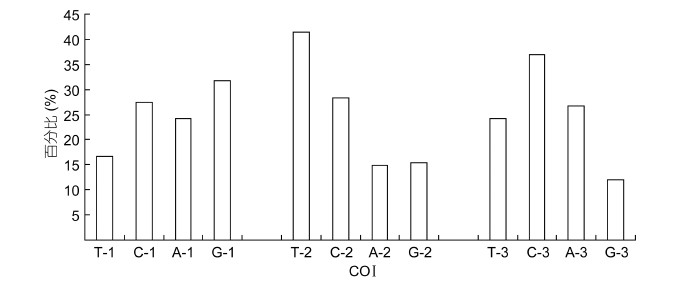 |
| 图 2 COⅠ基因第一、二、三位密码子碱基组成 Fig. 2 The base composition of the 1st,2nd,and 3rd codon of COⅠ gene |
基于Kimura-2-parameter模型计算出COⅠ序列中转换与颠换之比值为2.50,转换大于颠换,序列位点没有突变饱和,可直接用于分子系统进化树的构建分析。在所有位点中,不变位点有549个,转换位点68个,颠换位点34个。其中不变位点数在密码子第2位中最多,为217个; 而转换位点和颠换位点均属密码子第3位最多,分别为63个和34个(表 2)。碱基替换中转换形式以T-C转换为主,颠换形式A-T、A-C的颠换多于G-T、G-C(表 2)。
| 密码子 位点 | 不变位点ii(个) | 转换位点si(个) | 颠换位点sv(个) | R=si/sv |
| 所有位点 | 549 | 68 | 34 | 1.97 |
| 1st位点 | 214 | 4 | 0 | 15.76 |
| 2nd位点 | 217 | 0 | 0 | 3.86 |
| 3rd位点 | 119 | 63 | 34 | 1.86 |
基于Kimura 2-parameter模型计算29种石鲈亚科鱼类COⅠ基因序列种内与种间遗传距离如表 3所示,在所有石鲈亚科鱼类中,种内遗传距离为0.000— 0.009,平均值为0.004,显著低于Hebert等(2003a,b)所提出的0.020(2%)作为物种鉴定最小种间遗传距离。29种鱼类种间遗传距离范围在0.021— 0.240之间,平均值为0.184,98%以上遗传距离大于0.100,90%以上大于0.150,其中最大种间遗传距离为腋斑小仿石鲈与普氏仿石鲈(0.240); 最小种间遗传距离为黄鳍石鲈与切齿石鲈(0.021),均大于Hebert等(2003a,b)设定的2%(0.020)的遗传差异。石鲈属属内种间遗传距离为0.086—0.238,平均遗传距离0.189,石鲈属与石鲈亚科其它属的遗传距离范围是: 仿石鲈属(0.156—0.229),异孔石鲈属(0.151—0.212),八带石鲈(0.140—0.205),小仿石鲈属(0.142—0.234),锯鳃石鲈属(0.156—0.219),比较分析可知,石鲈属部分种类属内种间遗传距离甚至超出了其与石鲈亚科其它属之间的属间遗传距离: 如黄鳍石鲈(0.186— 0.237),佩氏石鲈(0.194—0.231),侧扁石鲈(0.184— 0.229)等,其与石鲈属其它种类遗传距离数据大多在0.200以上,显示石鲈属内部部分种类在分子遗传水平上已呈现出较大的分化。
 |
基于所得的COⅠ序列,以胡椒鲷亚科的种类作为外类群,采用ML法和MP法,构建分子系统进化树,两种方法构建的进化树拓扑结构大致相同(图 3)。树上各节点上的数值分别为Bootstrap统计分析后对该分支ML/MP的支持率。进化树上,石鲈亚科同一物种不同个体均能聚成一支,形成物种内的单系。不同物种之间,石鲈亚科7个属的种类大致形成8个分支,其中石鲈属鱼类未能形成一个严格的单系,在进化树上分成了5个小分支,除了分支IV由6种石鲈聚成的石鲈属一个主要类群外,其它石鲈属种类都分散在进化树其它位置,与石鲈亚科其它属的种类相聚。其中,侧扁石鲈与小仿石鲈属种类聚在一起,巴拿石鲈、纵带石鲈与锯鳃石鲈聚在一起,不过支持率相对较低,大棘石鲈与红海石鲈单独形成一支,佩氏石鲈,黄鳍石鲈,切齿石鲈聚在一起,单独形成一支,位于整个石鲈亚科类群进化树的基部。
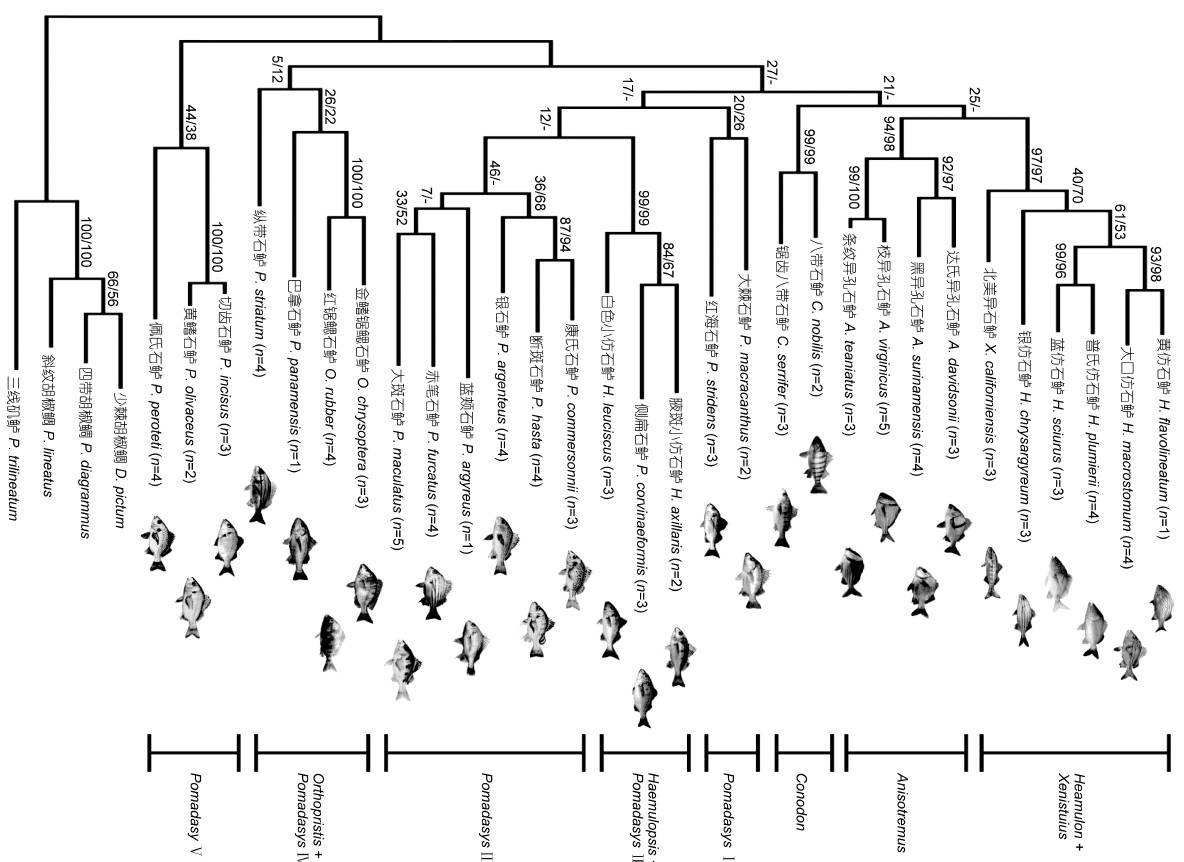 |
| 图 3 基于COⅠ序列的最大似然法与最大简约法构建的29种石鲈亚科鱼类分子系统进化树 Fig. 3 Molecular phylogenetic trees of 29Haemulinae fish in COⅠsequence constructed by maximum likelihood and maximum parsimonymethod |
自Hebert等(2003a,2003b)提出以COⅠ基因作为DNA条形码以来,该基因在不同生物类群中应用的有效性得到了越来越多的验证(Johnson et al,2008; Schlei et al,2008; Keskin et al,2013; Keskin et al,2013; 毛云涛等,2014)。Hebert等(2003a)对11个门13320个物种的COⅠ基因序列进行分析,发现COⅠ序列间的差异能够很好的区分所研究的物种,物种内的COⅠ遗传距离很少有大于2%,大部分小于1%。Hebert等(2003a,b)提出,物种种间遗传距离大于种内遗传距离是COⅠ序列能够进行物种有效鉴别的关键条件,并且两者距离差异大约为10倍。本研究分析了29种石鲈亚科鱼类91个个体的COⅠ序列,其种内遗传距离平均值为0.004,种间遗传距离平均值为0.184,种间遗传距离是种内的46倍。此外,石鲈亚科鱼类种内遗传距离范围为0.000—0.009,显著低于Hebert等(2003a)所推荐的0.200(2%)作为物种鉴定最小种间遗传距离; 而种间遗传距离范围为0.021— 0.240,均大于Hebert设定的2%的遗传差异。可知,在石鲈亚科鱼类物种鉴定研究中,COⅠ基因序列可对石鲈亚科鱼类进行有效的物种DNA鉴定。 3.2 基于DNA条形码序列石鲈亚科鱼类分子系统进化关系分析
DNA条形码基因COⅠ序列分析主要用于生物分类系统的分子鉴定,而许多研究也表明COⅠ在系统进化分析上也能提供丰富的进化信息。本研究基于COⅠ序列构建的系统进化树,石鲈亚科同种鱼类不同个体均可聚为同一分支,形成严格的单系。在属内种间分类关系中,大部分同属的物种均能聚在一起,形成属级阶元的分支,但也有个别类群无法形成单系,如石鲈属,所研究的石鲈属鱼类在进化树上形成多个分散的独立分支,无法构成单系。但该结果与近几年的研究报道一致,如Sanciangco等(2011)与Tavera等(2012)基于多个分子标记对东太平洋及周边水域的石鲈科鱼类的分类研究就揭示石鲈属是非单系群。在Sanciangco等(2011)研究中,10种石鲈属鱼类主要分成三个独立的分支(I: 东大西洋,II: 印度-西太平洋,III: 美洲海区),不同分支上的石鲈属鱼类与所在的地理分布存在一定的联系。Tavera等(2012)认为栖息地与地理隔离协同影响着石鲈属鱼类的多样性,其研究中分布美洲石鲈属鱼类与美洲石鲈亚科的物种聚在一起,相同的栖息地与分布环境影响着石鲈属鱼类的遗传分化。本研究中14种石鲈属鱼类共形成5个分支,其中类群V的三种石鲈(佩氏石鲈+黄鳍石鲈+切齿石鲈)分类地位较低,位于石鲈亚科类群基部,除了佩氏石鲈外,另外两种石鲈均分布在东大西洋。类群IV形成一个石鲈属较大的分支,其包括的6种石鲈主要分布在印度-西太平洋。其它石鲈均与分布于东太平洋(新大陆)的石鲈亚科其它属的种类聚一起,大棘石鲈与异孔石鲈属+仿石鲈属+八带石鲈,侧扁石鲈与小仿石鲈属,巴拿石鲈、纵带石鲈与锯鳃石鲈属,除了纵带石鲈外,其它三种石鲈均分布于东太平洋。
除了石鲈属鱼类初步揭示并非单系群外,仿石鲈属(Haemulon)与小仿石鲈属(Haemulopsis)在进化树上同样显示为非单系群。在仿石鲈属的分支中包含了异石鲈属(北美异石鲈X. californiensis),小仿石鲈属分支中包含了石鲈属(侧扁石鲈 P. corvinaeformis)的种类。对于北美异石鲈,Jordan等(1882)曾采用多个形态学与解剖学的数据将其与仿石鲈属的种类进行比较分析,发现其与仿石鲈属的种类具有许多相似的形态学特征,Sanciangco等(2011)利用多个分子标记构建系统进化树也验证了北美异石鲈位于仿石鲈属类群内部,认为北美异石鲈应该归为仿石鲈属,学名改为Haemulon californiensis。对于侧扁石鲈 P.commersonnii,前人有研究认为它应该归为小仿石鲈属(Haemulopsis)(López,1981),Tavera等(2012)在北美石鲈亚科鱼类系统发育研究中也揭示,在分子水平上,侧扁石鲈 P. corvinaeformis位于小仿石鲈属(Haemulopsis)内部,学名改为: Haemulopsiscorvinaeformis。本研究的结果也支持这上述两种鱼类分类的观点。
4 结论本研究利用DNA条形码基因COⅠ序列分析了印度-太平洋海区石鲈亚科7个属29种鱼类分子系统进化关系,并探讨COⅠ序列在石鲈亚科鱼类物种分子鉴定的有效性。结果显示COⅠ序列在不同石鲈亚科鱼类之间差异均大于2%,能有效区分不同物种,可作为石鲈亚科物种有效鉴定的条形码基因。而基于COⅠ序列构建的分子系统进化树,石鲈亚科中归为同一属的物种并没有构成严格的单系群,但其分类关系与其各自的地理分布有一定的相关性。由于全球性分布的石鲈亚科鱼类数量众多,且不同地理分布的物种可能出现了较大的遗传分化,COⅠ序列长度仅为650 bp左右,能提供的系统发育信息有限。故要澄清石鲈属以及石鲈亚科各种属直接的分类进化关系,需要更广泛采集石鲈亚科各种属的代表性样品,结合多个分子标记的序列的分析才得以有效解决。
| 毛云涛,甘小妮,王绪祯, 2014.基于线粒体COⅠ基因的沙鳅亚科鱼类DNA条形码及其分子系统发育研究.水生生物学报, 38(4):737-744 |
| 成庆泰,郑葆珊, 1987.中国鱼类系统检索.北京:科学出版社, 339-341 |
| 朱世华,郑文娟,邹记兴等, 2006. 5种石鲈科鱼类细胞色素b基因序列及分子系统分析.热带海洋学报, 25(4):42-45 |
| 任岗,章群,钱开诚等, 2007. 12种石鲈科鱼类线粒体16S rRNA基因的部分序列分析.热带海洋学报, 26(3):48-52 |
| 沈世杰, 1993.台湾鱼类志.台北:台湾大学动物学系出版, 360-363 |
| 宋超,于亚男,张涛等, 2014.基于线粒体COⅠ基因部分序列的长江口舌鳎科鱼类系统分类研究.动物学杂志, 49(5):716-726 |
| 孟庆闻,苏锦祥,缪学祖, 1995.鱼类分类学.北京:中国农业出版社, 636-640 |
| 黄宗国, 1994.中国海洋生物种类与分布.北京:海洋出版社, 708-709 |
| Carpenter K E, Niem V H, 2001. FAO Species Identification Guide for Fishery Purposes. ISSN 1020-6868. The Living Marine Resources of the Western Central Pacific. Volume 5:Bony fishes part 3(Menidae to Pomacentridae). FAO, Rome,2961-2989 |
| FAO-FIES, 2014. Aquatic Sciences and Fisheries Information System (ASFIS) species list. http://www.fao.org/fishery/collection/asfis/en |
| Froese R, Pauly D, 2013. FishBase. World Wide Web Electronic Publication. www.fishbase.org, version (9/2013) |
| Hebert P D N, Cywinska A, Ball S L et al, 2003a. Biological identifications through DNA barcodes. Proc R Soc Lond B, 270(1512):313-321 |
| Hebert P D N, Ratnasingham S, deWaard J R, 2003b. Barcoding animal life:cytochrome c oxidase subunit 1 divergences among closely related species. Proc R Soc Lond B, 270(S1):S96-S99 |
| Hubert N, Hanner R, Holm E et al, 2008. Identifying Canadian freshwater fishes through DNA barcodes. Plos One, 3(6):e2490 |
| Johnson S B, Warén A, Vrijenhoek R C, 2008. DNA barcoding of Lepetodrilus limpets reveals cryptic species. J Shellfish Res, 27(1):43-51 |
| Jordan D S, Gilbert C H, 1882. Synopsis of the fishes of North America. Bulletin of the United States National Museum, 16:1-1018 |
| Keskin E, Ağdamar S, Tarkan A S, 2013. DNA barcoding common non-native freshwater fish species in Turkey:Low genetic diversity but high population structuring. Mitochondrial DNA, 24(3):276-287 |
| Keskin E, Atar H H, 2013. DNA barcoding commercially important fish species of Turkey. Mol Ecol Resour, 13(5):788-797 |
| López M, 1981. Los "roncadores" del género Pomadasys (Haemulopsis) (Pisces:Haemulidae) de la costa Pacífica de Centro América. Rev Biol Trop, 29(1):83-94 |
| Mckay R J, 1984. Haemulidae. In:Fischer W, Bianchi G eds. FAO species identification sheets for fishery purposes. Western Indian Ocean (Fishing Area 51). Vol.2. FAO, Rome |
| Pereira L H G, Hanner R, Foresti F et al, 2013. Can DNA barcoding accurately discriminate megadiverse Neotropical freshwater fish fauna? BMC Genet, 14:20 |
| Randall J E, 2005. Reef and Shore Fishes of the South Pacific:New Caledonia to Tahiti and the Pitcairn Islands. Honolulu, Hawaii:University of Hawaii Press, 720 |
| Sanciangco M D, Rocha L A, Carpenter K E, 2011. A molecular phylogeny of the Grunts (Perciformes:Haemulidae) inferred using mitochondrial and nuclear genes. Zootaxa, 2966:37-50 |
| Schlei O L, Crête-Lafrenière A, Whiteley A R et al, 2008. DNA barcoding of eight North American coregonine species. Mol Ecol Resour, 8(6):1212-1218 |
| Swofford D P, 2003. Phylogenetic Analysis Using Parsimony (and Other Methods) Version 10. Sunderland, MA:Sinauer |
| Tamura K, Peterson D, Peterson N et al, 2011. MEGA5:Molecular evolutionary genetics analysis using maximum likelihood, evolutionary distance, and maximum parsimony methods. Mol Biol Evol, 28(10):2731-2739 |
| Tavera J J, Acero P A, Balart E F et al, 2012. Molecular phylogeny of grunts (Teleostei, Haemulidae), with an emphasis on the ecology, evolution, and speciation history of New World species. BMC Evol Biol, 12:57 |
| Thompson J D, Higgins D G, Gibson T J, 1994. CLUSTAL W:improving the sensitivity of progressive multiple sequence alignment through sequence weighting, position-specific gap penalties and weight matrix choice. Nucl Acids Res, 22(22):4673-4680 |
| Ward R D, Zemlak T S, Innes B H et al, 2005. DNA barcoding Australia's fish species. Phil Trans R Soc B, 360:1847-1857 |
 2016, Vol. 47
2016, Vol. 47

