中国海洋湖沼学会主办。
文章信息
- 万茜, 张扬, 张跃环, 喻子牛. 2015.
- WAN Qian, ZHANG Yang, ZHANG Yue-Huan, YU Zi-Niu. 2015.
- 香港牡蛎(Crassostrea hongkongensis)水通道蛋白基因AQP1的克隆、分子特性和表达分析
- MOLECULAR CLONING, CHARACTERIZATION, AND EXPRESSION OF AQUAPORIN1 GENE IN CRASSOSTREA HONGKONGENSIS
- 海洋与湖沼, 46(5): 1078-1087
- Oceanologia et Limnologia Sinica, 46(5): 1078-1087.
- http://dx.doi.org/10.11693/hyhz20140400127
-
文章历史
- 收稿日期: 2014-04-27
- 收修改稿日期: 2014-08-19
2. 南海资源开发与保护协同创新中心 广州 510275;
3. 中国科学院大学 北京 100049
2. South China Sea Bio-resource Exploitation and Utilization Collaborative Innovation Center, Guangzhou 510275, China;
3. University of Chinese Academy of Sciences, Beijing 100049, China
水通道蛋白(Aquaporin,AQP)是一类参与生命活动中水分子跨膜运输的调控蛋白,属于主要内嵌蛋白超家族成员(major intrinsic protein superfamily),它主要介导水分由低渗区向高渗区的跨膜转运,也能转运甘油、尿素、氨水等中性小分子。自从Pretson等(1992)发现了第一个水通道蛋白1(CHIP28)以来,研究人员已经发现水通道蛋白广泛分布于古菌、真菌和真核生物组成的三界系统中。水通道蛋白在脊椎动物和植物中已经有很多研究报道,人类13种水通道蛋白(从水通道蛋白0到12)已经被发现并且鉴定; 但是,无脊椎动物水通道蛋白的研究相对较少,并且主要集中在模式生物线虫、果蝇和昆虫中; 在软体动物中研究甚少(Yang,2000; Huang et al,2007; Campbell et al,2008; Ishibashi et al,2011; Pieńkowska et al,2014)。水通道蛋白是一个多亚型的蛋白家族,根据氨基酸序列的同源性可以分为四种类型: 第一类为AQP1-like型(包括AQP0,1,2,4,5和6,属于典型的传统AQP,只转运水分子),第二类为AQP3-like型(包括AQP3,7,9和10,属于水甘油通道蛋白,除了水分子,还可以转运甘油、尿素、氨水等中性小分子),第三类为AQP8-like型(只包括AQP8这一种传统水通道蛋白),第四类为AQP11-like型(包括AQP11和12,属于非传统AQP)(Soto et al,2012)。所有的水通道蛋白都是以同源四聚体形式存在于细胞膜表面,每个单体包括6个跨膜区段(Ⅰ—Ⅵ)和5个环形结构(loopA-E)。根据“砂漏”模型(Agre et al,1993),B环和E环从两侧半嵌入膜内形成一个开放式狭窄的水通道。保守的NPA基序(Asn-Pro-Ala)和选择性水孔构件ar/R(aromatic/arginine,芳香族/精氨酸区)被认为是最主要的控制水或其它中性小分子渗透性的关键区域(Eric et al,2006)。
香港牡蛎(Crassostrea hongkongensis)是我国华南沿海养殖的主要经济种,主要分布在广东、广西等地,年产量在130多万t,产值在80—100亿元(中国渔业年鉴,2013)。香港牡蛎与其它牡蛎种类相比较,其价值主要体现在体大肉肥、味道鲜美,另外其市场价值远高于其它种类牡蛎,深受广东、广西、港澳、东南亚一些国家消费者青睐(张跃环等,2012)。作为河口区域典型牡蛎经济种(Lam et al,2003),它对低盐有较强的抗性,但对高盐仍具有一定的耐受性,更适宜于栖息在河口咸淡水环境(谢忠明,2003)。水通道蛋白作为促进水分子跨膜运输的重要蛋白,参与渗透平衡的调节,在盐度变化剧烈的环境中发挥重要作用。目前,对编码牡蛎水通道蛋白的基因研究甚少,且不清楚盐度胁迫下水通道蛋白基因的表达特性。因此,本文采用RACE技术首次克隆了香港牡蛎水通道蛋白1(AQP1)基因cDNA全长,分析该基因编码蛋白序列特征、理化性质和空间结构,通过实时荧光定量PCR技术分析该基因在香港牡蛎中的组织分布及7天盐度胁迫下的表达情况,旨在为进一步研究香港牡蛎的广盐适应机制提供分子理论基础。
1 材料与方法 1.1 供试材料、盐度处理及取样实验材料为壳高在120mm左右的2龄香港牡蛎,采自广东省阳江市阳西县程村。在实验室27°C,盐度为18海水中暂养一周后,选取健康香港牡蛎个体,快速吸取出淋巴液,离心(12000 r/min,30s)后弃上清,立即加入等量TRIzol reagent(Invitrogen公司)重悬沉淀,继续提取RNA或者冻存于-80°C。另外,取下小块外套膜、鳃、闭壳肌、消化腺和整个心脏,加入1mL TRIzol reagent,匀浆,继续提取RNA或者冻存于-80°C。
盐度处理时,将200只健康牡蛎随机分为4组,即4盐度组、18盐度组、32盐度组和42盐度组。由于香港牡蛎最适盐度范围为10—20(谢忠明,2003),所以本实验以18盐度组作为对照组。用0.8%扁藻喂养,并且每天换3次海水。处理开始后,在时间节点1天(24h)、3天(72h)、5天(120h)和7天(168h)对四组分别随机挑取5个个体,取出一小块鳃组织,立即加入TRIzol,匀浆后冻存。
1.2 试剂与仪器大肠杆菌菌株DH5α、克隆载体pMDTM19-T、exTaq酶、DNAmarker、T4 DNA连接酶、DNA片段纯化回收试剂盒、琼脂糖凝胶回收试剂盒、逆转录试剂盒PrimeScriptⅡ1st Str and cDNA Synthesis Kit 、用于定量实验的逆转录试剂盒Prime Script RT reagent Kit with gDNA Eraser 购于宝生物工程(大连)有限公司。TRIzol试剂购于Invitrogen公司。RACE试剂盒BD SMART RACE cDNA Amplification kit 购于Clontech公司。实时定量试剂盒2×SYBR Green Master Mix 购于Roche公司。用罗氏的LightCycler 480Ⅱ仪器进行Real-time PCR。引物合成和测序工作在北京六合华大进行。
1.3 香港牡蛎总RNA提取与cDNA第一链的合成根据TRIzol reagent(Invitrogen公司)的产品说明操作提取淋巴及匀浆后的各个组织总RNA,用Nanodrop 2000c(Thermo公司)测定RNA的纯度和浓度,并用电泳检测RNA质量。用PrimeScriptⅡ1st Str and cDNA Synthesis Kit进行cDNA第一链的合成,反转录产物用于PCR扩增或者保存在-20°C。
1.4 香港牡蛎AQP1基因片段的RT-PCR扩增根据在NCBI 太平洋牡蛎EST数据库中获得的一段序列,设计引物ChAQP1F1和ChAQP1R1(表 1),以混合各个组织cDNA第一链为模板进行PCR扩增。反应条件为: 94°C预变性3min; 94°C变性30s,50°C退火 30s,72°C延伸1min,共30个循环; 最后72°C延伸10min。PCR产物经1.2%琼脂糖凝胶电泳后,切胶回收纯化目的片段。将纯化产物连接至pMD19-T质粒载体上,转化至E. coli DH5α感受态细胞中,培养过夜,挑取单克隆进行菌落PCR验证,阳性克隆送北京六合华大基因科技股份有限公司测序。测序结果在NCBI上进行Blast比对,初步确定获得片段为香港牡蛎水通道蛋白1基因。
| 引物名称 | 引物序列(5'—3') | 注释 |
| GR5P | CGACTGGAGCACGAGGACACTGA | 5'接头引物 |
| GR5NP | GGACACTGACATGGACTGAAGGAGTA | |
| GR3P | GCTGTCAACGATACGCTACGTAACG | 3'接头引物 |
| GR3NP | CGCTACGTAACGGCATGACAGTG | |
| ChAQP1F1 | TACCTTGTTGTTTGTGTTCCTG | 中间片段引物 |
| ChAQP1R1 | TGTGTAGCCTCTTCGTTTGATA | |
| ChAQP1R2 | CTGGCTAATAACCCCAAGGAAACG | 5'RACE 引物 |
| ChAQP1R3 | CTCCAACGATGGCACCGATTATTT | |
| ChAQP1F2 | GGGACTATGACACACACTGGGTAT | 3'RACE 引物 |
| ChAQP1F3 | GATTGGTCTCACTGTTGCGATGCT | |
| ChAQP1F4 | AAATCAACGGCAGACAACAAACT | 全长引物 |
| ChAQP1R4 | ATGTGCGTCACTTCTAAATCCTT | |
| ChAQP1F5 | ATTCTGACATTTTGCCTCGTTTTCG | ChAQP1定量引物 |
| ChAQP1R5 | GGAGCATCGCAACAGTGAGACCAATC | |
| β-actin F1 | AAAAAGCTTCCATGAGGGTATTCT | 内参β-actin定量引物 |
| β-actin R1 | TTTCTCGAGTCGGTATCTATCTGGT | |
| GAPDHF1 | GGATTGGCGTGGTGGTAGAG | 内参GAPDH定量引物 |
| GAPDHR2 | GTATGATGCCCCTTTGTTGAGTC | |
| F代表正向引物, R代表反向引物 | ||
根据上述克隆获得的片段序列,设计用于5’RACE扩增的基因特异性引物ChAQP1R2和ChAQP1R3,用于3’RACE扩增的基因特异性引物ChAQP1F2和ChAQP1F3(表 1)。按照BD SMART RACE cDNA Amplification kit试剂盒说明,利用引物GR5P/ChAQP1R2和GR3P/ChAQP1F2进行5’RACE和3’RACE第一轮反应,反应条件为: 94°C 3min; 94°C 30s,72°C 2min,5个循环,94°C 30s,70°C 30s,72°C 2min,5个循环,94°C 30s,68°C 30s,72°C 2min,25个循环; 72°C 10min。以第一轮PCR产物为模板,引物GR5NP/ChAQP1R3和GR3NP/ChAQP1F3进行第二轮反应,反应程序为: 94°C 3min; 94°C 30s,55°C 30s,72°C 2min,25个循环; 72°C 10min。PCR产物进行切胶回收纯化,连T载体,转化,挑克隆,送测序,获得测序结果经Blast比对后,用DNAstar软件拼接所得序列片段,最后获得完整全长序列。
根据已拼接的基因序列设计特异性引物ChAQP1F4和ChAQP1R4(表 1),PCR扩增香港牡蛎AQP1的编码区,反应条件为: 94°C 3min; 94°C 30s,55°C 30s,72°C 2min,30个循环; 72°C 10min。1.2%琼脂糖凝胶电泳分析扩增结果,测序验证。
1.6 序列分析、结构预测及分子进化树的构建从NCBI蛋白数据库中获得水通道蛋白1同源序列,MatGAT2.02软件分析氨基酸序列的一致性(Tamura et al,2007)。ProtParam(http: //web.expasy.org/ protparam/)分析蛋白质基本理化性质。蛋白质保守结构域的预测由在线软件Simple Modular Architecture Research Tool(SMART)完成(http: //smart.emblheidel- berg.de/)。氨基酸多序列分析由ClustalX 1.81软件完成。分子进化树由MEGA 4.0软件构建获得。蛋白质亲水性分析通过ProtScale在线服务器预测(http: //web.expasy.org/cgi-bin/protscale/protscale.pl)(Kyte et al,1982)蛋白质三级结构预测在SWISS-MODEL中进行,用Rasmol 2.7.0.1查看预测的结构。
1.7 实时定量PCR分析以香港牡蛎不同组织总RNA及不同盐度胁迫下鳃组织中总RNA 1µg为底物,按照Prime Script RT reagent Kit with gDNA Eraser产品说明合成用于实时定量PCR的单链cDNA模板。根据全长AQP1序列信息,设计特异定量引物ChAQP1F5和ChAQP1R5; 以看家基因β-actin为内参基因,设计内参定量引物β-actin F1和β-actin R1(表 1),用于测定香港牡蛎AQP1基因在不同组织中的时空表达量; 以看家基因GAPDH为内参基因,设计内参定量引物GAPDHF1和GAPDH R1(表 1),用于测定香港牡蛎AQP1基因在不同盐度胁迫下的表达量; 定量PCR反应体系20µL为: 10 µmol/L的上、下游引物各2µL; 2×SYBR Green Master Mix(Roche,USA)10 µL; cDNA模板2 µL; 灭菌超纯水H2O 4µL。定量PCR反应条件为: 95°C预变性1min; 95°C变性10s,55°C退火15 s,72°C延伸20s 共40个循环。为检测定量PCR的特异性,在反应结束时进行熔融曲线分析。数据由LightCycler 480Ⅱ1.5软件生成并且记录。采用${{\text{2}}^{-\Delta \Delta \text{Ct}}}$法分析香港牡蛎AQP1 mRNA在不同组织中的相对表达量。
1.8 数据分析采用SPSS软件包单因素方差分析法(one-way ANOVA)中的Duncan多重比较检验法对实时定量PCR中的数据进行分析,比较香港牡蛎AQP1在组织分布及不同盐度胁迫下鳃中的相对表达量(平均值±标准差)差异。
2 结果 2.1 香港牡蛎ChAQP1基因的克隆和分析以香港牡蛎各个组织混合cDNA为模板,使用引物ChAQP1F1和ChAQP1R1进行PCR扩增,扩增产物经1%琼脂糖凝胶电泳检测获得约600bp的核酸条带(图 1a),测序获得核酸序列。将核酸序列在NCBI中Blast比对,初步确定获得香港牡蛎AQP1基因序列。再用特异性引物ChAQP1R2、ChAQP1R3和ChAQP1F2、ChAQP1F3分别进行5’RACE和3’RACE两轮PCR扩增,分别获得1条大小约503 bp(图 1b)和353 bp(图 1c)的核酸条带。将3段序列拼接后得到长度为1153 bp的香港牡蛎水通道蛋白1基因全长序列。其5’非编码区(UTR)长度为165 bp,3’非编码区长度为100 bp,基因编码区长为888 bp,编码295个氨基酸(图 2)。设计包含编码区及以外若干序列的特异引物ChAQP1F4和ChAQP1R4扩增获得1060 bp的核酸条带(图 1d),经测序验证为目的基因,将此基因命名为ChAQP1。将序列提交至GenBank,获得登录号KJ704847。
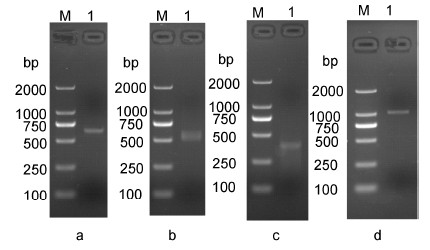 |
| 图 1 香港牡蛎ChAQP1 PCR 扩增结果 Fig. 1 The PCR amplification products of ChAQP1 in C. hongkongensis M: DL2000 marker; a: 中间片段; b: 5'端片段扩增; c: 3'端片段扩增; d: 全长cDNA片段 |
Protparam在线预测软件表明该蛋白分子式为C1463H2278N370O400S14,分子量为31.89 kDa,等电点为8.61,负电荷氨基酸总残基数(Asp+Glu)为21,正电荷氨基酸总残基数(Arg+Lys)为24,脂肪系数为99.19,亲水性总平均数为0.439,为脂溶性蛋白,不稳定系数为20.16,属于一类稳定的蛋白。
SMART在线服务软件对获得的ChAQP1基因进行蛋白质结构域预测,结果表明有1个与该基因匹配的蛋白质结构域MIP(major intrinsic protein superfamily),位于第31—243位氨基酸(图 2)。
 |
| 图 2 ChAQP1的全长cDNA序列及推导的氨基酸序列 Fig. 2 The cDNA sequence of ChAQP1 and deduced amino acid sequences ORF区用大写字母显示, 5’UTR和3’UTR用小写字母显示。灰色阴影标示SMART预测的MIP结构域。椭圆框显示的是NPA基序。终止子用“*”表示 |
ProtScale分析蛋白质疏水性表明,该蛋白二级结构中具有6个跨膜螺旋TM1—TM6,5个连接环A—E(图 3)。
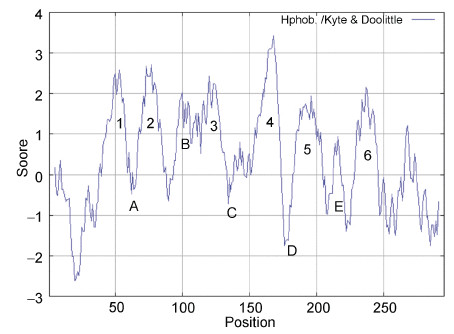 |
| 图 3 ChAQP1疏水性图 Fig. 3 Hydropathy plots of ChAQP1 数字1—6表示跨膜结构域, 字母A—E表示5个连接环 |
MatGAT分析表明,ChAQP1氨基酸序列除了与太平洋牡蛎氨基酸序列一致性为87.8%以外,与其它的都只有30%左右的一致性(表 2)。由于与人类AQP1氨基酸序列一致性为40.2%,故选择人类AQP1(PDB ID:1H6I)为模板,利用SWISS-MODEL中Target- Template Alignment进行ChAQP1蛋白三级结构预测。结果发现(用Rasmol查看)两者有相似的结构,都存在六个跨膜结构域,2个半跨膜结构域和5个连接环(图 4、图 5)。
| 物种名 | GenBank 登录号 | 氨基酸一致性(%) |
| 香港牡蛎( Crassostrea hongkongensis) | KJ704847 | 100.0 |
| 太平洋牡蛎( Crassostrea gigas) | EKC24616 | 98.5 |
| 人类( Homo sapiens) | NP_932766 | 40.3 |
| 牛( Bos taurus) | NP_777127 | 37.0 |
| 小家鼠( Mus musculus) | NP_031498 | 38.6 |
| 非洲爪蟾( Xenopus laevis) | NP_001085391 | 39.3 |
| 斑马鱼( Danio rerio ) | NP_996942 | 39.9 |
| 大西洋鲑鱼( Salmo salar) | NP_001133472 | 39.7 |
| 静水椎实螺( Lymnaea stagnalis) | AHL29026 | 33.2 |
| 沼泽椎实螺( Stagnicola palustris) | AHL29028 | 33.5 |
| 池塘椎实螺( Catascopia terebra) | AHL29027 | 32.6 |
| 线虫( Caenorhabditis elegans) | CCD66276.1 | 22.5 |
| 克什曼血吸虫( Schistosoma mansoni) | ACI31185 | 22.2 |
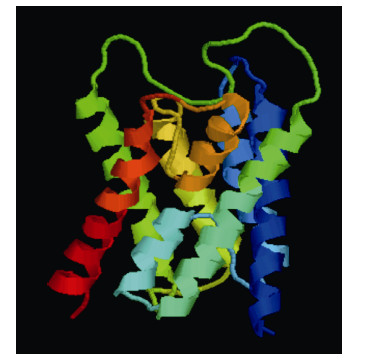 |
| 图 4 ChAQP1蛋白预测的三级结构 Fig. 4 The predicted 3D structure of ChAQP1 |
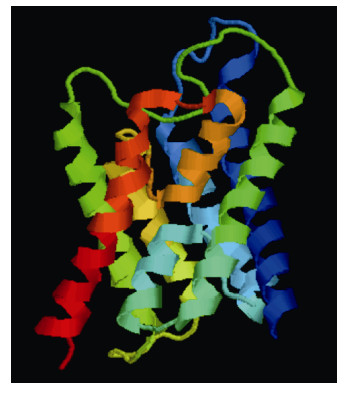 |
| 图 5 1H6I蛋白三级结构 Fig. 5 The 3D structure of 1H6I |
Clustal 同源多序列比对结果(图 6)表明ChAQP1有两个保守的NPA(天冬酰胺-脯氨酸-丙氨酸)结构基序,位于第93—95和第208—210位氨基酸处,这与其它物种AQP1中序列一致。ar/R选择性水孔构件由F73、H196、S205和R211四个位点构成,除了S205位点相对不保守,其它三个位点存在高度保守性,这表明ChAQP1属于传统的水通道蛋白,只允许水分子通过该孔道。另外,在第一个NPA基序上游发现有ISGGH序列(第87—91位),在其它传统AQP氨基酸序列中也普遍存在。此结果进一步说明,水通道蛋白1的NPA基序和ar/R结构在不同物种中存在很高的进化保守性。
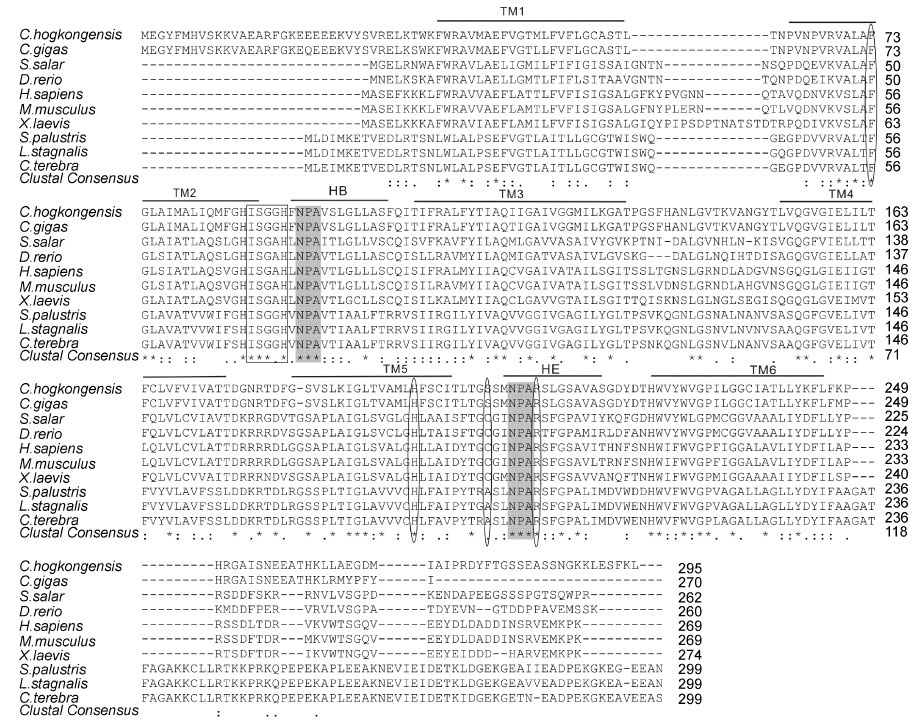 |
| 图 6 ChAQP1与其它物种AQP1氨基酸序列比对 Fig. 6 Multiple alignment of ChAQP1 amino acid sequences with other AQP1 proteins “*”代表一致, “: ”代表高度保守, “.”代表低度保守。2个阴影部分代表2个NPA基序, 椭圆标示ar/R结构并且方框标示ISSGGH序列。横线标示保守的跨膜结构域。AQPl 来源及GenBank登录号详见表2 |
使用MEGA 4.0软件中的N-J建树方法对ChAQP1蛋白序列和其它物种中的AQP1及AQP家族成员构建分子进化树(图 7)。结果显示,AQP家族成员聚类分成4个主要分支,即AQP1-like,AQP3-like,AQP8-like和AQP11-like四类,ChAQP1属于AQP1- like分支。将AQP1-like分支序列中AQP1序列重新构建进化树(图 8),结果表明,脊椎动物能较好地聚为一类,无脊椎动物则不能聚为一类。在无脊椎动物中,香港牡蛎与太平洋牡蛎聚为一类,亲缘关系最近,这显示了它们中水通道蛋白1相似性但也存在差异。
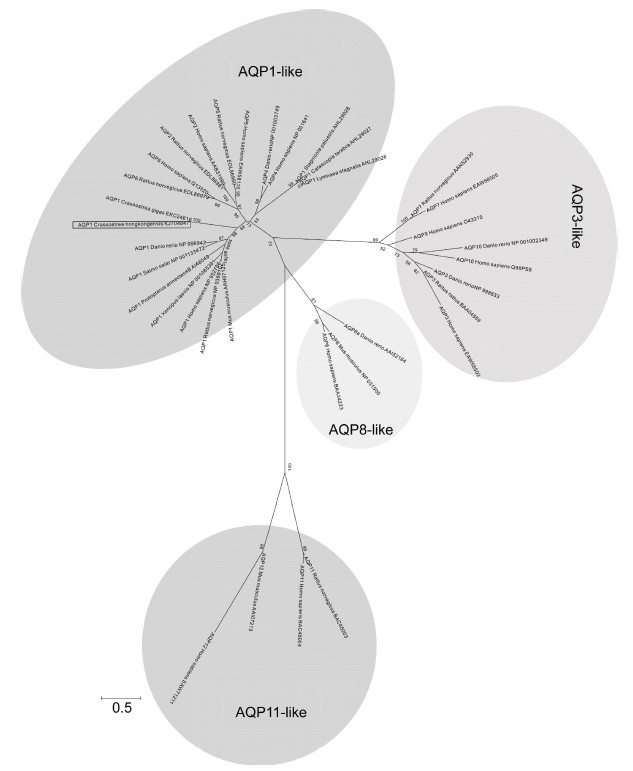 |
| 图 7 ChAQP1与其它物种水通道蛋白的系统进化分析 Fig. 7 Phylogenetic analysis of ChAQP1 with AQP family members of other species |
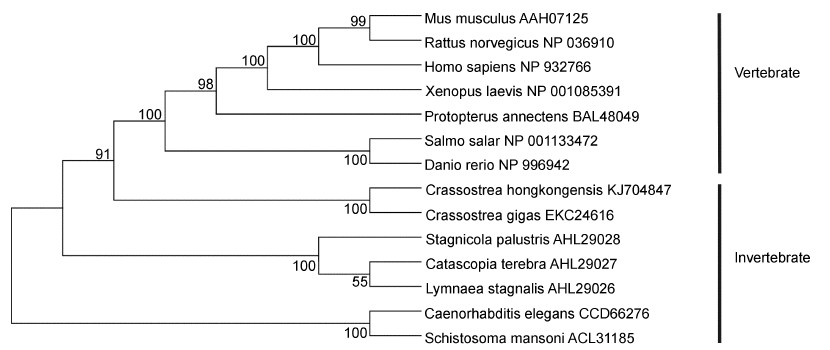 |
| 图 8 ChAQP1与其它物种AQP1的系统进化分析 Fig. 8 Phylogenetic analysis of ChAQP1 with AQP1 of other species |
ChAQP1 mRNA在各个组织中的表达量可以通过实时定量PCR的方法进行检测,结果表明ChAQP1基因在香港牡蛎鳃、外套膜、闭壳肌、消化腺、性腺、心脏和淋巴中都有表达,其中在闭壳肌中表达量最高,其次是外套膜、性腺、消化腺、鳃。心脏、淋巴中表达量较低(图 9)。
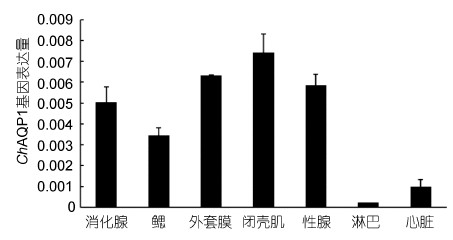 |
| 图 9 ChAQP1 mRNA在不同组织表达量分布图 Fig. 9 Tissue distribution of ChAQP1 mRNA expression level 每个bar代表平均值归一化的表达水平(n=3) |
7天盐度胁迫实验结果如图 10。香港牡蛎最适盐度为18,故以之作为对照组。低盐处理后(盐度为4),ChAQP1基因的表达量与对照组表达量相近,并随盐度胁迫时间的延长维持在一个相对稳定的水平,1天、3天、5天、7天分别为对照组的1.06、0.90、0.72、0.92倍。在32高盐胁迫下,ChAQP1基因的表达量在前五天呈现出略微下降的趋势,分别为对照组的0.65、0.76、0.86倍,而在第7天恢复正常水平(1.11倍)。42高盐胁迫下,ChAQP1基因的表达量在前五天迅速下降,随时间延长维持在一个较低水平,分别为对照组的0.18(P<0.01)、0.48、0.28(P<0.05)倍,在第7天恢复正常水平(1.03倍)。
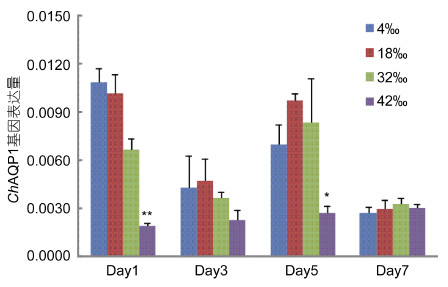 |
| 图 10 7天盐度处理对ChAQP1基因表达的影响 Fig. 10 Relative expression level of ChAQP1 was analyzed under salinity stress in 7 days 图中数据为平均值±S.E (n =5), 显著性差异用1个星号(P<0.05)和2个星号(P<0.01)标示 |
本研究采用RACE技术从香港牡蛎中克隆获得其香港牡蛎水通道蛋白1基因ChAQP1(KJ704847),对其进行蛋白保守区段预测发现,该蛋白具有一个典型的MIP结构域、6个跨膜螺旋、5个连接环。在氨基酸同源性序列比对中,ChAQP1含有两个保守的NPA基序,这与其它物种中水通道蛋白1一致; ar/R区氨基酸组成在F73、H196和R211这三个位点上与其它物种AQP1中的也是高度一致,这符合传统水通道蛋白的特性,ar/R区氨基酸形成的孔径正好允许水分子通过(Beitz et al,2006)。第一个NPA基序上游的ISGGH序列也是高度保守的,可见,ChAQP1是非常传统的,只允许水分子穿过双分子膜的水通道蛋白,在关键的功能结构域上具有较高的保守性。系统进化分析表明,该蛋白属于AQP-1like类型的水通道蛋白。对不同物种AQP1序列进行聚类分析发现,ChAQP1与同科同属的太平洋牡蛎AQP1遗传距离最近,与同为软体动物门的腹足纲蜗牛(Lymnaea stagnalis,Catascopia occulta和Stagnicola palustris)遗传距离比较近,而与哺乳动物中的人类及鼠遗传距离相对较远,符合其遗传进化关系。
组织分布结果显示ChAQP1 mRNA 在香港牡蛎主要组织鳃、外套膜、闭壳肌、消化腺、性腺、心脏和淋巴中都有表达,其中鳃、外套膜、闭壳肌、消化腺和性腺表达量相对较高些,这说明着ChAQP1可能在转运水分子的相关生理过程中起到重要作用。Moon等(1993)研究发现人类AQP1也存在各个组织器官组成型表达的现象,人类AQP1广泛分布于红细胞、肾、眼、肺和血管内皮细胞等组织中。Pieńkowska等(2014)研究蜗牛AQP1,发现在脑、肾、足和消化腺等组织器官中都有广泛分布。Tipsmark等(2010)发现大西洋鲑鱼AQP-1a也广泛分布在肠、肾、鳃、脑等组织中。在7天盐度胁迫实验中,以盐度18作为对照,当盐度达到42时,ChAQP1基因表达受到抑制,而在低盐(4)组没有太大的变化,在高盐(32)组呈现出略微下降的趋势。这与Meng等(2013)利用转录组数据分析太平洋盐度适应性时,发现的3个AQP基因在高盐(盐度为40)和低盐(盐度为10)下均为呈下调趋势的结果不太一致。这是因为太平洋牡蛎最适盐度为30,为了维持细胞膨胀与收缩的稳定,机体通过增加蛋白质磷酸化来降低水通道蛋白活性,从而保护机体免于更多水分的进入,而在盐度4到32范围内,香港牡蛎ChAQP1基因表达量和正常盐度下表达量波动不是很大,这说明在该盐度范围内AQP1基因参与盐度调节的能力是有限的,尤其是在低盐下。这种现象同样出现在鲈鱼中,Ivone等发现鲈鱼在低盐下,鳃组织中AQP1基因表达量并没有变化(Giffard-Mena et al,2007)。而在高盐下,AQP1基因表达量普遍存在下调趋势。在42盐度下,香港牡蛎ChAQP1基因表置信水平通过在每个分支节点上进行1000次重复获得。标尺显示发生0.2%概率氨基酸位点变异的长度。序列信息由缩写名、物种全名和登录号构成。方框标示ChAQP1蛋白达量显著下降,这说明机体通过降低该基因的表达来降低AQP1蛋白的活性,抑制机体内水分的流失,保持机体内水盐平衡,提高机体对逆境的耐受力。同样的现象也出现在大西洋鲑鱼中,从淡水到海水的过程中,鲑鱼AQP1-a基因在鳃中表达量也明显下调(Tipsmark et al,2010)。以上结果表明,在香港牡蛎中,AQP1参与了调节渗透压平衡,为探讨香港牡蛎广盐适应性提供一定的实验数据和根据。
| 农业部渔业局, 2013.中国渔业年鉴.北京:中国农业出版社 |
| 张跃环,王昭萍,闫喜武等, 2012.香港巨牡蛎和长牡蛎幼虫及稚贝的表型性状.生态学报32(4):1105-1114 |
| 谢忠明, 2003.海水经济贝类养殖技术.北京:中国农业出版社, 557-578 |
| Agre P, Preston GM, Smith BL et al, 1993. Aquaporin CHIP:the archetypal molecular water channel. American Journal of Physiology-Renal Physiology, 265(4):F463-F476 |
| Beitz E, Wu B H, Holm L M et al, 2006. Point mutations in the aromatic/arginine region in aquaporin 1 allow passage of urea, glycerol, ammonia, and protons. Proceedings of the National Academy of Sciences of the United States of America, 103(2):269-274 |
| Campbell E M, Ball A, Hoppler S et al, 2008. Invertebrate aquaporins:a review. Journal of Comparative Physiology B, 178(8):935-955 |
| Giffard-Mena I, Boulo V, Aujoulat F et al, 2007. Aquaporin molecular characterization in the sea-bass(Dicentrarchus labrax):The effect of salinity on AQP1 and AQP3 expression. Comparative Biochemistry and Physiology Part A:Molecular & Integrative Physiology, 148(2):430-444 |
| Huang C G, Lamitina T, Agre P et al, 2007. Functional analysis of the aquaporin gene family in Caenorhabditis elegans. American Journal of Physiology-Cell Physiology, 292(5):C1867-C1873 |
| Ishibashi K, Kondo S, Hara S et al, 2011. The evolutionary aspects of aquaporin family. American Journal of Physiology-Regulatory, Integrative and Comparative Physiology, 300(3):R566-R576 |
| Kyte J, Doolittle R F, 1982. A simple method for displaying the hydropathic character of a protein. Journal of Molecular Biology, 157(1):105-132 |
| Lam K, Morton B, 2003. Mitochondrial DNA and morphological identification of a new species of Crassostrea(Bivalvia:Ostreidae) cultured for centuries in the Pearl River Delta, Hong Kong, China. Aquaculture, 228(1-4):1-13 |
| Meng J, Zhu Q H, Zhang L L et al, 2013. Genome and transcriptome analyses provide insight into the euryhaline adaptation mechanism of Crassostrea gigas. PloS ONE, 8(3):e58563 |
| Moon C, Preston G M, Griffin C A et al, 1993. The human aquaporin-CHIP gene. Structure, organization, and chromosomal localization. The Journal of Biological Chemistry, 268(21):15772-15778 |
| Pieńkowska J R, Kosicka E, Wojtkowska M et al, 2014. Molecular Identification of First Putative Aquaporins in Snails. The Journal of Membrane Biology, http://dx.doi.org/10.1007/s00232-014-9629-0 |
| Preston G M, Carroll T P, Guggino W B et al, 1992. Appearance of water channels in Xenopus oocytes expressing red cell CHIP28 protein. Science, 256(5055):385-387 |
| Soto G, Alleva K, Amodeo G et al, 2012. New insight into the evolution of aquaporins from flowering plants and vertebrates:Orthologous identification and functional transfer is possible. Gene, 503(1):165-176 |
| Tamura K, Dudley J, Nei M et al, 2007. MEGA4:molecular evolutionary genetics analysis(MEGA) software version 4.0. Molecular Biology and Evolution, 24(8):1596-1599 |
| Tipsmark C K, Sørensen K J, Madsen S S, 2010. Aquaporin expression dynamics in osmoregulatory tissues of Atlantic salmon during smoltification and seawater acclimation. The Journal of Experimental Biology, 213(3):368-379 |
| Yang B X, 2000. The human aquaporin gene family. Current Genomics, 1(1):91-102 |
 2015, Vol. 46
2015, Vol. 46


