中国海洋湖沼学会主办。
文章信息
- 吴彪, 杨爱国, 董迎辉, 郑利兵, 刘志鸿, 周丽青, 孙秀俊, 郑言鑫. 2015.
- WU Biao, YANG Ai-Guo, DONG Ying-Hui, ZHENG Li-Bing, LIU Zhi-Hong, ZHOU Li-Qing, SUN Xiu-Jun, ZHENG Yan-Xin. 2015.
- 虾夷扇贝选育群体与野生群体基因组DNA甲基化的MSAP分析
- MSAP ANALYSIS ON GENOME-WIDE DNA METHYLATION IN SELECTED AND WILD JAPANESE SCALLOP PATINOPECTEN YESSOENSIS
- 海洋与湖沼, 46(4): 976-982
- Oceanologia et Limnologia Sinica, 46(4): 976-982.
- http://dx.doi.org/10.11693/hyhz20141000267
-
文章历史
- 收稿日期:2014-10-11
- 改回日期:2015-03-13
2. 浙江省水产种质资源高效利用技术研究重点实验室 浙江万里学院 宁波 315100;
3. 上海海洋大学水产与生命学院 上海 201306
2. Zhejiang Key Laboratory of Aquatic Germplasm Resources, Zhejiang Wanli University, Ningbo 315100, China;
3. College of Fisheries and Life Science, Shanghai Ocean University, Shanghai 201306, China
虾夷扇贝(Patinopecten yessoensis)是一种冷水性的扇贝,广泛分布于朝鲜半岛北部、日本北海道、俄罗斯远东地区的沿海,20世纪80年代初由辽宁省水产科学研究院从日本引入我国进行养殖(Li et al,2007; 常亚青等,2007)。经过多年的发展,虾夷扇贝在辽宁、山东等沿海形成了规模化养殖,近些年的年产量维持在20多万吨,已经成为我国重要的养殖经济贝类(吴彪等,2013)。但近些年来,虾夷扇贝在主养殖区的死亡率居高不下,有时夏季成贝的死亡率超过50%,严重影响了产业的健康稳定发展,其中过度养殖、环境恶化、病原侵袭、种质质量下降是造成虾夷扇贝大规模死亡的重要原因(刘卫东等,2009; 于佐安等,2011; 张明明等,2008)。为对虾夷扇贝进行种质改良,本课题组经过多年的累代选育,培育出一个左壳白色的“玉贝”虾夷扇贝新品系,并对其进行了生长、遗传等方面的研究,表明“玉贝”具有明显的生长和耐高温优势(程鹏等,2010,2011)。
DNA甲基化(DNA methylation)是指在DNA甲基化转移酶的作用下,以S-腺苷甲硫氨酸(S-adenosyl- L-methionine)作为供体,将甲基基团转移到基因组CpG二核苷酸的胞嘧啶5' 碳位,从而形成5-甲基胞嘧啶(Bird,1992)。它作为重要的表观遗传学修饰方式,广泛存在于动植物的基因组中,在基因表达、细胞分化、胚胎发育等生物学过程中发挥了重要作用,近年来已经成为遗传学的研究热点(Elango et al, 2008; 郭磊等,2010; Suarez-Alvarez et al, 2012)。除了生长发育过程中的调控作用,在植物和脊椎动物的研究中发现,DNA甲基化不仅与杂种优势具有一定的关联性(仪治本等,2005; 万亚琴,2008),还能够积极响应环境胁迫(周新文等,2001; 杨金兰等,2007; 潘雅姣等,2009; 朱华平等,2013),而且这种DNA甲基化还可以遗传给后代。研究DNA甲基化的方法很多,其中甲基化敏感扩增多态性(methylation-sensitive amplification polymorphism,MSAP)是目前研究DNA甲基化简便、高效而且非常可靠的一种技术,它在AFLP的基础上发展而来,主要是基于同裂酶MspⅠ和HpaⅡ的酶切识别位点都是CCGG,但对胞嘧啶甲基化敏感性不同的原理上而进行的。MSAP分析不需要获知基因组信息,所以在分析已知信息相对匮乏的基因组DNA甲基化上具有明显的优势。
运用MSAP技术分析DNA甲基化已经在植物和脊椎动物中广泛开展(陈培利等,2000;Hu et al, 2013 ; Powell et al, 2013),无脊椎动物基因组DNA甲基化的研究资料相对匮乏。但最近几年,无脊椎动物中MSAP研究的发展非常迅速。于涛(2010)、吴彪(2012)等人运用MSAP技术研究分析了扇贝杂交优势与DNA甲基化的相关性; 吕佳等(2013)人对栉孔扇贝(Chlamys farreri)、海湾扇贝(Argopecten irradians)、虾夷扇贝全基因组DNA甲基化水平进行了比较分析; Sun等(2014)分析比较了栉孔扇贝5个不同组织的DNA甲基化水平; Gavery等(2010)研究了太平洋牡蛎(Crassostrea gigas)基因内的甲基化模式,证实甲基化是太平洋牡蛎基因组的一种普遍特征,而且不同功能基因的甲基化水平存在显著差异。虽然,目前很多学者已经在贝类方面开展了表观遗传学方面的研究,但总体而言,研究起步较晚,资料匮乏。本研究运用MSAP技术分析了虾夷扇贝选育群体和对照群体的DNA甲基化水平和模式,初步探讨了选育对DNA甲基化的影响,为从表观遗传学在贝类选育方面的研究积累了资料,为结合经典遗传学分析评价选育效果提供了技术手段。
1 材料与方法 1.1 材料来源虾夷扇贝选育群体为本课题组通过累代选育获得的虾夷扇贝新品系-“玉贝”,对照群体是未经选育的群体,两群体的繁育及养殖条件相同。分别取贝龄为15个月的两组虾夷扇贝各15只,取闭壳肌置于-80°C中保存备用。
1.2 基因组DNA的提取DNA提取采用传统的酚-氯仿法进行,取闭壳肌约100 mg置于475 μL裂解液(10 mmol/L Tris-Cl,10 mmol/L EDTA)中剪碎,加入40 μL 10%的SDS和5 μL 20 mg/mL 的蛋白酶K,在55 °C中消化3 h至澄清,用25︰24︰1的酚-氯仿-异戊醇混合液抽提2次,然后用2倍体积预冷的无水乙醇和1/25体积的5 mol/L的NaCl在-20 °C中沉淀30 min后,离心,70%乙醇洗涤两次后在生物安全柜中风干,无菌蒸馏水溶解沉淀。通过1.5%琼脂糖电泳检测DNA是否降解,检测OD260/280以确定DNA的纯度,测定浓度后用三蒸水调整DNA浓度至300 ng/μL,存于-20 °C中备用。
1.3 MSAP分析MSAP操作按照吴彪等(2012)的方法稍作修改,对实验条件进行优化以建立适用于虾夷扇贝的MSAP分析方法。选用EcoRⅠ、MspⅠ和HpaⅡ三种酶进行酶切反应,每个样品DNA分为两种酶切组合,即: EcoRⅠ+ HpaⅡ和EcoRⅠ+ MspⅠ。酶切反应体系为: 300 ng DNA,5 U EcoRⅠ(TaKaRa),5 U HpaⅡ/ MspⅠ(TaKaRa),2 μL 10 × Buffer(TaKaRa),加灭菌ddH2O至20 μL。酶切体系经过37 °C水浴4 h、65 °C变性10 min后储存于-20 °C中。之后进行连接反应,体系为: 5 μL酶切产物,50 pmol HM接头,10 pmol E接头,5 U T4 DNA Ligase(TaKaRa),4 μL 5 × T4 DNA Ligase Buffer,补水至20 μL,16 °C连接过夜,产物稀释10倍后用于预扩增。
预扩增反应体系为20 μL : 2 μL稀释10倍后的连接产物,20 pmol预扩增引物 E0和HM0,0.5 U Taq酶,2 μL 10 × Taq buffer(含Mg2+),1.6 μL dNTPs(各2.5mmol/L),补水至20 μL。PCR扩增反应条件为: 94 °C预变性 3 min; 94 °C变性 30 s,56 °C退火 1 min,72 °C延伸 1 min,20 个循环; 最后72 °C延伸10 min。预扩增产物稀释20 倍后作为选择扩增的模板。
选扩体系为20 μL : 3 μL稀释20倍后的预扩增产物,20 pmol HMn和En选扩引物,其它反应体系中的组分与预扩反应体系相同。PCR反应程序为: 94 °C预变性 3 min; 第一轮扩增为13个循环的touchdown反应,包括94 °C变性 30 s,65 °C退火 1 min(每循环降低0.7 °C),72 °C延伸1 min; 第二轮扩增为25个循环,包括94 °C变性 30 s,56 °C退火 1 min,72 °C延伸1 min,最后72 °C延伸10 min。本实验所用的引物信息见表 1。
| 接头及引物 | EcoRⅠ | MspⅠ/ HpaⅡ | ||
| 接头(5′—3′) | EA1: CTCGTAGACTGCGTACC | HMA1: GATCATGAGTCCTGCT | ||
| 预扩引物(5′—3′) | EA2: AATTGGTACGCAGTCTAC | HMA2: CGA GCA GGA CTC AGA A | ||
| E0: GACTGCGTACCAATTC | HM0: ATCATGAGTCCTGCTCGGG | |||
| 选扩引物(5′—3′) | E1: GACTGCGTACCAATTCACA | HM1: ATCATGAGTCCTGCTCGGGCTGA | ||
| E2: GACTGCGTACCAATTCAGT | HM2: ATCATGAGTCCTGCTCGGGCTGT | |||
| E3: GACTGCGTACCAATTCAAC | HM3: ATCATGAGTCCTGCTCGGGCTAT | |||
| E4: GACTGCGTACCAATTCGTC | HM4: ATCATGAGTCCTGCTCGGGCTAC | |||
| E5: GACTGCGTACCAATTCGCT | HM5: ATCATGAGTCCTGCTCGGGCTCA | |||
| HM6: ATCATGAGTCCTGCTCGGGCTCT | ||||
| HM7: ATCATGAGTCCTGCTCGGGCTTC | ||||
| HM8: ATCATGAGTCCTGCTCGGGCTTA | ||||
PCR产物的电泳检测: 扩增产物与上样缓冲液2 : 1混合后,94 °C变性10 min后在冰浴环境中冷却5 min,6%聚丙烯酰胺凝胶电泳60 W恒功率电泳2.5 h,银染法染色。
1.4 MSAP数据统计及分析通过优化MSAP实验条件,从40对引物组合中筛选多态性好、重复性高、条带清晰的引物组合进行实验分析。每个DNA样品分别进行了两个组合的酶切,所以有两组扩增产物,即EcoRⅠ/HpaⅡ组和EcoR I/Msp I组,统计PAGE电泳图每个泳道中大小为50—1500 bp的条带,同一位点上有带记为1,无带记为0。计算两个群体的去甲基化率、全甲基化率、半甲基化率和总甲基化率,以及甲基化多态性比例,甲基化多态性分析参照杜盈等(2013)的方法进行分析。
2 结果 2.1 引物筛选两个群体虾夷扇贝混合基因组DNA通过酶切、连接和预扩增之后,运用5个E引物和8个HM引物的40个组合进行选择性扩增,在6%的聚丙烯酰胺凝胶上进行电泳、银染显色、成像。从40对引物组合中共筛选出了E1HM4、E1HM5、E2HM7、E3HM3、E3HM5、E3HM8、E4HM5、E5HM4、E5HM7、E5HM8、E5HM2等11对引物组合,这些引物组合能够稳定地扩增出清晰、多态的谱带,运用这11对引物对两群体进行DNA甲基化分析。图 1为部分引物电泳图。
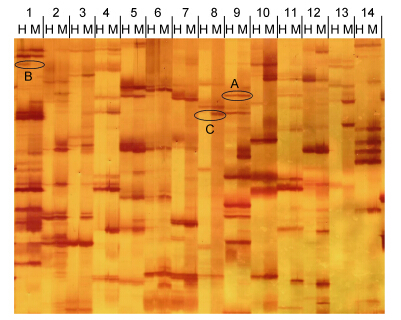 |
| 图 1 不同引物组合在虾夷扇贝“玉贝”基因组 DNA 中的MSAP 图谱 Fig. 1 An example of DNA methylation profiles in P. yessoensis amplified using different primers pairs 1—14: HMn和En的不同组合形式; H: EcoR I/Hpa II酶切产物,M: EcoRⅠ/MspⅠ酶切产物; A,B,C分别代表不同的甲基化模式,A: 去甲基化位点,B: 半甲基化位点,C: 全甲基化位点 |
在MSAP的丙烯酰胺凝胶电泳图谱上,每个DNA样品对应两个泳道,是EcoRⅠ分别与HpaⅡ、MspⅠ组合酶切处理的样品,将EcoRⅠ/HpaⅡ定义为H组,EcoR I/MspⅠ为M组。内切酶MspⅠ和Hpa Ⅱ的酶切识别位点都是CCGG,但由于它们对胞嘧啶甲基化敏感性不同,所以会产生不同的酶切效果。HpaⅡ对双链内部C碱基的全甲基化敏感,不能切开双链内部C碱基全甲基化的位点,但对任意一条单链外部C碱基甲基化不敏感,所以可以有效切开外部C碱基的半甲基化位点(HMeCCG); 而MspⅠ对甲基化的敏感性相反,能够切开内部甲基化位点(HMeCG或MeCG); 两个酶都不能有效切开外部C碱基的全甲基化(MeCCG)或内外C碱基的全甲基化(MeCMeCG)、全半甲基化(HMeCHMeCG)的位点。所以,根据扩增条带的有无,MSAP凝胶电泳图谱上每份DNA样品的同一个位点的两个泳道上会有以下三种模式:(1)两个泳道中都有条带(1,1),表示该位点去甲基化;(2)两泳道中H组中有带,M组中没有条带(1,0),表示该位点半甲基化;(3)两泳道中H组中没有条带,M组中有带(0,1),表示该位点全甲基化。运用筛选出的11对引物对“玉贝”和对照群体进行了扩增,结果表明,两群体中均存在上述的三种甲基化类型。两群体扇贝的扩增条带中都以Ⅰ型为主,均占总扩增条带的76%以上。各种甲基化类型如图 1所示。
2.3 DNA甲基化水平差异对MSAP电泳图谱上的条带进行统计,扩增条带类型及甲基化水平结果如表 2所示。结果表明,虾夷扇贝“玉贝”和对照群体的总扩增条带数分别为1432和1442条,两群体扇贝的扩增条带中都以Ⅰ型,即未甲基化为主,“玉贝”为1117条,对照群体为1101条; 甲基化条带总数分别为308和344,总甲基化率分别为21.5%和23.65%,“玉贝”群体甲基化扩增位点和总甲基化率都低于对照组; 两群体甲基化类型也存在差别,“玉贝”全甲基化位点和半甲基化位点数目和比例相近,分别为152(10.68%)和154(10.82%),对照群体中全甲基化位点明显高于半甲基化位点,为236(16.3%)和105(7.28%)。两群体扇贝的胞嘧啶甲基化模式存在差异,相对于对照群体,“玉贝”全甲基化率降低,半甲基化率升高。
| 扇贝 | 总扩增位点 | 非甲基化位点 | 甲基化位点 | ||
| 总甲基化位点 | 全甲基化位点 | 半甲基化位点 | |||
| 虾夷扇贝“玉贝” | 1432 | 1117(78.5%) | 306(21.5%) | 152(10.68%) | 154(10.82%) |
| 对照虾夷扇贝 | 1442 | 1101(76.35%) | 341(23.65%) | 236(16.37%) | 105(7.28%) |
每组15个样品在同一个扩增位点的扩增条带组成一个条带链,一个条带链中会出现不同类型甲基化模式的组合形式,由三种及以上不同模式组成的条带链较少,并且分析复杂,所以本研究中只统计了由两种甲基化模式组成的条带链,组合形式如表 3所示。可以看出,6种条带链类型中Class A、Class D、Class F为单一性位点,Class B、Class C、Class E为多态性位点。
| 条带类型 | Ⅰ型(11) | Ⅱ型(10) | Ⅲ型(01) |
| Ⅰ型 TypeⅠ(11) | Class A(11 11) | Class B(11 10) | Class C(11 01) |
| Ⅱ型 TypeⅡ(10) | Class B(10 11) | Class D(10 10) | Class E(10 01) |
| Ⅲ型 TypeⅢ(01) | Class C(01 11) | Class E(01 10) | Class F(01 01) |
6种类型的条带链统计结果见图 2和表 4。结果表明,“玉贝”和对照组的两个虾夷扇贝群体中,Ⅰ型及Ⅰ型条带组成的A类条带链占主导,数目最多,都占65%以上,其它两个单态性的D、F类条带链相对较少,对照群体中的D类仅为1.042%; 具有甲基化多态性的B、C、E类中,B类最多,C类次之,E类最少; 两个群体相比,每种类型的条带链数目和比例相近,“玉贝”群体的甲基化多态性条带所占的比例高于对照群体,分别为31%和29.167%,尤其是B类型条带链,“玉贝”群体明显高于对照群体。
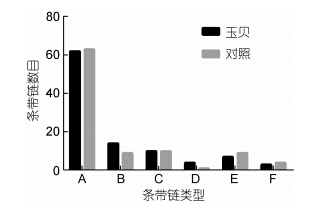 |
| 图 2 “玉贝”与对照群体中6种甲基化条带链的比例 Fig. 2 The percentage of six methylation chains in “Yubei” and the control P. yessoensis salinities |
| 扇贝 | 不同条带链类型的比例(%) | |||||
| Class A | Class B | Class C | Class D | Class E | Class F | |
| “玉贝” | 62 | 14 | 10 | 4 | 7 | 3 |
| 对照 | 65.625 | 9.375 | 10.417 | 1.047 | 9.375 | 4.167 |
表观遗传学是与经典遗传学相对应的概念,指基于非基因序列改变所致基因表达水平变化,如DNA甲基化和染色质构象变化等。表观遗传学概念的提出,使很多孟德尔核内遗传规律不能解释的现象得到了非常合理而完美的解释。近年来,DNA甲基化作为表观遗传学的重要研究内容,已经成为分子生物学研究热点,在涉及肿瘤学、心血管系统疾病学、神经学等医学领域和林业、畜牧与动物医学等生物学领域应用广泛,水产领域研究相对较少。但DNA甲基化在动植物育种中,尤其是抗逆性状的遗传改良方面具有巨大的应用潜力。这是因为,DNA携带了物种的基本遗传信息,决定着物种的特性和生物学行为,虽然能够通过重组获得新基因来抵抗、忍受逆境,但是这种改变远远滞后于环境变化,而在逆境胁迫下,甲基基团能够迅速、可逆地对DNA进行修饰,避免了不必要的基因重组(Boyko et al 2008)。而且,某些DNA甲基化在多种表观遗传机制中具有协调的中心作用,能够向后代稳定传递(Li et al, 2014)。MSAP能够在无基因组信息的情况下分析基因组DNA甲基化水平和模式,该方法简便、高效,而且成本较低,已经成功在很多动植物的研究中得到应用。DNA甲基化的相关研究在水产动物中起步晚,但发展较快,已经在尼罗罗非鱼(Oreochromis niloticus)(朱华平等,2013)、鲫鱼(Carassius auratus)(周新文等,2001)、泥鳅(Misgurnus anguillicaudatus)(王丙莲等,2006)、中国明对虾(Fenneropenaeus chinensis)(杜盈等,2013)、刺参(Apostichopus japonicus)(郭婷婷等,2013)、太平洋牡蛎(Gavery et al, 2010)以及几种扇贝(于涛等,2010; 吴彪等,2012; 吕佳等,2013; Sun et al,2014)等多个物种中有报道,研究主要集中在初步了解研究对象的甲基化水平以及外界环境条件对甲基化水平的影响。本研究结果表明,对照群体虾夷扇贝基因组DNA的总甲基化率为23.65%,“玉贝”群体总甲基化率低于对照组,为21.5%,下降了2.15%,中国对虾(杜盈等,2013)、罗非鱼(朱华平等,2013)的选育群体基因组DNA也有去甲基化趋势,本研究结果与其相一致。本研究中两群体的总甲基化率与之前报道的栉孔扇贝(20.94%—32.08%)(于涛等,2010; 吕佳等,2013; Sun et al,2014)、海参(28%—35.77%)(郭婷婷等,2013)、泥鳅(10.98%—16.72%)(王丙莲等,2006)等多种水产动物相近,但与其它已报道的哺乳动物以及鸟类(唐韶青等,2006)(40%—50%)相比,本研究中的虾夷扇贝总甲基化水平偏低,而且比其他学者报道的其它虾夷扇贝群体(于涛等,2010; 吕佳等,2013)(32.79%—34.97%)也偏低。这些研究数据表明,不同物种之间基因组DNA甲基化水平的高低不存在明显的规律,即便是同一物种,不同学者研究得出的研究结果也不尽相同,不过有学者通过比较发现甲基化水平在从低等向高等的物种过渡中有一定的上升趋势(郭婷婷等,2013)。这种差异可能是由多种原因导致的,如条带统计的范围不同,研究对象发育时期和生存环境不同等都能够造成基因组DNA甲基化水平的改变。本研究中的对照群体是课题组选育研究的对照组,其繁育过程、培育环境等严格与“玉贝”的操作流程一致,其甲基化水平的差异能够体现选育的作用效果。这说明选育不仅改变了DNA基化水平,还对甲基化模式产生了影响。与对照群体相比,“玉贝”全甲基化率降低5.69%,但半甲基化率升高了3.54%,而且多态性也有差别。这可能是选育压力影响了基因组原来的甲基化模式,启动并产生了应对胁迫的适应机制,使某些功能基因的转录表达等生物过程开启,而且这种机制在选育过程中传递给了后代。 表观遗传学是与经典遗传学相对应的概念,指基于非基因序列改变所致基因表达水平变化,如DNA甲基化和染色质构象变化等。表观遗传学概念的提出,使很多孟德尔核内遗传规律不能解释的现象得到了非常合理而完美的解释。近年来,DNA甲基化作为表观遗传学的重要研究内容,已经成为分子生物学研究热点,在涉及肿瘤学、心血管系统疾病学、神经学等医学领域和林业、畜牧与动物医学等生物学领域应用广泛,水产领域研究相对较少。但DNA甲基化在动植物育种中,尤其是抗逆性状的遗传改良方面具有巨大的应用潜力。这是因为,DNA携带了物种的基本遗传信息,决定着物种的特性和生物学行为,虽然能够通过重组获得新基因来抵抗、忍受逆境,但是这种改变远远滞后于环境变化,而在逆境胁迫下,甲基基团能够迅速、可逆地对DNA进行修饰,避免了不必要的基因重组(Boyko et al 2008Boyko et al 2008,)。而且,某些DNA甲基化在多种表观遗传机制中具有协调的中心作用,能够向后代稳定传递(Li et al, 2014)。MSAP能够在无基因组信息的情况下分析基因组DNA甲基化水平和模式,该方法简便、高效,而且成本较低,已经成功在很多动植物的研究中得到应用。DNA甲基化的相关研究在水产动物中起步晚,但发展较快,已经在尼罗罗非鱼(Oreochromis niloticus)(朱华平等,2013)、鲫鱼(Carassius auratus)(周新文等,2001)、泥鳅(Misgurnus anguillicaudatus)(王丙莲等,2006)、中国明对虾(Fenneropenaeus chinensis)(杜盈等,2013)、刺参(Apostichopus japonicus)(郭婷婷等,2013)、太平洋牡蛎(Gavery et al, 2010)以及几种扇贝(于涛等,2010; 吴彪等,2012; 吕佳等,2013; Sun et al,2014)等多个物种中有报道,研究主要集中在初步了解研究对象的甲基化水平以及外界环境条件对甲基化水平的影响。本研究结果表明,对照群体虾夷扇贝基因组DNA的总甲基化率为23.65%,“玉贝”群体总甲基化率低于对照组,为21.5%,下降了2.15%,中国对虾(杜盈等,2013)、罗非鱼(朱华平等,2013)的选育群体基因组DNA也有去甲基化趋势,本研究结果与其相一致。本研究中两群体的总甲基化率与之前报道的栉孔扇贝(20.94%—32.08%)(于涛等,2010; 吕佳等,2013; Sun et al,2014)、海参(28%—35.77%)(郭婷婷等,2013)、泥鳅(10.98%—16.72%)(王丙莲等,2006)等多种水产动物相近,但与其它已报道的哺乳动物以及鸟类(唐韶青等,2006)(40%—50%)相比,本研究中的虾夷扇贝总甲基化水平偏低,而且比其他学者报道的其它虾夷扇贝群体(于涛等,2010; 吕佳等,2013)(32.79%—34.97%)也偏低。这些研究数据表明,不同物种之间基因组DNA甲基化水平的高低不存在明显的规律,即便是同一物种,不同学者研究得出的研究结果也不尽相同,不过有学者通过比较发现甲基化水平在从低等向高等的物种过渡中有一定的上升趋势(郭婷婷等,2013)。这种差异可能是由多种原因导致的,如条带统计的范围不同,研究对象发育时期和生存环境不同等都能够造成基因组DNA甲基化水平的改变。本研究中的对照群体是课题组选育研究的对照组,其繁育过程、培育环境等严格与“玉贝”的操作流程一致,其甲基化水平的差异能够体现选育的作用效果。这说明选育不仅改变了DNA基化水平,还对甲基化模式产生了影响。与对照群体相比,“玉贝”全甲基化率降低5.69%,但半甲基化率升高了3.54%,而且多态性也有差别。这可能是选育压力影响了基因组原来的甲基化模式,启动并产生了应对胁迫的适应机制,使某些功能基因的转录表达等生物过程开启,而且这种机制在选育过程中传递给了后代。
DNA甲基化是基因表达调控的重要方式之一,面对环境的变化,生物体需要不断进行生物学调节来适应环境的变化。很多研究已经证明,外界压力的胁迫能够通过改变DNA甲基化从而调控基因的表达,改变生物体表型以适应新环境(彭海等,2009; Cao et al,2013)。如Choi等(2007)推测烟草(Nicotiana tabacum)基因组上大约10%的位点在非生物胁迫下发生低甲基化; 金属离子胁迫能够改变鱼类的DNA甲基化水平(周新文等,2001; 王丙莲等,2006); 太平洋牡蛎的相关研究结果还表明,贝类DNA甲基化能够调控功能基因来适应环境压力的胁迫(Gavery et al,2010)。选育是对胁迫不断强化并使适应这种胁迫条件的性状得以固定和遗传。本研究中“玉贝”是通过累代选育而得到的虾夷扇贝新品系,具有生长快速、耐高温能力强的特征,“玉贝”总甲基化率比普通群体降低了2.15%,而且甲基化模式变化较大,这种变化可能与优势性状关系密切。学者在研究中国对虾时发现,选育群体肌肉、鳃和血液三个组织的甲基化水平比对照群体低,耐寒罗非鱼品系甲基化水平相对于对照组都有所下降(杜盈等,2013; 朱华平等,2013)。这些数据都表明,基因组DNA甲基化水平与选育群体优良性状存在一定的相关性,选育不仅使原有优良性状的基因得以固定,还有可能增强了一些抗逆基因的表达。研究某些基因调控区的DNA甲基化与基因转录、表达的相关性已经在疾病诊断治疗等一些领域开展,但是目前,水产领域中的相关研究还基本没有涉及。在水产动物育种领域,从DNA甲基化研究入手,筛查与性状相关的特异性片段,进而克隆获得功能基因并研究基因的调控机制,能够为阐述DNA甲基化作用机制提供数据,能够为育种应用提供理论参考,有待进一步深入研究。
| 于 涛, 杨爱国, 吴 彪等, 2010.栉孔扇贝、虾夷扇贝及其杂交子代的MSAP分析.水产学报, 34(9): 1335-1342 |
| 于佐安, 李文姬, 张 明等, 2011.大连市长海县虾夷扇贝养殖海区浮游病毒的丰度.水产学报, 35(6): 911-917 |
| 万亚琴, 2008.DNA甲基化与肉牛杂种优势关系的初步研究.重庆: 西南大学硕士学位论文, 65 |
| 王丙莲, 张迎梅, 谭玉凤等, 2006.镉铅对泥鳅DNA甲基化水平的影响.毒理学杂志, 20(2): 78-80 |
| 仪治本, 孙 毅, 牛天堂等, 2005.高粱基因组DNA胞嘧啶甲基化在杂交种和亲本间差异研究.作物学报, 31(9): 1138-1143 |
| 吕 佳, 侯 睿, 李 宁等, 2013.应用MSAP技术研究扇贝全基因组DNA甲基化水平.中国海洋大学学报, 43(10): 48-53 |
| 朱华平, 卢迈新, 黄樟翰等, 2013.低温对罗非鱼基因组DNA甲基化的影响.水产学报, 37(10): 1460-1467 |
| 刘卫东, 鲍相渤, 宋文涛等, 2009.虾夷扇贝遗传连锁图谱的初步构建.遗传, 31(6): 629-637 |
| 杜 盈, 何玉英, 李 健等, 2013.野生和"黄海1号"中国明对虾不同组织基因组DNA的MSAP分析.中国水产科学, 20(3): 536-543 |
| 杨金兰, 柳李旺, 龚义勤等, 2007.镉胁迫下萝卜基因组DNA甲基化敏感扩增多态性分析.植物生理与分子生物学学报, 33(3): 219-226 |
| 吴 彪, 杨爱国, 刘志鸿等, 2012.两种扇贝杂交和自交家系早期生长及甲基化的比较分析.海洋科学, 36(2): 1-6 |
| 吴 彪, 迟长凤, 杨爱国等, 2013.虾夷扇贝C型凝集素母源传递与抑菌作用的初步研究.水产学报, 37(5): 777-783 |
| 张明明, 赵 文, 2008.我国虾夷扇贝死亡原因的探讨及控制对策.中国水产, (2): 65-66, 74 |
| 陈培利, 童坦君, 张宗玉, 2000.DNA甲基化对基因表达的影响及其在衰老过程中的表现.国外医学分子生物学分册, 22(3): 155-158 |
| 周新文, 朱国念, Mwalilino J等, 2001.Cu、Zn、Pb、Cd及其混合重金属离子对鲫鱼(Carassius auratus) DNA甲基化水平的影响, 中国环境科学, 21(6): 549-552 |
| 郭 磊, 李 慧, 韩之明, 2010.DNA甲基化和组蛋白修饰在克隆动物发育过程中的作用.遗传, 32(8): 762-768 |
| 郭婷婷, 孙国华, 杨建敏等, 2013.刺参(Apostichopus japonicus)不同组织基因组甲基化状态MSAP分析.海洋与湖沼, 44(1): 77-82 |
| 唐韶青, 张 沅, 徐 青等, 2006.不同动物部分组织基因组甲基化程度的差异分析.农业生物技术学报, 14(4): 507-510 |
| 常亚青, 陈晓霞, 丁 君等, 2007.虾夷扇贝(Patinopecten yessoensis)5个群体的遗传多样性.生态学报, 27(3): 1145-1152 |
| 彭 海, 张 静, 2009.胁迫与植物DNA甲基化: 育种中的潜在应用与挑战.自然科学进展, 19(3): 248-256 |
| 程 鹏, 杨爱国, 吴 彪等, 2011.微卫星标记在不同壳色虾夷扇贝家系亲权鉴定的适用性.水生生物学报, 35(5): 768-775 |
| 程 鹏, 杨爱国, 周丽青等, 2010.不同壳色虾夷扇贝家系F1幼虫生长及遗传结构的比较分析.中国水产科学, 17(5): 960-968 |
| 潘雅姣, 傅彬英, 王 迪等, 2009.水稻干旱胁迫诱导DNA甲基化时空变化特征分析.中国农业科学, 42(9): 3009-3018 |
| Bird A, 1992.The essentials of DNA methylation.Cell, 70(1): 5-8 |
| Boyko A, Kovalchuk I, 2008.Epigenetic control of plant stress response.Environmental and Molecular Mutagenesis, 49(1): 61-72 |
| Cao J X, Zhang H P, Du L X, 2013.Influence of environmental factors on DNA methylation.Hereditas, 35(7): 839-846 |
| Choi C-S, Sano H, 2007.Abiotic-stress induces demethylation and transcriptional activation of a gene encoding a glycerophosphodiesterase-like protein in tobacco plants.Molecular Genetics and Genomics, 277(5): 589-600 |
| Elango N, Yi S V, 2008.DNA methylation and structural and functional bimodality of vertebrate promoters.Molecular Biology and Evolution, 25(8): 1602-1608 |
| Gavery M R, Roberts S B, 2010.DNA methylation patterns provide insight into epigenetic regulation in the Pacific oyster (Crassostrea gigas).BMC Genomics, 11: 483 |
| Hu Y, Xu H, Li Z et al, 2013.Comparison of the genome-wide DNA methylation profiles between fast-growing and slow-growing broilers.PLoS One, 8(2): e56411 |
| Li Q, Eichten S R, Hermanson P J et al, 2014.Inheritance patterns and stability of DNA methylation variation in maize near-isogenic lines.Genetics, 196(3): 667-676 |
| Li Q, Xu K F, Yu R H, 2007.Genetic variation in Chinese hatchery populations of the Japanese scallop (Patinopecten yessoensis) inferred from microsatellite data.Aquaculture, 269(1-4): 211-219 |
| Powell C, Grant, A R, Cornblath E et al, 2013.Analysis of DNA methylation reveals a partial reprogramming of the Müller glia genome during retina regeneration.Proceedings of the National Academy of Science of the United States of America, 110(49): 19814-19819 |
| Suarez-Alvarez B, Rodriguez R M, Fraga M F et al, 2012.DNA methylation: a promising landscape for immune system-related diseases.Trends in Genetics, 28(10): 506-514 |
| Sun Y, Hou R, Fu X et al, 2014.Genome-wide analysis of DNA methylation in five tissues of zhikong scallop, Chlamys farreri.PLoS One, 9(1): e86232 |
 2015, Vol. 46
2015, Vol. 46


