中国海洋湖沼学会主办。
文章信息
- 刘青, 刘皓, 吴旭干, 何杰, 董鹏生, 王幼鹏, 成永旭. 2015.
- LIU Qing, LIU Hao, WU Xu-Gan, HE Jie, DONG Peng-Sheng, WANG You-Peng, CHENG Yong-Xu. 2015.
- 长江、黄河和辽河水系中华绒螯蟹野生和养殖群体遗传变异的微卫星分析
- GENETIC VARIATION OF WILD AND CULTURED POPULATIONS OF CHINESE MITTEN CRAB ERIOCHEIR SINENSIS FROM THE YANGTZE, HUANGHE, AND LIAOHE RIVER BASINS USING MICROSATELLITE MARKER
- 海洋与湖沼, 46(4): 958-968
- Oceanologia et Limnologia Sinica, 46(4): 958-968.
- http://dx.doi.org/10.11693/hyhz20150400129
-
文章历史
- 收稿日期:2015-04-18
- 改回日期:2015-05-19
2. 山西农业大学动物科技学院 太谷 030801;
3. 上海海洋大学 上海市教委水产动物遗传育种协同创新中心 上海 201306;
4. 江苏省宿迁旭邦水产科技有限公司 泗洪 223900
2. College of Animal Science and Veterinary Medicine, Shanxi Agricultural University, Taigu 030801, China;
3. Collaborative Innovation Center of Aquatic Animal Breeding Center Certificated by Shanghai Municipal Education Commission, Shanghai Ocean University, Shanghai 201306, China;
4. Suqian Xubang Fisheries Science and Technology Co. Ltd., Sihong 223900, China
中华绒螯蟹(Eriocheir sinensis)俗称河蟹(以下简称河蟹),是中国重要的淡水养殖种类,2012年全国养殖总产量达714400 t(王武等,2013)。我国河蟹天然群体曾广泛分布于辽河、黄河、长江、瓯江和闽江等水系,野生资源丰富(许加武等,1997; Sui et al,2009)。但是,20世纪70年代以来,由于过渡捕捞、入海和沿江河流的水闸阻挡河蟹洄游通道及环境污染等原因,各水系河蟹野生资源急剧下降,成蟹捕捞产量逐年下降,难以满足市场需求(Cheng et al,2008)。另一方面,随着人们生活水平的提高,河蟹的消费量不断增加,这种状况极大促进了河蟹增养殖业的迅速发展(Sui et al,2011)。20世纪80年代河蟹人工育苗技术取得突破以后,其人工养殖在我国大部分地区得以迅速发展,目前养殖较集中的区域为长江流域、黄河流域和辽河流域,其中长江流域养殖规模、总产量和成蟹平均规格均占首位(崔江华,2010; 王武等,2013; 赵娜,2014)。由于不同水系河蟹的养殖性能和蟹种价格相差较大(李勇等,2001; 李晨虹等,2002),通常认为长江水系河蟹养殖性能最佳,因此20世纪90年代其它水系的河蟹蟹种被引入长江流域满足蟹种需求的缺口; 另一方面,长江水系蟹种也被引入黄河和辽河流域进行池塘养殖希望获得更好的养殖效果,这种水系间的盲目引种及养殖导致河蟹种质混杂、养殖性能退化(王武等,2007)。此外,河蟹人工繁育过程中,许多育苗场为了降低亲本费用,通常采用小规格池塘养殖亲本进行育苗,经过多代人工繁育和养殖,致使河蟹养殖群体的种质退化,养殖性能下降(Sui et al,2011; 王武等,2013)。因此,系统研究我国现有主要野生和养殖群体河蟹的遗传多样性、遗传分化和种群结构等,对其种质资源保护和开发利用具有重要的现实意义(朱清顺等,2008; 张世勇等,2013)。
近10年来,由于河蟹养殖产量的增加和主要水系禁渔期的执行,使得野生河蟹的捕捞强度有所降低,加上各水系开展河蟹增殖放流和水域生态环境保护等,长江、黄河和辽河等水系河蟹野生资源在一定程度上得以恢复(周刚等,2003; 施德龙,2006; 刘凯等,2013),这为各水系野生河蟹的种质资源评价和优异种质发掘利用提供了物质基础。先前有研究报道了长江和辽河水系野生河蟹的遗传特性和种群结构等(李思发等,1999; 李海燕等,2003; Sui et al,2009; 龚疏影等,2009),但是这些研究样品多采集于2000年前后,无法准确反应当前各水系河蟹的种质资源的现状。另外,近10多年来各水系野生河蟹均经历了野生群体数量下降、水系间盲目引种、养殖群体逃逸和增殖放流等人工干扰,这可能会对三水系河蟹野生群体的遗传多样性和遗传结构造成一定的影响,因此需要对现存三水系野生群体的遗传特性进行科学评估。先前有关河蟹遗传特征的研究报道主要是针对各水系野生群体(Herborg et al,2007; Chang et al,2008; Sui et al,2009),尚缺乏对我国三个主产水系现存野生和养殖群体遗传特性的比较研究,这非常不利于我国河蟹种质资源保护和开发利用工作。
DNA微卫星(Simple Sequence Repeat,SSR)多态性来源于其核心序列重复次数的变化,具有信息含量丰富、多态性高、个体特异性强、共显性遗传等优点,比RFLP、RAPD、AFLP等分子标记更能揭示动物的遗传特性,因此在种质资源评价和遗传育种中得到了广泛的应用(Baker,1994; Bentzen et al,2001)。目前,SSR已经被应用于河蟹的种群遗传和遗传育种中(Herborg et al,2007; Chang et al,2008; Sui et al,2009; 李晓晖等,2010),但是以往研究采用的SSR标记数量较少,且没有同时对我国现有主要野生和养殖群体河蟹的遗传特征进行系统评估。鉴于此,本研究筛选了29对多态性较高的SSR标记,比较了长江、黄河和辽河水系野生及养殖群体的遗传多样性、遗传分化、瓶颈效应和遗传结构,以评估我国主要水系河蟹野生和养殖和群体的遗传特性,为进一步开展河蟹野生种质资源保护、优异种质挖掘利用和遗传育种等提供基础资料。
1 材料与方法 1.1 样本采集长江野生群体(YW)、黄河野生群体(HW)和辽河野生群体(LW)河蟹分别于2013年10—12月份采集于上海崇明县(121.18°E,31.76°N),山东东营市利津县(118.52°E,37.61°N),辽宁盘锦市大洼县(122.70°E,40.70°N); 长江养殖群体(YP)、黄河养殖群体(HP)和辽河养殖群体(LP)河蟹分别采集于上海市崇明县(121.26°E,31.74°N),山东东营市垦利县(118.74°E,37.59°N),辽宁盘锦市大洼县(122.08°E,41.01°N)的河蟹养殖场(图 1)。每个群体分别随机采集60只成蟹,体重为96—208g。分别取其附肢肌肉,用95%乙醇固定后在-20°C冰箱中保存备用。
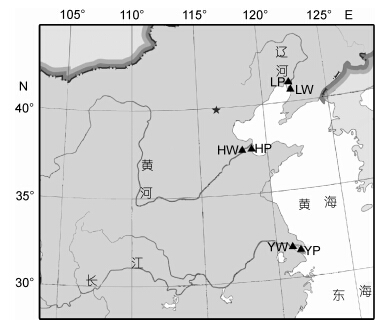 |
| 图 1 河蟹6群体的采样地点 Fig. 1Sampling location of six populationsof E. sinensis |
采用改进的酚-氯仿法提取河蟹基因组DNA(刘皓等,2015),采用琼脂糖凝胶电泳检测DNA的完整性,采用微量紫外分光光度计(型号: Q5000,美国Quawell公司生产)检测DNA质量和浓度,ddH2O稀释至50ng/µL待PCR用。
1.3 引物筛选和PCR扩增从已报道的河蟹SSR引物中选择29对多态性高、扩增效果好的引物用于本研究,其中Esin18,Esin36,Esin42,Esin55, Esin67, Esin87引物序列参考Hänfling等(2003); HLJEsd02,HLJEsc11,HLJEsc20,HLJEsb25,HLJEsc34,HLJEsa42,HLJEsd52, HLJEsc56, HLJEsc65,HLJEsa67,HLJEsa69,HLJEsa76,HLJEsc86, HLJEsb88引物序列参考Chang等(2006); JPX-3,JPX-10,JPX-13引物序列参考Cheng等(2010); Q67,Q703,Q726,Q732,Q840,Q934引物序列参考熊良伟(2014)。所有正向(5'端)引物采用荧光标记(FAM或HEX),反向(3'端)为普通引物。PCR反应体系总体积为10µL,包括50ng DNA模板、1µL10×PCR buffer、200µmol/L dNTP、各0.5µmol/L正反向引物和0.5U Taq聚合酶,然后用ddH2O补充至10µL,所需试剂均购于Takara公司)。采用降落式PCR反应程序进行SSR标记扩增,以提高荧光引物的结合效率,减少杂带(Sui et al,2009),具体PCR程序为: 94°C预变性3min; 93°C变性1min,(退火温度+6°C)退火30s,72°C延伸1min,2个循环; 93°C变性1min,(退火温度+4°C)退火30s,72°C延伸1min,2个循环; 89°C变性1min,(退火温度+2°C)退火30s,72°C延伸1min,5个循环; 89°C变性1min,(正常退火温度)退火30s,72°C延伸1min,20—25个循环; 72°C延伸10min。PCR产物于-20°C保存待用。
根据不同SSR位点的PCR产物片段长度范围的差别,挑选2—3个不同荧光标记且长度相差较大的SSR位点PCR产物,将其混合后利用后全自动DNA测序仪进行分析(型号: ABI-3730XL,美国ABI公司),采用GeneMaper-3.5软件扫描峰图后读取目的片段的长度,将数据导入Microsoft excel中整理以备分析(马雪霞等,2007; 刘皓等,2015)。
1.4 数据分析根据各位点基因片段的长度和,确定每个样品各SSR位点的基因型。利用 POPGEN 3.2软件计算各位点的观测等位基因数(Na)、有效等位基因数(Ne)、香农信息指数(I)、观测杂合度(Ho)、期望杂合度(He)、固定指数(FIS)(Nei,1978); 采用PIC_CALC 0.6软件计算多态信息含量(PIC)(王婷等,2014),按照参考文献Botstein等(1980)的标准,当某位点PIC>0.5时,该位点为高多态性位点; 利用 ARLEQUIN 3.5软件的马尔科夫链(MarkovChain)方法对各位点进行Hardy-Weinberg平衡检验(Exoffier et al,2010)。
利用POPGEN 32软件计算群体间的Nei’s遗传距离(Nei,1978),基于此距离采用MEGA 5.0软件作UPGMA系统树(Tamura et al,2011); 用ARLEQUIN 3.5软件计算群体遗传分化的F-统计量(F-statistics,FST),FST<0.05为轻微分化(Balloux et al,2002)。分子方差分析(AMOVA)分析变异来源(Exoffier et al,2010)。
基于无限等位基因模型(Infinite Allele Model,IAM)、双相突变模型(Two-phased Model of Mutation,TPM)及逐步突变模型(Step-wise Mutation Model,SMM),用BOTTLENECK 3.4软件进行瓶颈效应分析,采用符号检验(Sign Test)和Wilcoxon符号秩序测试(Wilcoxon Sign-rank Test)来确定杂合度过剩是否显著(Maruyama et al,1985); 采用STRUCTURE 2.3软件对群体结构进行分析,分析最佳K值,以确定群体遗传结构的理论群体数(Pritchard et al,2000)。
2 结果与分析 2.1 微卫星位点多态性及群体的遗传多样性本研究选用了29个河蟹微卫星位点,在6个群体中均实现成功扩增(表 1)。共检测到943个等位基因,各位点等位基因数(Na)介于11—52之间(图 2),平均为32.52,有效等位基因数(Ne)在1.844—31.269之间,平均为15.637; 各位点观测杂合度(Ho)在0.444—0.941之间,平均为0.723,期望杂合度He介于0.458—0.969,平均为0.876; 平均多态信息含量PIC=0.864,除位点Esin87外,其余位点均为高多态性位点(PIC>0.5); 平均香农信息指数I=2.732; 位点Esin87、HLJEsa42、HLJEsc34、Q67、Q703处于Hardy-Weinberg平衡,其余位点均显著或极显著偏离Hardy-Weinberg平衡(P<0.05)。
| 位点 | 等位基因 长度(bp) | 等位基 因数Na | 有效等位基 因数Ne | 观测杂 合度Ho | 期望杂 合度He | 香农信息指数I | 多态信息 含量PIC | 近交系数 FIS |
| Esin87 | 123—149 | 17 | 1.844 | 0.444 | 0.458 | 1.142 | 0.444 | 0.026 |
| HLJEsc56 | 193—236 | 12 | 4.809 | 0.669** | 0.793 | 1.847 | 0.766 | 0.146 |
| Esin36 | 212—336 | 52 | 22.936 | 0.821** | 0.958 | 3.617 | 0.955 | 0.140 |
| Esin67 | 139—165 | 18 | 3.786 | 0.742** | 0.737 | 1.761 | 0.711 | -0.016 |
| Esin42 | 223—253 | 24 | 11.805 | 0.801** | 0.917 | 2.657 | 0.909 | 0.119 |
| HLJEsa42 | 315—349 | 21 | 10.476 | 0.876 | 0.906 | 2.557 | 0.897 | 0.020 |
| HLJEsa67 | 213—309 | 49 | 25.211 | 0.632** | 0.962 | 3.421 | 0.959 | 0.337 |
| HLJEsc86 | 328—444 | 45 | 24.215 | 0.747** | 0.960 | 3.384 | 0.957 | 0.212 |
| HLJEsb88 | 213—270 | 47 | 31.269 | 0.693** | 0.969 | 3.572 | 0.967 | 0.277 |
| HLJEsc11 | 257—393 | 41 | 22.483 | 0.679** | 0.957 | 3.324 | 0.954 | 0.283 |
| HLJEsc65 | 321—341 | 20 | 11.073 | 0.899** | 0.911 | 2.558 | 0.903 | 0.003 |
| HLJEsb25 | 211—280 | 41 | 23.316 | 0.756** | 0.959 | 3.333 | 0.955 | 0.204 |
| HLJEsd52 | 53—171 | 37 | 21.886 | 0.619** | 0.956 | 3.264 | 0.952 | 0.342 |
| Esin55 | 82—160 | 47 | 19.743 | 0.857 | 0.951 | 3.280 | 0.947 | 0.092 |
| Esin18 | 154—196 | 34 | 22.738 | 0.756** | 0.957 | 3.236 | 0.954 | 0.200 |
| HLJEsd02 | 125—176 | 23 | 9.131 | 0.576** | 0.892 | 2.439 | 0.881 | 0.349 |
| HLJEsc20 | 193—257 | 27 | 12.101 | 0.831** | 0.919 | 2.698 | 0.911 | 0.086 |
| HLJEsc34 | 134—189 | 51 | 28.032 | 0.941 | 0.966 | 3.501 | 0.963 | 0.017 |
| HLJEsa69 | 231—244 | 21 | 6.390 | 0.774** | 0.845 | 2.172 | 0.827 | 0.074 |
| HLJEsa76 | 182—247 | 52 | 27.656 | 0.642** | 0.965 | 3.530 | 0.963 | 0.328 |
| JPX-3 | 297—339 | 42 | 16.589 | 0.615** | 0.941 | 3.083 | 0.937 | 0.339 |
| JPX-10 | 137—205 | 38 | 18.243 | 0.696** | 0.947 | 3.068 | 0.942 | 0.260 |
| JPX-13 | 166—214 | 39 | 21.273 | 0.929** | 0.954 | 3.232 | 0.951 | 0.015 |
| Q67 | 117—189 | 41 | 21.655 | 0.932 | 0.955 | 3.261 | 0.952 | 0.016 |
| Q703 | 139—154 | 15 | 2.64 1 | 0.599 | 0.622 | 1.325 | 0.580 | 0.029 |
| Q726 | 74—104 | 19 | 3.948 | 0.648** | 0.748 | 1.775 | 0.722 | 0.124 |
| Q732 | 104—119 | 11 | 3.604 | 0.589** | 0.724 | 1.564 | 0.682 | 0.180 |
| Q840 | 113—212 | 46 | 22.045 | 0.796** | 0.956 | 3.326 | 0.953 | 0.158 |
| Q934 | 139—175 | 13 | 2.564 | 0.405** | 0.611 | 1.296 | 0.560 | 0.327 |
| 平均 | 32.517 | 15.637 | 0.723 | 0.876 | 2.732 | 0.864 | 0.166 | |
| *表示显著偏离Hardy-Weinberg平衡(P<0.05),**表示极显著偏离Hardy-Weinberg平衡(P<0.01) | ||||||||
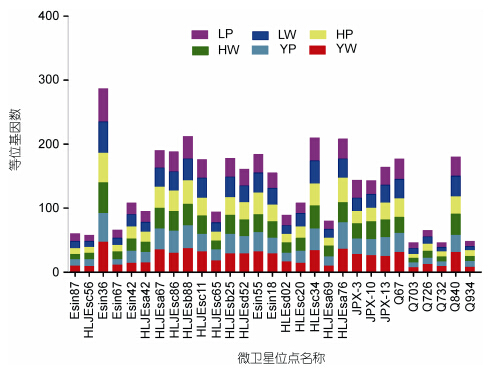 |
| 图 2 6个群体河蟹各微卫星位点的等位基因数量 Fig. 2 Number of Allele for the different SSR loci in the six populations of E. sinensis |
6个群体的遗传多样性数据见表 2。整体上,6个群体的平均等位基因数(Na)、平均有效等位基因数(Ne)、平均观测杂合度(Ho)、平均期望杂合度(He)和平均香农指数(I)接近,各群体的遗传多样性水平均较高。就近交系数(FIS)值而言,6个群体间存在一定的差异,其中辽河养殖群体最高,黄河养殖群体最低,大小顺序依次为: 辽河养殖>长江野生>辽河野生>长江养殖=黄河野生>黄河养殖。就各位点群体内FIS而言,除Esin67位点为负值(-0.016)外,其余均为正值。
| 统计指标 | 群体 | |||||
| YW | YP | HW | HP | LW | LP | |
| 平均等位基因数Na | 22.897 | 22.724 | 22.586 | 22.034 | 22.379 | 22.552 |
| 平均有效等位基因数Ne | 14.006 | 14.078 | 14.099 | 13.55 | 13.08 | 13.757 |
| 平均观测杂合度Ho | 0.713 | 0.727 | 0.733 | 0.744 | 0.715 | 0.702 |
| 平均期望杂合度He | 0.881 | 0.87 | 0.872 | 0.874 | 0.87 | 0.877 |
| 平均香农信息指数I | 2.621 | 2.605 | 2.602 | 2.58 | 2.566 | 2.594 |
| 平均近交系数FIS | 0.178 | 0.151 | 0.151 | 0.139 | 0.163 | 0.187 |
6个群体的遗传分化情况见表 3。除长江野生与黄河野生群体(FST=0.0040)、长江野生与辽河养殖群体(FST=0.0050)、黄河养殖与辽河养殖群体(FST=0.0034)的遗传分化极显著(P<0.01)或显著(P<0.05)外,其余群体之间的遗传分化不显著; 所有种群间的遗传分化均属轻微程度的分化(FST<0.05)。Nei’s遗传距离数据(Ds)与F-统计值(FST)的结果基本一致,长江野生与辽河养殖群体遗传距离最大(Ds= 0.0989),其次为长江野生与黄河野生(Ds=0.0968),长江养殖与黄河野生群体的遗传距离最小(Ds=0.0664)(表 3)。
| 种群 | YW | YP | HW | HP | LW | LP |
| YW | **** | 0.0028 | 0.0040** | 0.0011 | 0.0026 | 0.0050** |
| YP | 0.0855 | **** | 0.0008 | 0.0008 | 0.0013 | 0.0028 |
| HW | 0.0968 | 0.0664 | **** | 0.0013 | 0.0017 | 0.0016 |
| HP | 0.0848 | 0.0745 | 0.0752 | **** | 0.0021 | 0.0034 |
| LW | 0.0907 | 0.0791 | 0.0705 | 0.0827 | **** | 0.0013 |
| LP | 0.0989 | 0.0841 | 0.0750 | 0.0884 | 0.0699 | **** |
| “”表示分化达到显著水平(P<0.05); “**”表示分化达到极显著水平(P<0.01) | ||||||
基于Nei’s遗传距离的UPGMA进化树结果显示,长江野生群体单独聚为一支,其余5群体共同聚为大一支,其中长江养殖群体、黄河野生群体与黄河养殖群体聚为一小支,辽河野生群体和养殖群体聚为另一小支,这两小支共同形成一大支,它们与长江野生群体并列。分子方差分析(AMOVA)结果显示,14.02%的遗传变异来自种群内个体间,85.88%的变异来自个体内,来源于群体之间的差异仅占到0.10%(表 4)。
| 变异来源 | 自由度(df) | 平方和(MS) | 方差组分(Va) | 变异组成(%) | ||||
| 种群间 | 5 | 50.89 | 0.00806 | 0.10 | ||||
| 种群内 | 354 | 3255.750 | 1.13185** | 14.02 | ||||
| 个体内 | 360 | 2496.000 | 6.93333** | 85.88 | ||||
| 总变异 | 719 | 5802.569 | 8.07324 | |||||
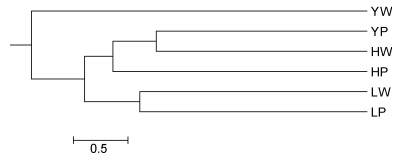 |
| 图 3 基于Nei’s遗传距离的6个河蟹群体UPGMA聚类图Fig. 3 UPGMA clustering tree of E. sinensis based on Nei’ s genetic distance among populations |
29个微卫星位点的期望杂合度(He)与在3个模型下的平均期望杂合度(Heq)见表 5。在IAM模型下,除Esin87、Esin67之外,其它位点的He均大于Heq,有13个位点无显著差异,其余位点均有显著或极显著差异; 在TPM模型下,8个位点的He小于Heq,其余位点He大于Heq,4个位点差异显著(P<0.05),其余位点无显著差异; 在SMM模型下,位点17个位点的He小于Heq,其余位点He大于Heq,共5个位点差异显著(P<0.05),其中位点Esin87、Esin67在TPM和SMM模型下均差异极显著(P<0.01)。
| 位点 | IAM | TPM | SMM | |||||||
| He | Heq | DH/SD | P | Heq | DH/SD | P | Heq | DH/SD | P | |
| Esin87 | 0.458 | 0.759 | -3.339 | 0.010 | 0.848 | -10.055 | 0.000** | 0.899 | -23.640 | 0.000** |
| HLJEsc56 | 0.793 | 0.677 | 0.919 | 0.147 | 0.783 | 0.156 | 0.491 | 0.854 | -1.997 | 0.053 |
| Esin36 | 0.958 | 0.821 | 1.520 | 0.008** | 0.871 | 1.442 | 0.025 | 0.887 | 0.779 | 0.193 |
| Esin67 | 0.737 | 0.766 | -0.310 | 0.275 | 0.857 | -3.311 | 0.008** | 0.905 | -9.960 | 0.000** |
| Esin42 | 0.917 | 0.832 | 1.335 | 0.002** | 0.898 | 0.800 | 0.219 | 0.928 | -0.469 | 0.159 |
| HLJEsa42 | 0.906 | 0.805 | 1.412 | 0.008** | 0.881 | 0.837 | 0.204 | 0.919 | -0.637 | 0.150 |
| HLJEsa67 | 0.962 | 0.943 | 1.089 | 0.079 | 0.957 | 0.156 | 0.569 | 0.963 | -0.530 | 0.191 |
| HLJEsc86 | 0.960 | 0.932 | 0.720 | 0.246 | 0.949 | -0.422 | 0.278 | 0.956 | -1.080 | 0.079 |
| HLJEsb88 | 0.969 | 0.948 | 1.664 | 0.000** | 0.961 | 0.767 | 0.036 | 0.966 | 0.732 | 0.201 |
| HLJEsc11 | 0.957 | 0.938 | 1.300 | 0.022 | 0.952 | 0.574 | 0.276 | 0.959 | 0.024 | 0.437 |
| HLJEsc65 | 0.911 | 0.855 | 1.340 | 0.020 | 0.895 | 0.850 | 0.177 | 0.918 | -0.008 | 0.430 |
| HLJEsb25 | 0.959 | 0.929 | 1.395 | 0.031 | 0.946 | 0.688 | 0.255 | 0.953 | 0.040 | 0.459 |
| HLJEsc52 | 0.956 | 0.925 | 0.458 | 0.381 | 0.944 | -0.497 | 0.173 | 0.953 | -1.263 | 0.020 |
| Esin55 | 0.951 | 0.935 | 1.005 | 0.125 | 0.951 | 0.060 | 0.491 | 0.957 | -0.314 | 0.160 |
| Esin18 | 0.957 | 0.929 | 1.304 | 0.020 | 0.946 | 0.640 | 0.221 | 0.954 | 0.049 | 0.449 |
| HLJEsd02 | 0.892 | 0.831 | 1.013 | 0.092 | 0.878 | 0.543 | 0.341 | 0.907 | -0.563 | 0.178 |
| hljesc20 | 0.919 | 0.809 | 1.508 | 0.004** | 0.859 | 1.363 | 0.019 | 0.892 | 0.882 | 0.190 |
| HLJEsc34 | 0.966 | 0.946 | 1.400 | 0.017 | 0.958 | 0.542 | 0.179 | 0.964 | 0.260 | 0.528 |
| HLJEsa69 | 0.845 | 0.719 | 1.202 | 0.036 | 0.789 | 0.964 | 0.141 | 0.842 | 0.157 | 0.527 |
| HLJEsa76 | 0.965 | 0.947 | 1.363 | 0.015 | 0.960 | 0.593 | 0.231 | 0.965 | 0.156 | 0.609 |
| JPX-3 | 0.941 | 0.928 | 1.439 | 0.013 | 0.943 | 0.473 | 0.169 | 0.953 | 0.206 | 0.533 |
| JPX-10 | 0.947 | 0.909 | 1.091 | 0.051 | 0.935 | 0.352 | 0.400 | 0.946 | -0.296 | 0.242 |
| JPX-13 | 0.954 | 0.907 | 1.553 | 0.005** | 0.932 | 1.137 | 0.082 | 0.943 | 0.283 | 0.435 |
| Q67 | 0.955 | 0.933 | 1.476 | 0.008** | 0.949 | 0.756 | 0.129 | 0.957 | 0.194 | 0.539 |
| Q703 | 0.622 | 0.619 | 0.016 | 0.418 | 0.696 | -0.835 | 0.171 | 0.766 | -2.616 | 0.023 |
| Q726 | 0.748 | 0.772 | 0.415 | 0.428 | 0.832 | -0.548 | 0.213 | 0.870 | -2.610 | 0.019 |
| Q732 | 0.724 | 0.696 | 0.553 | 0.340 | 0.766 | -0.119 | 0.373 | 0.823 | -1.728 | 0.068 |
| Q840 | 0.956 | 0.933 | 1.177 | 0.050 | 0.950 | 0.314 | 0.447 | 0.957 | -0.182 | 0.255 |
| Q934 | 0.611 | 0.659 | 0.436 | 0.415 | 0.740 | -0.354 | 0.286 | 0.798 | -1.886 | 0.055 |
| DH/SD表示He与Heq的差与标准偏差之比 | ||||||||||
河蟹6群体突变-漂移平衡分析如表 6所示。在符号检验中,IAM模型下6个群体均表现为杂合度过高,显著或极显著偏离突变-漂移平衡; TPM模型下几个群体亦表现为杂合过度,但只有黄河野生和黄河养殖群体显著偏离平衡(P<0.05); 在SSM模型下,所有群体均表现出一定程度的杂合不足,除黄河养殖群体外,其余5个群体显著或极显著偏离平衡。Wilcoxon符号秩序检验中,IAM模型下所有群体均极显著(P<0.01)偏离突变-漂移平衡; TPM模型下,长江野生群体显著偏离平衡(P<0.05),黄河养殖群体极显著偏离平衡(P<0.01),其余各群体则处在突变-漂移平衡中; SMM模型下,长江野生和辽河养殖群体显著偏离平衡(P<0.05),辽河野生群体极显著偏离平衡(P<0.01),其余群体未偏离突变-漂变平衡。
| 群体 | 符号检验 | Wilcoxon符号秩序检验 | |||||||||
| IAM | TPM | SMM | IAM | TPM | SMM | ||||||
| HE/HD | P | HE/HD | P | HE/HD | P | P | P | P | |||
| YW | 27/2 | 0.00013** | 21/8 | 0.16042 | 13/16 | 0.03732 | 0.00002** | 0.04775 | 0.03793 | ||
| YP | 25/4 | 0.00377** | 22/7 | 0.09214 | 11/18 | 0.00680** | 0.00015** | 0.08145 | 0.05050 | ||
| HW | 25/4 | 0.00381** | 23/6 | 0.03763 | 12/17 | 0.01460 | 0.00009** | 0.18578 | 0.19356 | ||
| HP | 27/2 | 0.00014** | 24/5 | 0.01166 | 14/15 | 0.07855 | 0.00006** | 0.00606** | 0.14377 | ||
| LW | 24/5 | 0.01225 | 20/9 | 0.27527 | 9/20 | 0.00039** | 0.00011** | 0.49323 | 0.00117** | ||
| LP | 25/4 | 0.00378** | 18/11 | 0.54331 | 13/16 | 0.03988 | 0.00004** | 0.21827 | 0.03575 | ||
| HE/HD表示杂合子过剩与杂合子缺失位点个数比 | |||||||||||
本研究预热次数设为10000,预设聚类数值(K值)为2—10个,每个K值下重复运算5次,结果发现每个K值下差异不大,说明运算长度符合要求。根据K值对应的相关参数的趋势,未发现明显的折点,未能成功划分6群体河蟹的理论群体数。图 4为在K=2、3、4时的遗传结构图,图中各种颜色分布相互混乱,不能聚成相对独立的群体,这暗示三水系6群体河蟹的遗传组成混杂严重,遗传分化不明显。
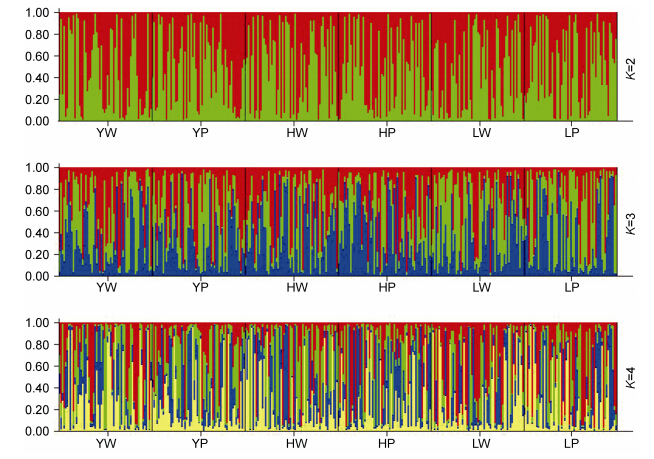 |
| 图 4 河蟹群体在不同假设K值下的遗传结构图Fig. 4 STRUCTURE genetic cluster analysis for the six populations of E. sinensis |
本研究筛选了中华绒螯蟹29个的微卫星位点,在360只个体DNA样本中进行扩增,并利用高分辨率的荧光标记毛细管电泳技术进行等位基因分型,从研究结果看,本研究中29个SSR位点的平均等位基因数(Na=32.5170)和平均多态信息含量(PIC=0.864)多高于先前河蟹种群SSR遗传分析的研究报道(Chang et al,2008; Sui et al,2009; 熊良伟等,2012; 唐刘秀等,2013),除位点Esin87外,其余位点均为高多态性位点。本研究每个群体选择样本数60个,多于其上述研究样本数量(28—40个),在一定范围之内,样本量的大小与所观测到的不同基因座等位基因检出数量之间存在正相关关系(高雅等,2008)。整体上,对相同群体和相同SSR位点而言,本研究中等位基因数量、期望杂合度和观测杂合度在大多数位点高于先前报道(Hänfling et al,2003; Chang et al,2006; 唐刘秀等,2013),这可能与样品数量和检测技术不同有关。通常大样本量和毛细管电泳分型技术能够提供更可靠的遗传多样性信息(刘皓等,2015)。由此可见,本研究选用的SSR位点、样本量和毛细管电泳分型技术,可以较全面地反映各水系河蟹的遗传多样性信息,能够有效评估其群体遗传特性。
本研究中6个群体平均期望杂合度均较高(He=0.870—0.881),显示本研究中野生和养殖河蟹群体均具有较高的遗传多样性水平,这与先前对我国几个野生群体(Sui et al,2009)和养殖群体(朱泽远等,2007; 熊良伟等,2012)的研究结果相似。就野生群体而言,香农信息指数反映的遗传多样性依次为: 长江野生>黄河野生>辽河野生。遗传多样性水平由南向北依次降低,这种地理差异往往和生长环境有关,长江水系流域面积广,与干流相通的河道与湖泊多,适宜河蟹生长和育肥,因而种群数量较大,相比之下,黄河及辽河水系由于所处纬度较高,水温较低,流域面积相对较小,因此种群规模相对较小。3个养殖群体的遗传多样性依次为: 长江养殖>辽河养殖>黄河养殖,本研究中长江和辽河水系养殖群体的遗传多样性情况与熊良伟等(2012)研究结果基本一致,黄河养殖群体遗传多样性低于长江和辽河养殖群体,这可能是由于黄河流域河蟹养殖规模相对较小,其人工繁殖的基础群体亲本数量较少,经过多代人工繁殖后导致其遗传多样性进一步下降(熊良伟等,2012)。
从等位基因数和香农信息指数看,本研究中长江和黄河的养殖群体略低于相应的野生群体,这与熊良伟等(2012)的研究结果相似,究其原因可能是两个养殖群体的有效亲本数量较少造成的始祖效应(founder effect),后续传代育苗过程中主要通过表型选择亲本,容易增加近交几率,类似的情况已经在大西洋鲑(Salmon salar)中被证实(Skaala et al,2004)。辽河养殖群体的等位基因数和香农信息指数高于辽河野生群体,可能有两方面的原因。其一,由于辽河水系面积相对较小,其野生河蟹群体数量远小于长江水系和黄河水系,加之由于过渡捕捞、环境污染和洄游通道被水闸阻挡等原因,其野生群体经历过有效种群下降,导致其遗传多样性较低(王武等,2013); 其二,由于长江水系河蟹规格较大,长江水系河蟹曾经被引入辽河流域进行池塘养殖,因此辽河水系养殖群体亦或曾受到长江群体的影响,导致种质混杂,其遗传多样性偏高(王武等,2007)。
3.2 群体的遗传结构分析本研究中6个群体的河蟹种群的遗传距离(DA)与地理距离呈现一定的相关性,但并不完全吻合。如果没有发现不同地理种群间的遗传分化,基因流常被作为“默认”的解释(Sui et al,2009)。有研究者基于微卫星标记的瓶颈效应和基因流动分析研究欧亚多刺水蚤(Bythotrephes longimanus)入侵的报道提出,迅速蔓延的多产物种有可能获得来自多个种群的基因流,而导致其群体遗传分化较低(Colautti et al,2005)。本研究天然地理种群结合地理相关养殖群体,能对地理种群间天然的基因流和因养殖活动中跨区域引种和养殖群体逃逸产生的基因流动作出侧面的反映。从基于遗传距离的聚类分析结果来看,相对其它水系而言,辽河水系的野生与养殖群体组成了一个较为封闭的地理种群单位,而长江、黄河养殖及野生群体可能因跨地域相互引种而导致有一定程度的混杂。李晓晖等(2010)对河蟹长江水系野生群体和几个养殖群体进行分析后,发现射阳地区养殖群体遗传多样性指数高于其它几个群体,同样认为是不同水系河蟹种质混杂的结果。
群体的遗传分化值数(FST)能揭示种群间的遗传分化程度,FST值在0—0.05之间,群体的遗传分化较弱; FST值在0.05—0.15之间时,遗传分化水平中等,FST值大于0.15时,遗传分化较大(Balloux et al,2002)。本研究的几个群体间的FST值在0.0008— 0.0050之间,远小于0.05,遗传分化水平微弱,这与我国6个水系的11个野生群体的微卫星遗传多样性研究结果基本吻合(FST=0.0001—0.0400)(Sui et al,2009)。而在中国沿海5个三疣梭子蟹(Portunus trituberculatus)地理种群中FST值为0.0548— 0.1083(李晓萍,2010),日本沼虾(Macrobrachium nipponense)的几个中国地理种群中,该值达0.0542— 0.1559(Yang et al,2010),克氏原螯虾(Procambarus clarkii)的长江下游几个地理种群中,该值亦高达0.0405—0.1559(王长忠等,2009),日本对虾(Marsupenaeus japonicas)野生和人工养殖种群的FST值略低为0.023(Luan et al,2006),这几种重要经济甲壳动物地理种群的遗传分化均高于河蟹。Brown等(1989)认为异交物种90%以上的遗传变异来自群体内部,本研究中分子方差分析结果显示,群体之间的变异仅占变异总数的0.10%,部分变异来源于群体内个体间(14.02%),绝大部分变异来自个体内部(85.88%),表明河蟹群体变异程度较低。与其它甲壳动物相比,河蟹具有复杂的生活史和开放的生命周期,长距离江海洄游习性使其面临多种差异较大的生境。由此,与其它甲壳动物相比,推测河蟹更容易因某些等位基因的丢失造成生命周期中某个环节的环境适应能力减弱或丧失,进而导致变异个体死亡或不能成功繁殖,而表现出较强的遗传保守性。
3.3 群体动态分析6个种群的瓶颈分析结果中,在3种模型下,均有部分位点呈现出He与Heq的显著或极显著差异,在符号检验和Wilcoxon符号秩序检验中,IAM模型下,显示几个群体近期经历了不同程度的瓶颈效应,SMM模型下,除了黄河养殖群体外,其余5个群体均存在瓶颈效应。朱泽远等(2007)在研究中发现几个河蟹养殖群体均存在瓶颈效应,王中清等(2013)在2004、2006、2007和2011年长江野生群体中亦发现瓶颈效应,结合上述研究,表明中国大陆河蟹种群正面临着持续的种质衰退。遗传结构分析结果表明,本研究的河蟹野生群体和养殖群体基因混杂现象严重,这一结果与徐灿(2007)的观点相符,其研究认为自1982年起,中国大陆多个水系的河蟹便开始通过养殖活动中的无序引种和增殖放流交互混杂。
本研究中29个微卫星位点中,有24个位点偏离Hardy-Weinberg平衡,观测杂合度小于期望杂合度。Hardy-Weinberg平衡的偏离可能由多种原因导致,如非随机交配、选择、突变、无效等位基因等,这样就会造成群体内存在的等位基因的丢失,纯合子增加,种群的遗传多样性降低,从而增加近交衰退的几率(杨锐等,2000)。当群体内观测杂合度小于期望杂合度,同时FIS为正值,则表示群体内近交程度较为严重(Frankham,2010),本研究的28个位点的FIS均为正值,说明三水系的河蟹野生和养殖群体内近交程度严重,存在种质衰退的风险。先前研究表明,长江野生扣蟹的养殖性能优于多年家养的池塘群体(He et al,2014),这也暗示了长江水系养殖群体种质退化严重,遗传多样性较低。
综上,6个种群遗传多样性较高,但存在明显的种质混杂和群内近交,加之瓶颈效应的造成的影响,这可能使其优异种质退化,不利于河蟹种质资源的保护和开发利用,因此需要制订科学的野生种质保护方案和良种选育策略。对河蟹野生群体进行保护,在进行水利工程时应充分考虑对河蟹洄游的影响,天然水域增殖放流应保证苗种来源于相应的水系,从而避免种质混杂。对河蟹进行良种选育,应尽量扩大有效有效群体数量,降低近交衰退几率,进一步采取家系选育技术,筛选与表型相关的分子标记进行多性状复合育种提高选育效率。本研究为河蟹种质资源评价、良种选育和分子标记开发提供了重要的基础资料。
致谢 上海海洋大学水产与生命学院邱高峰教授提供部分微卫星引物,傅建军博士后在数据分析和论文写作过程给予热情指导,谨致谢忱。| 马雪霞, 王 凯, 郭旺珍等, 2007.棉花SSR多重PCR技术的初步研究和利用.分子植物育种, 5(5): 648-654 |
| 王 武, 王成辉, 马旭洲, 2013.河蟹生态养殖.北京: 中国农业出版社, 59-84 |
| 王 武, 成永旭, 李应森等, 2007.河蟹的生物学.水产科技情报, 35(1): 25-84 |
| 王 婷, 黄智慧, 马爱军等, 2014.基于转录组数据的大菱鲆(Scophthalmus maximus)SNP标记开发及多态性分析.海洋与湖沼, 45(6): 1300-1307 |
| 王中清, 黄 姝, 茅海成等, 2013.中华绒螯蟹奇、偶年天然群体的遗传差异分析.上海海洋大学学报, 22(5): 657-664 |
| 王长忠, 李 忠, 梁宏伟等, 2009.长江下游地区4个克氏原螯虾群体的遗传多样性分析.生物多样性, 17(5): 518-523 |
| 朱泽远, 王亚菊, 施用晖等, 2007.荧光标记微卫星分析人工饲养中华绒螯蟹的遗传多样性.中国海洋大学学报, 37(4): 591-596 |
| 朱清顺, 夏爱军, 潘建林等, 2008."苏蟹一号" F3代选育群体生物学特性分析.水产养殖, 29(6): 8-12 |
| 刘 凯, 汤 滔, 段金荣等, 2013.长江口九段沙水域中华绒螯蟹汛期特征及影响因子.中国水产科学, 20(3): 614-623 |
| 刘 皓, 刘 青, 吴旭干等, 2015.中华绒螯蟹两种微卫星DNA分型方法的应用比较.生物学杂志, 32(1): 90-94 |
| 许加武, 任明荣, 李思发等, 1997.长江、辽河、瓯江中华绒螯蟹种群的形态判别.水产学报, 21(3): 269-274 |
| 李 勇, 李思发, 王成辉等, 2001.三水系中华绒螯蟹幼蟹形态判别程序的建立和使用.水产学报, 25(2): 120-126 |
| 李思发, 邹曙明, 1999.中国大陆沿海六水系绒螯蟹(中华绒螯蟹和日本绒螯蟹)群体亲缘关系: RAPD指纹标记.水产学报, 23(4): 325-330 |
| 李晓晖, 许志强, 潘建林等, 2010.中华绒螯蟹人工选育群体的遗传多样性.中国水产科学, 17(2): 236-242 |
| 李晓萍, 2010.三疣梭子蟹微卫星富集文库的构建及五个野生地理群的多样性分析.青岛: 中国海洋大学硕士学位论文 |
| 李海燕, 侯 林, 魏凤艳, 2003.中华绒螯蟹辽河种群野生、养殖及"性早熟"个体RAPD分析.水产科学, 22(3): 1-3 |
| 李晨虹, 李思发, 邢益于等, 2002.池养长江蟹、辽河蟹生长性能及其遗传-环境交互作用分析.水生生物学报, 26(4): 335-341 |
| 杨 锐, 喻子牛, 陈再忠等, 2000.山东沿海褶牡蛎与太平洋牡蛎等位基因酶的遗传变异.水产学报, 24(2): 130-133 |
| 张世勇, 傅洪拓, 乔 慧等, 2013.中华绒螯蟹遗传育种研究进展.中国农学通报, 29(20): 39-45 |
| 周 刚, 朱清顺, 胡本龙, 2003.不同水系中华绒螯蟹生长比较的初步研究.水产养殖, 24(6): 34-37 |
| 赵 娜, 2014.盘山县稻蟹生态种养模式的研究.延吉: 延边大学硕士学位论文 |
| 施德龙, 2006.关于对2006年长江口蟹苗汛的预测及对长江口蟹苗的捕捞、运输、放养方法介绍.渔业致富指南, (8): 10-11 |
| 徐 灿, 2007.不同水系河蟹种质混杂的初步研究.上海: 上海水产大学硕士学位论文, 2-4 |
| 高 雅, 李生斌, 2008.STR遗传多态性研究中样本数量对等位基因检出数量的影响.遗传, 30(3): 313-320 |
| 唐刘秀, 许志强, 葛家春等, 2013.中华绒螯蟹3个育种基础群体遗传特征的微卫星分析.南京师大学报(自然科学版), 36(2): 84-90 |
| 龚疏影, 王 茜, 汤华鑫等, 2009.长江、辽河野生与天津养殖中华绒螯蟹同工酶的比较.天津师范大学学报(自然科学版), 29(1): 51-54 |
| 崔江华, 2010.黄河口中华绒螯蟹养殖技术研究与应用.北京: 中国农业科学院硕士学位论文 |
| 熊良伟, 2014.中华绒螯蟹遗传连锁图谱构建及经济相关性状QTL定位.上海: 上海海洋大学硕士学位论文, 64-67 |
| 熊良伟, 李 真, 马克异等, 2012.利用微卫星DNA分子标记分析中华绒螯蟹养殖群体遗传分化.农业生物技术学报, 20(12): 1441-1448 |
| Baker J S F, 1994.A global protocol for determining genetic distances among domestic livestock breeds.In: Proceedings of the 5th World Congress on Genetics Applied to Livestock Production.Guelph and Ontario, Canada, 501-508 |
| Balloux F, Lugon-Moulin N, 2002.The estimation of population differentiation with microsatellite markers.Mol Ecol, 11(2): 155-165 |
| Bentzen P, Olsen J B, McLean J E et al, 2001.Kinship analysis of Pacific salmon: Insights into mating, homing, and timing of reproduction.Journal of Heredity, 92(2): 127-136 |
| Botstein D, White R L, Skolnick M et al, 1980.Construction of a genetic linkage map in man using restriction fragment length polymorphisms.Am J Hum Genet, 32(3): 314-331 |
| Brown A H D, Clegg M T, Kahler A L et al, 1989.Plant population genetics, breeding, and genetic resources.Sunderland Massachusetts Sinauer Associates, 43-63 |
| Chang Y M, Liang L Q, Li S W et al, 2006.A set of new microsatellite loci isolated from Chinese mitten crab, Eriocheir sinensis.Mol Ecol Notes, 6(4): 1237-1239 |
| Chang Y M, Liang L Q, Ma H T et al, 2008.Microsatellite analysis of genetic diversity and population structure of Chinese mitten crab (Eriocheir sinensis).J Genet Genomics, 35(3): 171-176 |
| Cheng Q X, Yuan C H, Wang J et al, 2010.Development of 20 microsatellite loci in the Japanese mitten crab Eriocheir japonica and cross-amplification in the Chinese mitten crab Eriocheir sinensis.Conserv Genet Resour, 2(1): 47-50 |
| Cheng Y X, Wu X G, Yang X Z et al, 2008.Current trends in hatchery techniques and stock enhancement for Chinese mitten crab, Eriocheir japonica sinensis.Rev Fish Sci, 16(1-3): 377-384 |
| Colautti R I, Manca M, Viljanen M et al, 2005.Invasion genetics of the Eurasian spiny waterflea: evidence for bottlenecks and gene flow using microsatellites.Mol Ecol, 14(7): 1869-1879 |
| Exoffier L, Lischer H E L, 2010.Arlequin suite ver 3.5: a new series of programs to perform population genetics analyses under Linux and Windows.Mol Ecol Resour, 10(3): 564-567 |
| Frankham R, Ballou J D, Briscoe D A, 2010.Introduction to Conservation Genetics.Cambridge: Cambridge University Press, 260-308 |
| Hänfling B, Weetman D, 2003.Characterization of microsatellite loci for the Chinese mitten crab, Eriocheir sinensis.Mol Ecol Notes, 3(1): 15-17 |
| He J, Wu X G, Li J Y et al, 2014.Comparison of the culture performance and profitability of wild-caught and captive pond-reared Chinese mitten crab (Eriocheir sinensis) juveniles reared in grow-out ponds: Implications for seed selection and genetic selection programs.Aquaculture, 434: 48-56 |
| Herborg L, Weetman D, Van Oosterhout C et al, 2007.Genetic population structure and contemporary dispersal patterns of a recent European invader, the Chinese mitten crab, Eriocheir sinensis.Molecular Ecology, 16(2): 231-242 |
| Luan S, Kong J, Wang Q Y, 2006.Genetic variation of wild and cultured populations of the Kuruma prawn Marsupenaeus japonicus (Bate 1888) using microsatellites.Aquac Res, 37(8): 785-792 |
| Maruyama T, Fuerst P A, 1985.Population bottlenecks and nonequilibrium models in population genetics.II.Number of alleles in a small population that was formed by a recent bottleneck.Genetics, 111(3): 675-689 |
| Nei M, 1978.Estimation of average heterozygosity and genetic distance from a small number of individuals.Genetics, 89(3): 583-590 |
| Pritchard J K, Stephens M, Donnelly P, 2000.Inference of population structure using multilocus genotype data.Genetics, 155(2): 945-959 |
| Skaala Ø, Høyheim B, Glover K et al, 2004.Microsatellite analysis in domesticated and wild Atlantic salmon (Salmo salar L.): allelic diversity and identification of individuals.Aquaculture, 240(1-4): 131-143 |
| Sui L Y, Wille M, Cheng Y X et al, 2011.Larviculture tecniques of Chinese mitten crab Eriocheir sinensis.Aquaculture, 315: 16-19 |
| Sui L Y, Zhang F M, Wang X M et al, 2009.Genetic diversity and population structure of the Chinese mitten crab Eriocheir sinensis in its native range.Marine Biology, 156(8): 1573-1583 |
| Tamura K, Peterson D, Peterson N et al, 2011.MEGA5: molecular evolutionary genetics analysis using maximum likelihood, evolutionary distance, and maximum parsimony methods.Mol Biol Evol, 28(10): 2731-2739 |
| Xiong L W, Wang Q, Qiu G F, 2012.Large-Scale isolation of microsatellites from Chinese mitten crab Eriocheir sinensis via a solexa genomic survey.Int J Mol Sci, 13(12): 16333-16345 |
| Yang P, Chen L Q, Wang W et al, 2010.Genetic diversity of oriental river prawn (Macrobrachium nipponense De Haan) revealed by ISSR markers.Journal of Fishery Sciences of China, 17(5): 913-921 |
 2015, Vol. 46
2015, Vol. 46


